Ancient variants of the Epstein–Barr virus (Herpesviridae, Lymphocryptovirus, HHV-4): hypotheses and facts
- Authors: Smirnova K.V.1, Senyuta N.B.2, Lubenskaya A.K.2, Dushenkina T.E.2, Gurtsevich V.E.2
-
Affiliations:
- PhD, Head of the Laboratory of Viral Carcinogenesis of the N.N. Blokhin NMRC of Oncology, Moscow, 115478, Russia
- Research Institute of Carcinogenesis, N.N. Blokhin National Medical Research Center of Oncology
- Issue: Vol 65, No 2 (2020)
- Pages: 77-86
- Section: ORIGINAL RESEARCHES
- URL: https://virusjour.crie.ru/jour/article/view/282
- DOI: https://doi.org/10.36233/0507-4088-2020-65-2-77-86
- ID: 282
Cite item
Full Text
Abstract
Introduction. Molecular studies have shown that viruses appeared in the early stages of the evolution of life. For millions of years, viruses have evolved by changing old and acquiring new sequences in their RNA or DNA. It is assumed that most viruses have common ancestors. Such an ancestor, an ancient strain, probably existed for Epstein-Barr virus (EBV) as well.
Aim. To find out whether ancient strains of EBV persist in modern Russian ethnic groups today.
Material and methods. The object of the study was the EBV LMP1oncogene, which is most suitable for molecular genetic analysis. LMP1 was amplified from the oral cavity washings obtained from representatives of two ancient ethnic groups of Russia - Tatars and Slavs. The LMP1 amplicons were sequenced in both directions; their nucleotide sequences translated into amino acid (LMP1) were evaluated using the classification suggested by Edwards et al. 1999. To establish genetic relationships between LMP1 variants, a phylogenetic tree was constructed by the neighbor-joining method using the MEGA software package.
Results and discussion. Analysis of LMP1 sequences from washings of the Slavs oral cavity demonstrated the presence of LMP1 variants with varying degrees of transforming potential: B98.5/A, China1, Med-, and NC. The analysis of LMP1 sequences from washings of Tatar oral cavity also made it possible to identify oncoprotein variants such as B95.8/A, China1, Med-, as well as a group of variants out of classifications (LMP1-OK). An important finding was the identification of 7 variants of LMP1 from Tatars, designated as LMP1-TatK, that contained two unique deletions of 5 aa in codons 312-316 and 382-386, which were absent in the LMP1 variants from Slavs and from previously examined cancer patients and healthy individuals, as well as in sequences from open computer databases. The uniqueness of the LMP1-TatK variant is confirmed both by phylogenetic analysis of LMP1 sequences of Tatar origin and by the analysis of 11 aa repeats and 5 aa insertions in the C-terminal region of the oncoprotein. The morbidity and mortality rates from neoplasms, including EBV-associated pathologies, did not differ significantly between two studied ethnic groups infected with different EBV strains.
Conclusion. The data obtained allowed us to suggest that: 1) LMP1-TatK could be refered to an evolutionarily ancient EBV strain that persists among Tatars and; 2) LMP1-TatK belongs to the so-called “Volga” EBV virus strain, the common strain among the population of the Volga region. Extended studies of EBV isolates from residents of this region may probably shed the light on the origin of LMP1-TatK.
Full Text
Введение
Молекулярные исследования показали, что вирусы появились на ранних этапах эволюции жизни [1] и, вероятно, могли возникать многократно [2]. В настоящее время обсуждаются три классические гипотезы о происхождении вирусов. Согласно первой из них, вирусы являются потомками бактерий или других одноклеточных организмов, претерпевших дегенеративную эволюцию. Согласно второй гипотезе, вирусы являются потомками древних доклеточных форм жизни, перешедших к паразитическому способу существования. Согласно третьей, вирусы являются дериватами клеточных генетических структур, ставших относительно автономными, но сохранивших зависимость от клеток [3]. Учитывая разнообразие мира вирусов, каждая из этих гипотез базируется на соответствующем наборе доказательств, но ни одна не является общепризнанной. В последнее время вирусологи начали пересматривать и переоценивать все три гипотезы [4].
Эволюция существующих и появление новых групп вирусов происходили за счёт изменения старых и приобретения новых последовательностей в их РНК или ДНК [5][6]. Причём мутировавшие вирусы, как правило, начинали доминировать среди «устаревших» аналогов. Генетические изменения в клетках, воспроизводимые вирусами, приводили к генетическим изменениям и в вирусах, ускоряя их эволюцию [7][8].
В настоящее время известно, что у большинства видов вирусов есть общие предки, и хотя гипотеза «первичности вирусов» ещё не получила полного признания, нет сомнений в том, что тысячи видов современных вирусов развились из менее многочисленных древних [7]. Если это так, то возникает вопрос: можно ли в наше время обнаружить древние варианты вирусов, например, для вируса Эпштейна–Барр (ВЭБ).
Известно, что ВЭБ принадлежит к большому семейству герпесвирусов человека. Это наиболее яркий представитель нескольких известных родственных вирусов, эндогенных для обезьян Старого и Нового Света. Весьма вероятно, что ВЭБ развивался вместе со своим хозяином (Homo sapiens и его предками) в течение миллионов лет. Однако эволюция генетической изменчивости ВЭБ изучена недостаточно. Поскольку ген латентного мембранного белка-1 (LMP1) ВЭБ обладает выраженным полиморфизмом, его последовательности могут быть полезны в качестве инструмента для идентификации генотипа вируса, его штамма [9]. Три типа генетической изменчивости способствуют эволюции гена LMP1. Первый – это точечная мутация. Вариация нуклеотидной последовательности субстрата либо отражает географическую вариацию штамма, либо возникает de novo во время продуктивной репликации ВЭБ [9]. Второй тип – делеции последовательностей и дублирование. Генотипы ВЭБ с делециями LMP1 существуют в популяции человека как независимые штаммы [10][11]. Третий – гомологичная рекомбинация. Внутриштаммовая гомологичная рекомбинация происходит в области повтора LMP1, а также в других повторяющихся последовательностях ВЭБ [12][13]. Потомство продуктивной репликации ВЭБ часто имеет иное количество повторяющихся единиц LMP1, чем родительский штамм.
Цель настоящего исследования – на основании вышеприведённых данных выяснить, существуют ли в наше время древние варианты ВЭБ.
Было принято решение осуществлять поиск среди вариантов гена LMP1 в штаммах вируса, персистирующих у представителей двух древних этносов: славян и татар. Учитывая тот факт, что спектр штаммов вируса (и, соответственно, вариантов LMP1 с разным онкогенным потенциалом), инфицирующих население самого крупного мегаполиса – Москвы и Республики Татарстан, может отличаться, представлялось важным сравнить в этих популяциях показатели заболеваемости и смертности от новообразований, в состав которых входят опухоли, ассоциированные с ВЭБ.
Материал и методы
Материалы. Объектом исследования стали образцы онкогена ВЭБ LMP1 в инфицированных вирусом клетках, содержащихся в смывах полости рта славян и татар. Этнические славяне (славяне не менее чем в трёх поколениях) были представлены группой взрослых жителей Москвы (21 мужчина и 19 женщин; средний возраст – 47,5 года). Этнические татары (татары не менее чем в трёх поколениях) были представлены 60 студентами ФГБОУ ВО «Казанский ГМУ» Минздрава России (15 мужчин и 45 женщин, средний возраст – 21,5 года). Все обследуемые лица, включённые в исследование в результате случайной выборки, подписали информированное согласие на участие.
Сравниваемые группы отличались не только вероисповеданием (православные христиане и мусульмане), но и генетически: поволжские татары входят в урало-каспийский кластер с генофондом гаплогрупп Y-хромосомы N1c-LLY22g и I1-M253, а славяне характеризуются четырьмя наиболее распространёнными гаплогруппами: R1a1, N1c1, I2 и I1.
Каждый смыв представлял собой суспензию инфицированных вирусом отшелушившихся эпителиальных клеток, полученных индивидуально от каждого лица, после полоскания полости рта 20 мл стерильного физиологического раствора в течение 30 с. Образцы смывов, собранные в герметично закрывающиеся пластиковые пробирки, хранили при температуре +4 °С не более 2 сут до изучения.
Экстракция ДНК и амплификация вирусных генов. Из клеток, собранных после центрифугирования смывов полости рта методом фенол-хлороформной экстракции, выделяли ДНК. Наличие вирусной ДНК в выделенных образцах анализировали методом полимеразной цепной реакции (ПЦР) в реальном времени, описанном нами ранее [14]. Для определения вирусной ДНК штаммов ВЭБ 1-го и 2-го типа использовали метод гнёздной ПЦР с ранее разработанными парами праймеров [15]. Амплификацию онкогена LMP1 про водили в два этапа с соответствующими внешними и внутренними праймерами по ранее использованной нами методике [16]. Каждый ПЦР-продукт очищали на мини-колонке Qiagen (Германия), согласно инструкции производителя. Для реакции использовали примерно 100–200 нг ПЦР-продукта, концентрацию ДНК оценивали визуально в агарозном геле. В качестве положительного контроля применяли 1 мкг ДНК, выделенной из используемой в качестве стандарта клеточной линии В95.8, а в качестве отрицательного контроля – дистиллированную воду.
Секвенирование ПЦР-продуктов LMP1. Ампликоны онкогена LMP1 секвенировали в обоих направлениях с помощью набора реактивов ABI PRISM® BigDye™ Terminator 3.1 (США) с последующим анализом продуктов реакции на автоматическом секвенаторе ДНК ABI PRISM 3100-Avan (США). Обработку данных секвенирования выполняли с использованием программ Chromas 230 и Vector NTI (Invitrogene, США).
Классификация LMP1. Нуклеотидные последовательности образцов LMP1, амплифицированных из ДНК смывов полости рта и транслированных в аминокислотные последовательности, классифицировали на основании совокупности мутаций, ведущих к заменам отдельных аминокислот, формированию делеций и дупликаций. За основу была принята известная в литературе классификация, сформированная на базе сиквенсного анализа белковых аналогов гена LMP1, полученных от больных ВЭБ-ассоциированной патологией и здоровых вирусоносителей из различных географических регионов мира [17]. При этом показано, что вариант LMP1-China1 является аналогом высокотрансформирующего варианта LMP1-Сао, а вариант LMP1-B95.8 – прототипным вариантом гена с низкой трансформирующей активностью [17][18].
Филогенетический анализ. Для установления генетического родства между образцами LMP1, полученными от татар, их нуклеотидные последовательности транслировали в аминокислотные последовательности, соответствующие кодонам 212–387 прототипного варианта LMP1-В95.8 (NCBI, валидированная последовательность X01995), с помощью пакета программ Vector NTI (Invitrogene, США). Дистанционные матрицы рассчитывали с использованием двухпараметрической модели Кимура. Филогенетическое древо строили на основе полученных данных методом «присоединения соседа» (neighbour joining) с использованием пакета программ MEGA. Достоверность построенного древа оценивали методом бутстреп-анализа. Для филогенетического сравнения использовали доступные в GenBank основные варианты LMP1 ВЭБ (Alaskan, NC, B95.8/A, Med-, China1, China2).
Статистический анализ. Стандартизированные показатели (SR) для заболеваемости и смертности и их стандартные ошибки (SE) в 2015–2017 гг. для некоторых злокачественных опухолей, среди которых встречаются ВЭБ-ассоциированные, в Москве и Татарстане, были получены из публикации [19] . Средние значения SR и их SE были проанализированы для заболеваемости и смертности в 2015–2017 гг. для обоих полов. 95% доверительные интервалы (ДИ) были рассчитаны как SR ± 1,96 SE. Отсутствие перекрытия ДИ для Москвы и Татарстана свидетельствует о статистически значимой разнице между SR на уровне p = 0,05, а перекрытие ДИ показывает, что разница незначительна.
Результаты
Выбор славян и татар для поиска древних вариантов ВЭБ обусловлен их достаточно высокой генетической чистотой и обитанием на территории страны с давних времён. Следует отметить, что не менее половины (а по данным ряда источников, даже более половины) населения Москвы составляют представители славянской национальности. В то же время в Республике Татарстан, в частности в Казани, этнический состав более однороден, а численность татар ненамного, но всё же превышает число представителей славянских народов.
О штаммах ВЭБ, циркулирующих среди московских славян и казанских татар, можно судить по полиморфным формам специфических фрагментов генома ВЭБ, а также вирусного онкогена LMP1, обнаруженных в вирусных изолятах обследованных (табл. 1). Показано, что штаммы ВЭБ с определёнными генетическими мутациями обладают разной трансформирующей способностью. В частности, основное фенотипическое различие известных двух типов вируса состоит в том, что ВЭБ 1-го типа способен трансформировать В-лимфоциты человека в лимфобластоидную клеточную линию более эффективно нежели ВЭБ 2-го типа.
Анализ персистирующих штаммов ВЭБ среди изученной группы татар показал доминирование ВЭБ 1-го типа (84,3% против 15,7% ВЭБ 2-го типа). При этом результаты наших исследований согласуются с данными других авторов, согласно которым ВЭБ 1-го типа наиболее распространён в Европейских странах и на территории США, в то время как ВЭБ 2-го типа – среди населения Африканского континента. Помимо вышеперечисленного изучаемые штаммы вируса отличаются и на молекулярном уровне в плане существенных изменений последовательностей, кодирующих EBNA3A, 3B и 3С, что также отражается на функциональных особенностях изучаемых штаммов.
Анализ нуклеотидных и дедуктивных аминокислотных последовательностей 40 образцов LMP1, полученных из 40 смывов полости рта славян, показал доминирование среди них низкотрансформирующего варианта LMP1-B95.8 (n=33, 82,5%). Обнаружено также несколько вариантов онкобелка с более выраженным по сравнению с B95.8 трансформирующим потенциалом: NC (n=3, 7,5%) и Med- (n=1, 2,5%), а также высокотрансформирующий вариант China1 (n=3, 7,5%).
Анализ 41 ампликона LMP1, полученного из 60 смывов полости рта татар, позволил идентифицировать по классификации R. Edwards и соавт. [17] лишь 21 образец. В том числе низкотрансформирующий вариант B95.8/А (n=12, 29,3%), а также варианты, обладающие умеренным и высокотрансформирующим потенциалом, соответственно Med- (n=6, 14,6%) и China1 (n=3, 7,3%). Среди оставшихся 20 образцов LMP1, полученных от этнических татар, 13 не поддавались классификации из-за набора в них нетипичных хаотично расположенных мутаций. Эта группа образцов LMP1 была обозначена как ВК (вне классификации). Но группа из 7 образцов представила особый интерес благодаря содержанию в них уникальной пары делеций 5 а.к. в кодонах 312–316 и 382–386 (табл. 2).
Указанный вариант LMP1, обозначенный как LMP1-TatK, не был характерен для прототипного варианта LMP1-B95.8 и высокотрансформирующего варианта LMP1-Cao, не обнаружен он и у этнических славян. Указанную делецию нам также не удалось обнаружить в образцах гена, амплифицированных в предыдущие годы из плазмы крови и смывов полости рта больных злокачественными и доброкачественными опухолями из разных регионов России [20] и доноров крови (табл. 3). Не было их и в компьютерной базе данных.
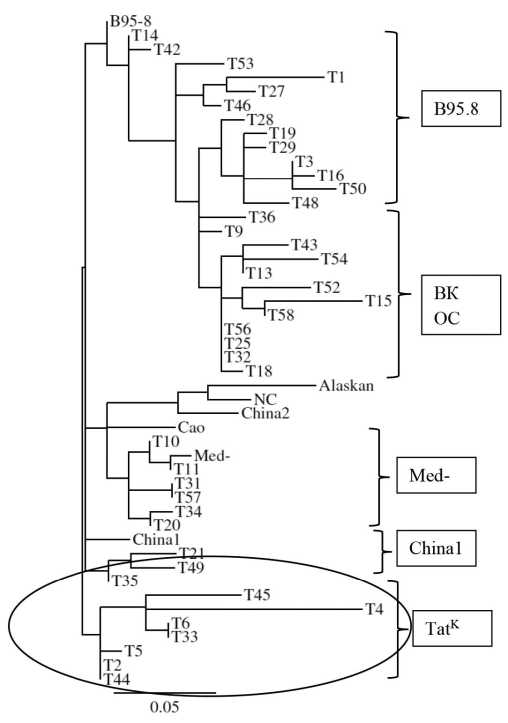
Учитывая генеалогиюизучаемыхлиц(татарыпокрайней мере в третьем поколении), нельзя исключить, что монолитная группа образцов LMP1 с двумя уникальными мутациями, названная LMP1-TatK, относится к эволюционно древнему штамму ВЭБ, обозначенному нами как ВЭБ-TatK. Уникальность варианта LMP1-TatK чётко прослеживается и при филогенетическом анализе образцов LMP1 татарского происхождения (рис. 1).
В созданном древе для сравнения использовали аминокислотные последовательности прототипных вариантов LMP1-В95.8 и LMP1-Сао. Выявлено, что спектр вариантов образцов LMP1 среди этнических татар (по классификации R. Edwards и соавт., 1999) [17] был ограничен вариантами B95.8, Med- и China1. Образцы варианта LMP1-TatK и группы ВК в филогенетическом древе сформировали два отдельно расположенных кластера. Обращает на себя внимание близость образцов кластера LMP1-TatK к образцам кластера LMP1-China1, обладающих высоким трансформирующим и онкогенным потенциалом, и, наоборот, удалённость образцов кластера LMP1-TatK от образцов кластера LMP1 B95.8 с невысокой трансформирующей активностью. Уникальность образцов, относящихся к варианту LMP1-TatK, также видна при анализе аминокислотных повторов и вставок в С-терминальной области онкобелка. Ранее было показано, что эта область LMP1 содержит варьирующее число 11-членных аминокислотных повторов (PQDPDNTDDNG) на участке 253–306 а.к. [20]. Прототипный вариант LMP1-В95.8 имеет 4 подобных повторяющихся элементов и две 5-аминокислотные (PHDPL) вставки: одна расположена между 2-м и 3-м повторами (275–279-й аминокислотные остатки), а вторая в конце повторов (302–306-й аминокислотные остатки) [21]. Высокотрансформирующий вариант LMP1-Сао содержит семь 11-членных повторов и не содержит 5-членных вставок.
На рис. 2 представлены результаты анализа 11 аминокислотных повторов и пятичленных вставок С-терминальной области белка в образцах LMP1 татар.
Большинство образцов уникального варианта LMP1-TatK (5/7) характеризуется семью повторами, состоящими из 11 а.к. и, за исключением двух (Т33 и Т45) из семи образцов, отсутствием вставок 5 а.к. между 2-м и 3-м повторами, что характерно для высокотрансформирующего варианта Сао. Для большинства образцов варианта TatK (5/7) также типична точковая мутация – замена аспарагиновой кислоты на глицин (D → G). Для большей части остальных татарских образцов (23/34) были характерны вставки 5 а.к. между 2-м и 3-м повторами, типичные для прототипного варианта B95.8, а также частые четырёхчленные 11 а.к. повторы (17/34) и достаточно редко встречающаяся точковая мутация D → G, отсутствующая у прототипного варианта B95.8. Таким образом, можно сделать вывод, что по структуре аминокислотных повторов и вставок в С-терминальной области образцы варианта LMP1-TatK существенно отличаются от остальных образцов LMP1 этнических татар и схожи с высокотрансформирующим вариантом LMP1-Сао.
Обнаружение среди населения Татарстана персистенции штаммов ВЭБ, кодирующих высокотрансформирующий вариант LMP1-Сhina1 и Сао-подобный вариант LMP1-TatK (отсутствующий у представителей славян), стимулировало нас сравнить показатели заболеваемости и смертности от новообразований, в состав которых входят опухоли, ассоциированные с ВЭБ, для населения Республики Татарстан и Москвы (рис. 3).
По данным рис. 3, а, заболеваемость раком желудка среди населения Татарстана была выше (116,7), чем среди населения Москвы (90,0) при статистически значимом различии (p<0,05). Показатели заболеваемости злокачественными опухолями, возникающими в полости рта и глотки, были также несколько выше среди татарского населения, хотя заболеваемость злокачественными лимфомами оказалась выше у москвичей. Различия во всех этих случаях были статистически недостоверными (p>0,05). Показатели смертности от всех вышеперечисленных патологий (см. рис. 3, б) были несколько выше для населения Татарстана, но различия также были статистически недостоверными. Таким образом, можно сделать вывод, что персистенция среди населения Татарстана штаммов ВЭБ, кодирующих высокотрансформирующий вариант LMP1-Сао и Сао-подобный вариант LMP1-TatK, не оказывает влияния на показатели заболеваемости и смертности от злокачественных патологий, включая случаи, ассоциированные с ВЭБ. Такой вывод представляется логичным, учитывая тот факт, что процент опухолей, в возникновении которых ВЭБ играет роль этиологического фактора, как правило, крайне незначителен (1–3%), особенно для неэндемичных регионов, каковыми являются Республика Татарстан и центральные области России, в частности Москва и Московская область.
Таблица 1. Полиморфизм LMP1 в штаммах вируса Эпштейна–Барр, циркулирующих среди московских славян и казанских татар
Table 1. Polymorphism of LMP1 in Epstein–Barr virus strains circulating among Slavs and Tatars

Примечание. LMP1 – латентный мембранный белок 1 вируса Эпштейна–Барр; B95.8/A, Med-, China1, NC – варианты онкобелка LMP1.
Note. LMP1 – Epstein–Barr virus latent membrane protein 1; B95.8/A, Med-, China1, NC – LMP1 oncoprotein variants.
Таблица 2. Специфические мутации С-терминального домена у 7 образцов LMP1-TatK
Table 2. Specific mutations of the C-terminal domain in 7 samples of LMP1-TatK

Таблица 3. Варианты LMP1 у больных ВЭБ-ассоциированной патологией и здоровых лиц в России
Table 3. LMP1 variants in Russian patients with EBV-associated pathologies and healthy individuals


Рис. 1. Филогенетический анализ образцов LMP1 из смывов полости рта татар. Филогенетическое древо С-концевых последовательностей 41 образца LMP1, полученных от этнических татар, сконструировано с использованием метода «присоединения соседа» (neighbour joining). ВК – вне классификации.
Fig. 1. Phylogenetic analysis of LMP1 sequences isolated from oral washes obtained from the Tatars. The phylogenetic tree of 41 sequences of LMP1 C-terminal domain obtained from ethnic Tatars was constructed using the of neighbor-joining method. OC – out of classification.

Рис. 2. Варианты 11 а.к. повторов в домене 253–306 а.к. С-терминального сайта LMP1 в изолятах ВЭБ от этнических татар. Т – обозначение образцов LMP1; PQDPDNTDDNG – повторяющиеся элементы 11 а.к.; PHDPL – вставка 5 а.к. последовательностей; D→G – точечные мутации в области 11 а.к.
Fig 2. Variants of 11 aa repeats of the C-terminal domain in LMP1 isolates from Tatars. T – denotes the LMP1 isolates; PQDPDNTDDNG – 11 aa repeated elements; PHDPL – 5 aa insertion; D→G – site-specific mutations in 11 aa region.

Рис. 3. Стандартизованные показатели заболеваемости (а) и смертности (б) от новообразований, включая случаи, ассоциированные с вирусом Эпштейна–Барр, среди населения Татарстана и Москвы в 2019 г. (оба пола).
Fig. 3. Standardized morbidity (a) and mortality (b) for some types of tumors including cases associated with the EBV among populations of Tatarstan and Moscow in 2019 (both of sexes).
Обсуждение
Результаты проведённых исследований позволили нам высказать предположение, что штамм ВЭБ-TatK, содержащий уникальный вариант LMP1, LMP1-TatK, с двумя делециями 5 а.к., в кодонах 312–316 и 382–386 а.к., относится к древним вариантам вируса. В пользу высказанного предположения, по-видимому, свидетельствует тот факт, что ВЭБ-TatK обнаружен только у представителей одного из древнейших этносов страны, татар, генетически ведущих свой род от монголо-татарских племён, сформировавших Казанское ханство в Поволжье после выделения его из состава Золотой Орды ещё в 1438 г. Указанный штамм вируса не был обнаружен ни среди представителей этнических славян, ни в компьютерной базе данных. Не выявлен он и у россиян, представителей других национальностей, при изучении образцов опухоли, крови и смывов полости рта от больных ВЭБ-ассоциированным раком носоглотки, больных с другими опухолями полости рта, раком желудка, лимфомой Ходжкина и другими патологиями, а также доноров крови.
Однако следует признать, что указанных аргументов недостаточно для утверждения древности штамма ВЭБ-TatK. Известно, что миллионы лет назад в процессе эволюции вновь возникающие вирусы оказывались более приспособленными к изменяющимся условиям окружающей среды, чем их предшественники. Старые штаммы, скорее всего, постепенно вытеснялись новыми, более жизнеспособными штаммами. Определённую ясность в этот непростой вопрос могло бы, вероятно, внести обнаружение родственных ВЭБ-TatK штаммов вируса у предшественников человека – человекообразных обезьян, которые предположительно могли сохранить до наших дней аналоги древних штаммов вируса. Действительно, у приматов ВЭБ-подобные лимфокриптовирусы ассоциированы с лимфопролиферативными нарушениями и злокачественными новообразованиями, схожими с описанными у людей с иммунодефицитными состояниями [22]. Известно, также, что ВЭБ-подобные лимфокриптовирусы выявлены у человекообразных обезьян. Два типа лимфокриптовирусов, схожих с ВЭБ 1-го и 2-го типа у людей, были обнаружены у западных равнинных горилл и орангутангов, а лимфокриптовирус, схожий с ВЭБ 1-го типа, был продемонстрирован у шимпанзе [23][24].
В связи с полученными данными возникает вопрос, не является ли обнаруженный нами уникальный штамм ВЭБ с вариантом LMP1-TatK так называемым локальным Волжским штаммом вируса, подобно географически привязанным штаммам ВЭБ в классификации R. Edwards и соавт. [17]. Как известно, эта классификация состоит из средиземноморских (Med+ и Med-), китайских (China1, 2, 3) и американских (NC и Alaskan) вариантов LMP1. Ответ на поставленный нами вопрос, вероятно, можно будет получить, изучив изоляты ВЭБ от других этнических представителей Поволжья, в частности, жителей республик Калмыкия, Марий Эл и Чувашия.
В связи с обнаружением штамма ВЭБ, содержащего LMP1 с ранее не встречавшимися мутациями, делециями 5 а.к. в кодонах 312–316 и 382–386 а.к., также представляется важным выяснить функциональные свойства этого онкогена, его трансформирующий потенциал и другие свойства, что станет предметом наших дальнейших исследований. Дело в том, что последствия эволюционных изменений в гене LMP1 практически не изучены. Неизвестно, являются ли новые или рекомбинантные варианты LMP1 более патогенными или специфически ответственными за высокий уровень заболеваемости ВЭБ-ассоциированными патологиями, в том числе у людей с ослабленным иммунитетом. В настоящее время мало данных для ответов на поставленные вопросы. Можно предположить, что новый вариант аминокислотных последовательностей LMP1, благодаря включению двух уникальных делеций 5 а.к. в кодонах 312–316 и 382–386, может изменить функцию или иммуногенность белка LMP1, тем самым изменив его роль в патогенезе заболевания. Попытка выявить повышенные показатели заболеваемости или смертности от злокачественных опухолей, включая ассоциированные с ВЭБ, среди жителей Татарстана, носителей уникального штамма вируса, ВЭБ-TatK, оказалась несостоятельной, скорее всего, в силу низкого числа ВЭБ-ассоциированных случаев в каждой изученной нозологической группе опухолей. Изучение патогенеза и клинических проявлений опухолевого процесса в случаях новообразований, ассоциированных с ВЭБ-TatK, возможно, прольёт свет на функциональные и другие свойства LMP1-TatK вируса.
Благодарность. Авторы выражают признательность и глубокую благодарность С.В. Петрову, доктору медицинских наук, профессору Республиканского клинического онкологического диспансера Министерства здравоохранения Республики Татарстан, за помощь в организации и сборе биологического материала от представителей татар в третьем поколении.
About the authors
Kseniya V. Smirnova
PhD, Head of the Laboratory of Viral Carcinogenesis of the N.N. Blokhin NMRC of Oncology, Moscow, 115478, Russia
Author for correspondence.
Email: skv.lab@yandex.ru
ORCID iD: 0000-0001-6209-977X
Natalya B. Senyuta
Research Institute of Carcinogenesis, N.N. Blokhin National Medical Research Center of Oncology
Email: noemail@neicon.ru
ORCID iD: 0000-0001-8915-8274
Russian Federation
Alexandra K. Lubenskaya
Research Institute of Carcinogenesis, N.N. Blokhin National Medical Research Center of Oncology
Email: noemail@neicon.ru
ORCID iD: 0000-0003-3953-7449
Russian Federation
Tatyana E. Dushenkina
Research Institute of Carcinogenesis, N.N. Blokhin National Medical Research Center of Oncology
Email: noemail@neicon.ru
ORCID iD: 0000-0001-8279-514X
Russian Federation
Vladimir E. Gurtsevich
Research Institute of Carcinogenesis, N.N. Blokhin National Medical Research Center of Oncology
Email: noemail@neicon.ru
ORCID iD: 0000-0003-1840-4364
Russian Federation
References
- Koonin E.V., Dolja V.V., Krupovic M. Origins and evolution of viruses of eukaryotes: The ultimate modularity. Virology. 2015; 479-480: 2-25. DOI: http://doi.org/10.1016/j.virol.2015.02.039
- Ehlers B., Spiess K., Leendertz F., Peeters M., Boesch C., Gatherer D., McGeoch D.J. Lymphocryptovirus phylogeny and the origins of Epstein-Barr virus. J.Gen. Virol. 2010; 91: 630-642. doi: 10.1099/vir.0.017251-0
- Zhdanov V.M., Tikchonenko T.I. Viruses as a factor of evolution: exchange of genetic information in the biosphere. Adv. Virus Res. 1974; 19: 361-94. DOI: http://doi.org/10.1016/s0065-3527(08)60664-8
- Forterre P. Giant viruses: conflicts in revisiting the virus concept. Intervirology. 2010; 53(5): 362-78. DOI: http://doi.org/10.1159/000312921
- Krupovic M., Dolja V.V., Koonin E.V. Origin of viruses: primordial replicators recruiting capsids from hosts. Nat. Rev. Microbiol. 2019; 17(7): 449-58. DOI: http://doi.org/10.1038/s41579-019-0205-6
- Leppard K., Dimmock N., Easton A. Introduction to Modern Virology. Oxford: Blackwell Publishing Ltd; 2007.
- Mahy B.W. Strategies of virus persistence. Br. Med. Bull. 1985; 41(1): 50-5. DOI: http://doi.org/10.1093/oxfordjournals.bmb.a072024
- Mahy B.W. Human viral infections: an expanding frontier. Antiviral Res. 1997; 36(2): 75-80. DOI: http://doi.org/10.1016/s0166-3542(97)00042-9
- Walling D.M., Shebib N., Weaver S.C., Nichols C.M., Flaitz C.M., Webster-Cyriaque J. The molecular epidemiology and evolution of Epstein-Barr virus: sequence variation and genetic recombination in the latent membrane protein-1 gene. J. Infect. Dis. 1999; 179(4): 763-74. DOI: http://doi.org/10.1086/314672
- Itakura O., Yamada S., Narita M., Kikuta H. High prevalence of a 30-base pair deletion and single-base mutations within the carboxy terminal end of the LMP-1 oncogene of Epstein-Barr virus in the Japanese population. Oncogene. 1996; 13(7): 1549-53.
- Kingma D.W., Weiss W.B., Jaffe E.S., Kumar S., Frekko K., Raffeld M. Epstein-Barr virus latent membrane protein-1 oncogene deletions: correlations with malignancy in Epstein-Barr virus-associated lymphoproliferative disorders and malignant lymphomas. Blood. 1996; 88(1): 242-51.
- Falk K., Gratama J.W., Rowe M., Zou J.Z., Khanim F., Young L.S., et al. The role of repetitive DNA sequences in the size variation of Epstein-Barr virus (EBV) nuclear antigens, and the identification of different EBV isolates using RFLP and PCR analysis. J. Gen. Virol. 1995; 76(Pt. 4): 779-90. DOI: http://doi.org/10.1099/0022-1317-76-4-779
- Walling D.M., Raab-Traub N. Epstein-Barr virus intrastrain recombination in oral hairy leukoplakia. J. Virol. 1994; 68(12): 7909-17.
- Гурцевич В.Э., Яковлева Л.С., Щербак Л.Н., Гончарова Е.В., Смирнова К.В., Дидук С.В. и др. Сиквенсные варианты онкогена LMP1 у больных опухолями полости рта, ассоциированными и не ассоциированными с вирусом Эпштейна-Барр. Молекулярная биология. 2013; 47(6): 987. DOI: http://doi.org/10.7868/S0026898413050042
- Parra B., Slots J. Detection of human viruses in periodontal pockets using polymerase chain reaction. Oral. Microbiol. Immunol. 1996; 11(5): 289-93. DOI: http://doi.org/10.1111/j.1399-302x.1996.tb00183.x
- Hahn P., Novikova E., Scherback L., Janik C., Pavlish O., Arkhipov V., et al. The LMP1 gene isolated from Russian nasopharyngeal carcinoma has no 30-bp deletion. Int . J. Cancer. 2001; 91(6): 815-21. DOI: http://doi.org/10.1002/1097-0215(200002)9999:9999<::AIDIJC1122>3.0.CO;2-W
- Edwards R.H., Seillier-Moiseiwitsch F., Raab-Traub N. Signature amino acid changes in latent membrane protein 1 distinguish Epstein-Barr virus strains. Virology. 1999; 261(1): 79-95. DOI: http://doi.org/10.1006/viro.1999.9855
- Miller W.E., Edwards R.H., Walling D.M., Raab-Traub N. Sequence variation in the Epstein-Barr virus latent membrane protein 1. J. Gen. Virol. 1994; 75(Pt. 10): 2729-40. DOI: http://doi.org/10.1099/0022-1317-75-10-2729
- Каприн А.Д., Старинский В.В., Петрова Г.В. Состояние онкологической помощи населению России в 2018 году. М.; 2019.
- Senyuta N., Yakovleva L., Goncharova E., Scherback L., Diduk S., Smirnova K., et al. Epstein-Barr virus latent membrane protein 1 polymorphism in nasopharyngeal carcinoma and other oral cavity tumors in Russia. J. Med. Virol. 2014; 86(2): 290-300. DOI: http://doi.org/10.1002/jmv.23729
- Kanai K., Satoh Y., Saiki Y., Ohtani H., Sairenji T. Difference of Epstein-Barr virus isolates from Japanese patients and African Burkitt’s lymphoma cell lines based on the sequence of latent membrane protein 1. Virus Genes. 2007; 34(1): 55-61. DOI: http://doi.org/10.1007/s11262-006-0010-y
- Voevodin A.F., Hirsch I. Immunoprecipitation of Epstein-Barr virus (EBV)-specific proteins by prelymphomatous and normal baboon sera containing antibodies reactive with EBV early antigen. Acta Virol. 1985; 29(3): 242-6.
- Ehlers B., Spiess K., Leendertz F., Peeters M., Boesch C., Gatherer D., et al. Lymphocryptovirus phylogeny and the origins of EpsteinBarr virus. J. Gen. Virol. 2010; 91(Pt. 3): 630-42. DOI: http://doi.org/10.1099/vir.0.017251-0
- Ehlers B., Ochs A., Leendertz F., Goltz M., Boesch C., Mätz-Rensing K. Novel simian homologues of Epstein-Barr virus. J. Virol. 2003; 77(19): 10695-9. DOI: http://doi.org/10.1128/jvi.77.19.10695-10699.2003
Supplementary files