Prevalence and genetic diversity of the Alongshan virus (Flaviviridae) circulating in ticks in the south of Eastern Siberia
- Authors: Kartashov M.Y.1, Krivosheina E.I.1, Kurushina V.Y.1, Moshkin A.B.2, Khankhareev S.S.3, Biche-ool C.R.4, Pelevina O.N.5, Popov N.V.6, Bogomazova O.L.6, Ternovoi V.A.1
-
Affiliations:
- State Research Center of Virology and Biotechnology «Vector»
- Chita anti-plague station
- Department of the Federal Service for Supervision of Consumer Rights Protection and Human Welfare of the Republic of Buryatia
- Center of Hygiene and Epidemiology in the Republic of Tuva
- Center for Hygiene and Epidemiology in the Republic of Khakassia
- Center for Hygiene and Epidemiology in the Irkutsk region
- Issue: Vol 69, No 2 (2024)
- Pages: 151-161
- Section: ORIGINAL RESEARCHES
- URL: https://virusjour.crie.ru/jour/article/view/16612
- DOI: https://doi.org/10.36233/0507-4088-223
- EDN: https://elibrary.ru/jwvfoe
- ID: 16612
Cite item
Full Text
Abstract
Introduction. Tick-borne infections are of great importance for many regions of Russia, including Eastern Siberia. This unfavorable epidemiological situation can be characterized not only by the circulation of well-known tick-borne infections, but also by the identification of new pathogens, the role of which remains little or generally unexplored. Multicomponent flavi-like viruses can cause infectious diseases in humans and pose a threat to public health.
The purpose of the study was the identification and molecular genetic characterization of the Alongshan virus (Flaviviridae, ALSV) isolates, transmitted by ticks in the south of Eastern Siberia.
Materials and methods. Total 1060 ticks were collected and analyzed from the territory of the Republics of Khakassia, Tuva, Buryatia, Irkutsk Region and Transbaikal Territory (Zabaykalsky Krai) in the spring-summer period 2023. ALSV RNA was detected by RT-PCR followed by nucleotide sequence determination and phylogenetic analysis for each segment of the genome.
Results. The ALSV infection rate in Ixodes persulcatus ticks collected in the Republic of Khakassia was 3.3% (95% CI: 1.4–7.5); in Irkutsk Oblast – 1.0% (95% CI: 0.3–3.7); in the Republic of Tuva – 0.9% (95% CI: 0.3–3.4) and in Transbaikal Krai – 0.7% (95% CI: 0.2–3.6). Sequences of all four segments of ALSV genetic variants circulating in I. persulcatus ticks in the south of Eastern Siberia are grouped with sequences found in China and clustered into the Asian subgroup transmitted by taiga ticks. The level of difference in the nucleotide sequences of genome fragments among the identified genetic variants of ALSV ranged from 2 to 3%.
Conclusion. The article shows the widespread distribution of ALSV in I. persulcatus ticks in the Republics of Khakassia and Tyva, Irkutsk Oblast and Transbaikal Territory. The obtained data actualize monitoring of changes in the area of distribution of potentially dangerous for humans flavi-like viruses and their vectors.
Full Text
Введение
Флавивирусы, переносимые клещами, широко распространены по всему миру и представляют серьезную медицинскую проблему, вызывая значительное количество инфекционных заболеваний среди людей [1]. Среди флавивирусов, переносчикам которых являются клещи, на территории России встречаются вирус Повассан, вирус омской геморрагической лихорадки, вирус шотландского энцефаломиелита овец (Louping ill virus) и вирус клещевого энцефалита [2–4]. Несмотря на достаточно большое видовое разнообразие, геном всех флавивирусов имеет типичное строение и представляет собой несегментированную оц(+)РНК длиной около 11 kb, кодирующую одну протяженную открытую рамку считывания, по краям которой находятся 5’- и 3’-нетранслируемые области [5]. Однако за последнее десятилетие были выделены флавиподобные вирусы, которые отличаются от «классических» флавивирусов принципиально другим строением генома и представляют собой обособленную группу в семействе Flaviviridae. Такие вирусы обладают сегментированным одноцепочечным РНК-геномом положительной полярности, в структуре которого имеется гомология с РНК-зависимой РНК-полимеразой (NS5) и хеликазой (NS3) «классических» флавивирусов. В эту группу входят вирус Алонгшан (Alongshan virus, ALSV), вирус Джингмен (Jingmen tick virus), вирус Янггоу (Yanggou tick virus) и другие вирусы группы Jingmenvirus.
Геномы многокомпонентных флавиподобных вирусов включают от 4 (характерно для большинства вирусов, изолированных из клещей, летучих мышей, обезьян и человека) до 5 (встречается у вирусов, выделенных из комаров) сегментов [6]. Сегмент 1 кодирует неструктурный белок NS5-like, имеющий гомологию с белком NS5 (РНК-зависимая РНК-полимераза) классических флавивирусов. Сегмент 3 кодирует неструктурный белок, имеющий высокую степень гомологии с флавивирусным белком NS3. Белок NS3, наряду с NS5, играет центральную роль в репликации вируса и является одним из наиболее изученных неструктурных белков. N-концевой домен NS3 обладает протеазной активностью, а C-концевой домен выполняет функцию хеликазы. Протеиназная активность необходима для процессинга полипротеина, хеликазный домен участвует в кэппировании и синтезе вирусной РНК. На сегодняшний день хорошо изучена структура белка NS3 у большинства несегментированных флавивирусов и показана высокая гомология не только в строении, но также в механизмах гидролиза АТФ, распознавания и раскручивания РНК [7]. Структурные белки VP1, VP2 VP3 закодированы в сегментах 2 и 4 и не имеют известных гомологов как среди семейства Flaviviridae, так и среди других известных на сегодняшний день вирусов. Сегмент 2 у ALSV кодирует предполагаемые гликопротеины VP1a и VP1b, а также маленький белок с тремя трансмембранными доменами, функция которого не известна. Белки VP2 (предполагаемый капсидный белок) и VP3 (предполагаемый вирусный мембранный белок) закодированы в сегменте 4 и имеют частично перекрывающиеся рамки трансляции.
Благодаря достижениям в области высокопроизводительного секвенирования и вирусной метагеномики варианты флавиподобных вирусов были обнаружены в различных образцах на территории 4 континентов: в Азии (Китай, Лаос, Япония, Турция), Европе (Финляндия, Италия, Косово, Румыния, Россия), Южной Америке (Бразилии, Тринидад и Тобаго, Колумбия) и Африке (Уганда, Кения и Гвинея) (табл. 1). На территории России также показано широкое распространение сегментированных флавиподобных вирусов (ALSV и вирус Янггоу). Так, генетический материал ALSV был обнаружен в образцах голодных клещей I. persulcatus, I. ricinus, Dermacentor reticulatus и D. nuttalli, собранных в Калининградской, Ульяновской, Челябинской областях, а также в республиках Карелия, Татарстан, Горный Алтай и Тыва [8–10].
Таблица 1. Распространение ALSV в мире
Table 1. Distribution of ALSV in the world
Вид хозяина Host species | Страна Country | Номер последовательности в GenBank GenBank accession number | Ссылка Reference |
I. persulcatus | Китай China | MH158439–MH158440, MH678646–MH678648 | [11] |
I. persulcatus | Китай China | MT246198–MT246199, MT514916–MT514917, MT536950–MT536953 | |
I. ricinus | Финляндия Finland | MN107153–MN107160 | [12] |
I. ricinus | Франция France | MN095519–MN095522 | [13] |
I. ricinus | Сербия Serbia | MT822179–MT822180 | [14] |
I. ricinus, D. reticulatus | Германия Germany | MW094148–MW094151 | [15] |
I. ricinus | Швейцария Switzerland | OP921096–OP921099 | [16] |
Hae. longicornis | Китай China | MZ676705 | |
I. persulcatus, D. nuttalli, Hae. concinna, I. ricinus, D. reticulatus | Россия Russia | MN604229, MN648770–MN648777, MT210218–MT210225, MW525311, MW525312, MW525314–MW525321, MW52531–MW52533, MW525284–MW525310, MW556738–MW556741, MW584331 | |
Culex tritaeniorhynchus | Китай China | MK213942 | |
Bos taurus | Китай China | MK122718, MK122720, MN218594, MN218595 | [18] |
Homo sapiens | Китай China | MH158415–MH158438 | [18] |
В настоящее время показана патогенность многокомпонентных флавиподобных вирусов для сельскохозяйственных животных и человека. Однако эти сведения носят фрагментарный и ограниченный характер. Не исключена вероятность того, что роль флавиподобных вирусов в инфекционной патологии домашних животных и человека может быть существенно более значимой, чем принято считать. Так, ALSV был выделен из крови пациента с лихорадкой на северо-востоке Китая [9]. У 86 из 384 пациентов с лихорадкой и 18 пациентов с укусами клещей в анамнезе был выявлен ALSV с помощью молекулярно-генетических и серологических диагностических тестов. Пациенты, инфицированные ALSV, в анамнезе имели лихорадку, головную боль и другие симптомы, которые напоминают проявления других инфекций, переносимых клещами. Близкородственный ALSV вирус Джингмен был обнаружен в крови приматов в Уганде, в крови крупного рогатого скота в Бразилии и в крови пациентов с диагнозом «геморрагическая лихорадка Крым-Конго» в Косово [19–21]. Эти результаты также указывают на то, что сегментированные флавиподобные вирусы могут являться причиной инфекционных заболеваний человека и создавать угрозу для здоровья населения.
Проблема инфекций, переносчиками которых являются иксодовые клещи, актуальна для многих регионов России, в том числе и для Восточной Сибири, где отмечается беспрецедентный рост заболеваемости населения вирусным клещевым энцефалитом, иксодовым клещевым боррелиозом и клещевым риккетсиозом. Показано, что экосистемы юга Восточной Сибири и севера Монголии являются оптимальными для существования разнообразных переносчиков и возбудителей трансмиссивных инфекций, переносимых клещами [22]. Обширная территория юга Восточной Сибири граничит или территориально близка к внутренним районам Китая, где впервые и была обнаружена циркуляция ALSV. Тем не менее в литературе имеются данные лишь об единичной находке ALSV на территории Республики Тыва.
Цель данной работы ‒ поиск и молекулярно-генетическая характеристика изолятов ALSV, переносимых клещами, на юге Восточной Сибири.
Материалы и методы
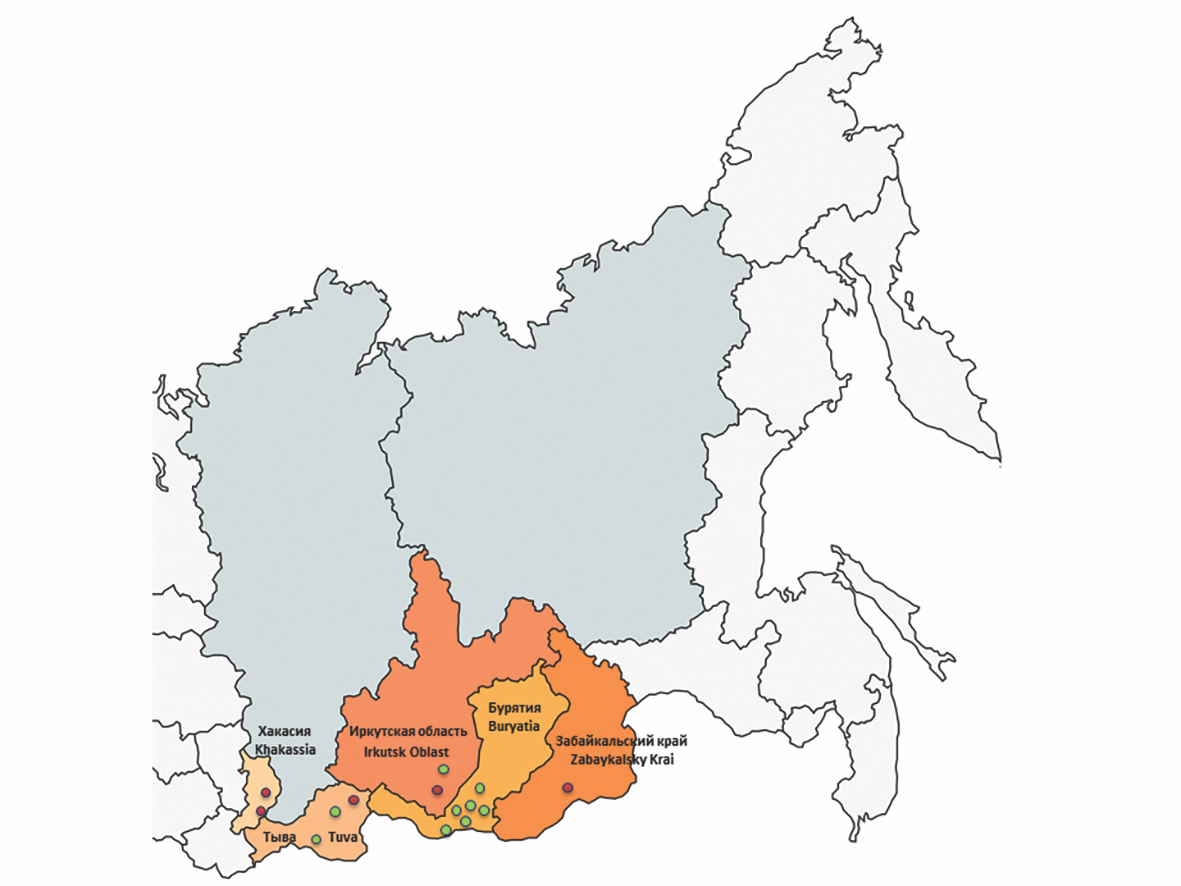
Всего для исследования было проанализировано 1060 индивидуальных проб клещей, отловленных на территории республик Хакасия, Тыва, Бурятия, Иркутской области и Забайкальского края в весенне-летний период 2023 г. (рис. 1).
Рис. 1. Места отлова исследуемых клещей. Красным цветом отмечены локации, где в собранных клещах была выявлена РНК ALSV; зеленым ‒ локации, где РНК ALSV не была обнаружена.
Fig. 1. Locations where the studied ticks were captured. Locations where ticks positive for ALSV RNA were collected are marked in red; Locations where no ALSV RNA was detected are indicated in green.
Сбор образцов клещей производили с растительности методом «на флаг». Клещей дважды отмывали 70% этанолом для удаления внешних загрязнений и наружной микрофлоры, после чего хранили при температуре −80 °C до проведения дальнейших исследований. Видовую принадлежность клещей устанавливали по морфологическим характеристикам согласно определителю с последующей таксономической верификацией проб, положительных на наличие ALSV, путем определения нуклеотидной последовательности фрагмента гена цитохромоксидазы, локализованного в митохондриальном геноме клеща.
Гомогенизацию полученных образцов осуществляли с использованием лабораторного гомогенизатора TissueLyserLT (Qiagen, Германия) в 300 мкл стерильного физраствора. Выделение суммарной РНК из исследуемых образцов проводили методом фенол-хлороформной экстракции с использованием TRIzol Reagent.
Таблица 2. Используемые в работе праймеры
Table 2. Primers used in this work
Мишень Target | Праймер Primer | Структура праймера (5’→3’) Primer sequence (5’→3’) | Длина Amplicon length (bp) | Тотжига (°С) Annealing T (°С) | Ссылка Reference |
cox1 | IpCX _f | ATTAGGAGCACCTGATATAGCTTTCCC | 660 | 60 | [24] |
IpCX _r | GCTGTAAATAAGCTCGAGTGTCGATAA | ||||
Сегмент 2 Segment 2 | Miass_gly_3F | TGGATCAGCTCACACCACAC | 333 | 53 | [25] |
Miass_gly_3R | TCACCGTCACAGTGGAATGG | ||||
Сегмент 1 Segment 1 | MiassF | GGTACACGGACCTGGGATCCTATTG | 825 | 50 | |
MiassR | TCTCTGACTCCTGTTCTAATC | ||||
Сегмент 2 Segment 2 | AL2_2F | CTTGCTACATCGGAATCATGCC | 1091 | 56 | Данная работа Current study |
AL2_2R | GATAAGCCCTCTCGATACCTC | ||||
Сегмент 3 Segment 3 | AL3_2F | TGCTGTCCATAGCAATCATACC | 865 | 56 | |
AL3_2R | GTAGGACACGTCCTTTGCGA | ||||
Сегмент 4 Segment 4 | AL4_2F | GGAAGAGCTGGATACCGAACTG | 1274 | 56 | |
AL4_2R | TGCCAGATGTGTAGCTTCCC |
Скрининг полученных образцов на наличие РНК флавиподобных вирусов выполняли с помощью метода полимеразной цепной реакции с обратной транскрипцией (ОТ-ПЦР), используя праймеры Miass_gly_3F/Miass_gly_3R, комплементарные фрагменту генома сегмента 2 (табл. 2). Постановку ОТ-ПЦР производили на термоциклере Т-1000 («Bio-Rad», США) в 25 мкл реакционной смеси. Для положительных образцов были получены ампликоны, частично перекрывающие все 4 сегмента генома ALSV. Для исключения возможной интеграции фрагментов вирусного генома ALSV в геном клеща [23] с положительными пробами параллельно была проведена ПЦР без этапа обратной транскрипции, в результате которой ни один ампликон наработан не был.
Детекцию полученных ампликонов проводили методом гель-электрофореза в 2% агарозном геле в трис-ацетатном буфере, содержащем 0,1% бромида этидия. Секвенирование по Сэнгеру осуществляли с использованием набора BigDye Terminatorv.3.1 Cycle Sequencing Kit (Applied Biosystems, США). Определение нуклеотидных последовательностей выполняли на основе капиллярного электрофореза с помощью автоматического секвенатора 3130xl Genetic Analyzer (Applied Biosystems, США). Анализ полученных нуклеотидных последовательностей проводили с использованием программного продукта UniproUGENE v. 1.48. Филогенетический анализ нуклеотидных последовательностей исследуемых фрагментов генома проводили методом максимального правдоподобия с использованием модели эволюции Tamura-Nei в программе MEGA Х. Показатели статистической надежности узлов филогенетического дерева рассчитывали с помощью бутстреп-анализа с использованием 1000 случайных реплик; 95% доверительный интервал (ДИ) уровня инфицированности клещей изучаемыми патогенами рассчитывали с использованием онлайн-сервиса (https://www.pedro.org.au/english/downloads/confidence-interval-calculator).
Результаты
Все образцы были протестированы методом ОТ-ПЦР с использованием скрининговых праймеров для сегмента 2 генома ALSV. Встречаемость РНК среди клещей I. persulcatus, собранных в Республике Хакасия, составила 3,3% (95% ДИ 1,4–7,5). Аналогичный показатель для Иркутской области составил 1% (95% ДИ 0,3–3,7), для Республики Тыва – 0,9% (95% ДИ 0,3–3,4) и для Забайкальского края – 0,7% (95% ДИ 0,2–3,6). Среди клещей D. nuttalli, отловленных на территории Республики Бурятия, РНК ALSV обнаружено не было, что, вероятно, связано с гораздо более низкой вирусофорностью данного вида клещей (табл. 3).
Таблица 3. Встречаемость ALSV в исследуемых пробах клещей, обитающих на юге Восточной Сибири
Table 3. Detection rates of ALSV in the studied ticks living in the south of Eastern Siberia
Регион Region | Вид клещей Tick species | Район District | Географические координаты Latitude, Longitude | Число проанализированных образцов Number of analysed ticks | Число положительных образцов Number of virus-positive |
Забайкальский край Zabaykalsky Krai | I. persulcatus | Дульдургинский район, Национальный парк Алханай Duldurga district, Alkhanai National Park | 50.873671, 113.398280 | 150 | 1 |
Республика Хакасия Republic of Khakassia | I. persulcatus | Бейский район, долина реки Табат Beysky district, Tabat river valley | 52.947886, 90.721274 | 100 | 2 |
г. Саяногорск, дачное общество Большой Карак Sayanogorsk, Bolshoi Karak | 53.032989, 91.415743 | 50 | 3 | ||
Иркутская область Irkutsk region | I. persulcatus | Иркутский район, Байкальский тракт, 20 км Irkutsk district, Baikalsky tract, 20 km | 52.189528, 104.489475 | 100 | 0 |
Иркутский район, Байкальский тракт, 26 км Irkutsk district, Baikalsky tract, 26 km | 52.162501, 104,524191 | 100 | 2 | ||
Республика Тыва Republic of Tyva | I. persulcatus | Каа-Хемский район, местечко Копту Kaa-Khem district, Koptu | 51.757770, 95.418196 | 20 | 0 |
I. persulcatus | Тоджинский район, перевал Тожу Todzhinsky district, Tozhu pass | 52.701197, 96.802060 | 170 | 2 | |
I. persulcatus | Тандинский район, местечко Дургенский каньон Tandinsky district, Durgen Canyon | 51.049461, 94.646579 | 20 | 0 | |
Республика Бурятия Republic of Buryatia | D. nuttalli | Джидинский район, с. Гэгэтуй Dzhidinsky district, village Gegetui | 50.683752, 105.181199 | 50 | 0 |
Тарбагатайский район, с. Саратовка Tarbagatai district, village Saratovka | 51.537704, 107.346951 | 50 | 0 | ||
Тарбагатайский район, гора Спящий лев Tarbagatai district, Mount «Sleeping Lion» | 51.537704, 107346951 | 50 | 0 | ||
Тарбагатайский район, окрестности с. Саянтуй Tarbagatai district, village Sayantui | 51,694972, 107.448139 | 50 | 0 | ||
Иволгинский район, окрестности с. Ключи Ivolginsky district, village Kluchi | 51.687606, 107.173963 | 50 | 0 | ||
Селенгинский район, окрестности г. Гусиноозерск Selenginsky district, Gusinoozersk | 50.632847, 105.382886 | 50 | 0 | ||
Заиграевский район, окрестности с. Эрхирик, Онохой Zaigraevsky district, village Erhirik | 51.898049, 108.006255 | 50 | 0 |
Выявленные в работе нуклеотидные последовательности были депонированы в базу данных GenBank под номерами OR620965–OR620974 для фрагмента сегмента 1, OR620975–OR620984 для фрагмента сегмента 2, OR620985–OR620994 для фрагмента сегмента 3 и OR620995–OR621004 для фрагмента сегмента 4.
Филогенетические деревья демонстрируют, что генетические варианты ALSV, циркулирующие в клещах I. persulcatus на территории юга Восточной Сибири, по всем четырем сегментам группируются с последовательностями, обнаруженными в Китае (рис. 2).
Рис. 2. Филогенетические деревья выявленных вариантов ALSV.
Fig. 2. Phylogenetic trees of identified ALSV variants.
Различия в нуклеотидных последовательностях между выявленными вариантами по определенным в работе фрагментам генома составили от 2 до 3%. Различия в выведенных аминокислотных последовательностях варьировали от 0,5 до 1,5% (рис. 3).
Рис. 3. Уровень различия нуклеотидных и аминокислотных последовательностей выявленных вариантов ALSV на юге Восточной Сибири.
Fig. 3. Level of differences in nucleotide and amino acid sequences of identified ALSV variants in the south of Eastern Siberia.
Генетическое сходство нуклеотидных последовательностей между полученными в настоящем исследовании вариантами ALSV и рядом прототипных изолятов флави- и флавиподобных вирусов приведено в табл. 4.
Таблица 4. Уровень гомологии нуклеотидной последовательности (%) ряда выявленных вариантов ALSV по сравнению с прототипными изолятами флави- и флавиподобных вирусов
Table 4. Level of nucleotide sequence similarity (%) of a number of identified ALSV variants in comparison with prototype isolates of flavi- and flavi-like viruses
Прототипные изоляты Prototype isolates | Варианты ALSV, полученные в работе ALSV variants identified in this study | ||||
Chita 2023-1 | Khakassia 2023-1 | Tuva 2023-1 | Irkutsk 2023-1 | ||
ALSV H3 | Сег. 1/Seg.1 | 96,3 | 95,8 | 96,2 | 96,1 |
Сег. 2/Seg.2 | 93,9 | 94,4 | 93,9 | 93,8 | |
Сег. 3/Seg.3 | 96,3 | 97,1 | 96,4 | 96,3 | |
Сег. 4/Seg.4 | 97,3 | 96,5 | 97,3 | 96.5 | |
ALSV Miass502 | Сег. 1/Seg.1 | 89,7 | 89,8 | 89,5 | 89,4 |
Сег. 2/Seg.2 | 98,0 | 97,4 | 98 | 97,2 | |
Сег. 3/Seg.3 | 90,9 | 90,4 | 91,4 | 89,9 | |
Сег. 4/Seg.4 | 90,2 | 90,2 | 91,1 | 90,3 | |
ALSV Kuutsalo | Сег. 1/Seg.1 | 88,9 | 89,5 | 89,5 | 89,1 |
Сег. 2/Seg.2 | 91,7 | 91,6 | 91,3 | 91,5 | |
Сег. 3/Seg.3 | 90,7 | 90,7 | 91,2 | 90,6 | |
Сег. 4/Seg.4 | 91,2 | 90,9 | 91,5 | 90,8 | |
Jingmen tick virus isolate 21GZ1 | Сег. 1/Seg.1 | 75,2 | 76,5 | 75,3 | 75,2 |
Сег. 2/Seg.2 | 50,3 | 50,3 | 50,4 | 50,4 | |
Сег. 3/Seg.3 | 74,5 | 74,2 | 75,8 | 74,5 | |
Сег. 4/Seg.4 | 63,3 | 63,2 | 64,4 | 64,1 | |
Yanggou tick virus isolate Republic Altay/997/2016 | Сег. 1/Seg.1 | 73,4 | 73,2 | 74,2 | 73,7 |
Сег. 2/Seg.2 | 61,9 | 61,9 | 62,1 | 61,9 | |
Сег. 3/Seg.3 | 76,2 | 76,5 | 76,4 | 75,7 | |
Сег. 4/Seg.4 | 70,2 | 70,4 | 70,3 | 69,8 | |
Takachi virus isolate IM-OI110 | Сег. 1/Seg.1 | 74,2 | 75,2 | 74,5 | 75,3 |
Сег. 2/Seg.2 | 65,7 | 66,1 | 66,3 | 66,3 | |
Сег. 3/Seg.3 | 77,3 | 77,2 | 77,4 | 77,2 | |
Сег. 4/Seg.4 | 74,6 | 74,5 | 74,9 | 75,4 | |
Tick-borne encephalitis viru strain Zausaev | Сег. 1/Seg.1 | 54,5 | 54,7 | 54,6 | 54,3 |
Сег. 2/Seg.2 | – | – | – | – | |
Сег. 3/Seg.3 | 41,6 | 41,4 | 41,7 | 41,5 | |
Сег. 4/Seg.4 | – | – | – | – | |
Наиболее высокий уровень гомологии варианты ALSV из Восточной Сибири проявляли с китайским изолятом ALSV H3 (94–97%). Уровень гомологии изучаемых вариантов с прототипным изолятом ALSV Miass502, выделенным из клеща I. persulcatus на территории Челябинской области, составил 90–97%. Аналогичный показатель при сравнении с вариантами ALSV, выделенными из I. ricinus в Европе, колебался в пределах 90%. Гомология с последовательностями других флавиподобных вирусов (Джингмен, Янггоу, Тачен) составляла от 50 до 75% для разных сегментов генома. Гомология сегмента 1 (NS-5 like) и сегмента 3 (NS-3 like) при сравнении с геномом вирусом клещевого энцефалита составила 54 и 41%, соответственно.
Обсуждение
Полученные в настоящей работе данные об уровне встречаемости ALSV на территории южных регионов Восточной Сибири в целом соответствуют аналогичным данным, полученным для других регионов России. Наибольшая инфицированность клещей обнаружена в Хакасии (3,3%), что приближается к вирусофорности клещей из Челябинской области (4,3%) и Калининградской области (7,8%). Уровень инфицированности для других регионов Восточной Сибири (0,7–1%) соответствует ранее полученным показателям для Республики Тыва (0,6%), Республики Карелия (0,8%) и Республики Татарстан (1,4%) [8–10]. На основе полученных нами данных можно предположить наличие некоего градиента в возрастании инфицированности клещей ALSV с востока на запад, однако это предположение следует подтвердить проведением более обширного мониторинга и увеличением исследуемых проб клещей.
Согласно данным литературы, при филогенетическом анализе сегмента 2, кодирующего поверхностные структурные вирусные белки, показано, что основными факторами эволюции ALSV являются вид основного переносчика и место изоляции вируса. Предположительно изоляты ALSV разделяются в соответствии с основным видом переносчика на группы «I. ricinus» и «I. persulcatus». Группа «I. persulcatus» делится на две подгруппы: европейскую (республики Карелия и Алтай, Челябинская область) и азиатскую (Китай, республики Алтай, Тыва и Карелия, Челябинская и Ульяновская области и Алтайский край) [9]. Данное предположение нашло подтверждение в настоящем исследовании, т.е. все изоляты ALSV, циркулирующие на юге Восточной Сибири в клещах I. persulcatus, однозначно кластеризовались в азиатскую подгруппу соответствующего переносчика при анализе по каждому из сегментов генома.
В проведенной ранее работе было показано, что наибольший уровень внутривидового различия в нуклеотидных последовательностях ALSV характерен для сегмента 1, кодирующего аналог NS5 белка «классических» флавивирусов [23]. Это подтверждается на примере выявленных вариантов ALSV. Однако при анализе аминокислотных последовательностей видно, что аналоги белков NS5 и NS3 более консервативны по сравнению со структурными белками ALSV (рис. 3).
Можно предположить, что для вирусов с многокомпонентным геномом характерны события перегруппировки/рекомбинации, которые могут вносить вклад в их генетическую изменчивость. Топология филогенетических деревьев, построенных для фрагментов всех 4 сегментов, как у выявленных генетических вариантов ALSV, так и у прототипных изолятов практически совпадает. Имеющиеся данные свидетельствуют о том, что геном флавпиподобных вирусов чрезвычайно стабилен среди позвоночных и беспозвоночных. Этот факт позволяет предположить, что вирус уже хорошо адаптирован к обоим хозяевам.
Показано, что на территории Республики Тыва существуют сочетанные очаги флавивирусов (вирус клещевого энцефалита) и флавиподобных вирусов (ALSV и вирус Янггоу) [9]. В связи с широким распространением вируса клещевого энцефалита в Восточной Сибири и обнаружением в клещах данного региона ALSV, наличие таких сочетанных очагов можно предположить и на других изучаемых территориях. Это поднимает вопрос о возможной коциркуляции этих вирусов, их взаимного влияния друг на друга и коинфицировании человека при укусе клеща.
Заключение
Современная неблагополучная эпидемиологическая ситуация, сложившаяся в России в отношении инфекций, переносимых клещами, характеризуется не только ростом заболеваемости уже известными «клещевыми» инфекциями, но и выявлением новых нозологических форм и возбудителей, роль которых в региональной инфекционной патологии остается малоизученной или вообще неизученной. Данные о циркуляции флавиподобных вирусов, потенциально опасных для человека, могут служить основой для коррекции профилактических и противоэпидемических мероприятий. Для понимания актуальной обстановки, проведения регионально-ориентированной лабораторной диагностики и осуществления своевременных и адекватных профилактических мероприятий по отношению к инфекциям, переносимым клещами на территории Восточной Сибири, необходима достоверная информация о спектре циркулирующих на территории региона переносимых клещами патогенов и их генетической вариабельности. В работе показано широкое распространение вируса Алонгшан на территории республик Хакасия и Тыва, Иркутской области и Забайкальского края. Для выявленных вариантов вируса проведена молекулярно-генетическая характеристика и получены частичные нуклеотидные последовательности всех 4 сегментов генома. Эти данные актуализируют необходимость проведения мониторинга за изменением границ распространения флавиподобных вирусов, потенциально опасных для человека, и их переносчиков, что позволит спрогнозировать варианты распространения инфекции.
About the authors
Mikhail Yu. Kartashov
State Research Center of Virology and Biotechnology «Vector»
Author for correspondence.
Email: mikkartash@yandex.ru
ORCID iD: 0000-0002-7857-6822
Ph.D. (Biol.), Senior Researcher
Russian Federation, 630559, Koltsovo, Novosibirsk regionEkaterina I. Krivosheina
State Research Center of Virology and Biotechnology «Vector»
Email: katr962@mail.ru
ORCID iD: 0000-0001-5181-0415
Researcher
Russian Federation, 630559, Koltsovo, Novosibirsk regionValentina Yu. Kurushina
State Research Center of Virology and Biotechnology «Vector»
Email: kurushina.valenti@gmail.com
ORCID iD: 0009-0005-8148-5242
Junior Researcher
Russian Federation, 630559, Koltsovo, Novosibirsk regionAlexander B. Moshkin
Chita anti-plague station
Email: pchs.chita@mail.ru
ORCID iD: 0009-0007-8491-2363
Director
Russian Federation, 672014, ChitaSergey S. Khankhareev
Department of the Federal Service for Supervision of Consumer Rights Protection and Human Welfare of the Republic of Buryatia
Email: org@03.rospotrebnadzor.ru
ORCID iD: 0000-0002-4884-7919
Ph.D. (Med.), Director
Russian Federation, 670013, Ulan-UdeChoigana R. Biche-ool
Center of Hygiene and Epidemiology in the Republic of Tuva
Email: priem@17cgie.ru
ORCID iD: 0009-0002-1622-0553
Head of the Epidemiological Surveillance Department
Russian Federation, 667010, KyzylOksana N. Pelevina
Center for Hygiene and Epidemiology in the Republic of Khakassia
Email: pelevina_on@fbuz19.ru
ORCID iD: 0009-0004-9507-4199
Entomologist, Center for Hygiene and Epidemiology
Russian Federation, 655017, AbakanNikita V. Popov
Center for Hygiene and Epidemiology in the Irkutsk region
Email: ento-molog@mail.ru
ORCID iD: 0009-0002-6276-2388
Head of Department-Entomologist
Russian Federation, 664047, IrkutskOlga L. Bogomazova
Center for Hygiene and Epidemiology in the Irkutsk region
Email: entomolog@sesoirk.irkutsk.ru
ORCID iD: 0009-0009-6892-1125
Entomologist
Russian Federation, 664047, IrkutskVladimir A. Ternovoi
State Research Center of Virology and Biotechnology «Vector»
Email: tern@vector.nsc.ru
ORCID iD: 0000-0003-1275-171X
Ph.D. (Biol.), Head of Department State Research Center of Virology and Biotechnology
Russian Federation, 630559, Koltsovo, Novosibirsk regionReferences
- Pierson T.C., Diamond M.S. The continued threat of emerging flaviviruses. Nat. Microbiol. 2020; 5(6): 796–812. https://doi.org/10.1038/s41564-020-0714-0
- Leonova G.N., Kondratov I.G., Ternovoi V.A., Romanova E.V., Protopopova E.V., Chausov E.V., et al. Characterization of Powassan viruses from Far Eastern Russia. Arch. Virol. 2009; 154(5): 811–20. https://doi.org/10.1007/s00705-009-0376-y
- Růžek D., Yakimenko V.V., Karan L.S., Tkachev S.E. Omsk haemorrhagic fever. Lancet. 2010; 376(9758): 2104–13. https://doi.org/10.1016/S0140-6736(10)61120-8
- Lvov D.K., Al’khovskiy S.V., Zhirnov O.P. 130th anniversary of virology. Voprosy virusologii. 2022; 67(5): 357–84. https://doi.org/10.36233/0507-4088-140 https://elibrary.ru/qhembl
- Garcia-Blanco M.A., Vasudevan S.G., Bradrick S.S., Nicchitta C. Flavivirus RNA transactions from viral entry to genome replication. Antiviral Res. 2016; 134: 244–9. https://doi.org/10.1016/j.antiviral.2016.09.010
- Colmant A.M.G., Charrel R.N., Coutard B. Jingmenviruses: Ubiquitous, understudied, segmented flavi-like viruses. Front. Microbiol. 2022; 13: 997058. https://doi.org/10.3389/fmicb.2022.997058
- Gao X., Zhu K., Wojdyla J.A., Chen P., Qin B., Li Z., et al. Crystal structure of the NS3-like helicase from Alongshan virus. IUCrJ. 2020; 7(Pt. 3): 375–82. https://doi.org/10.1107/S2052252520003632
- Kholodilov I.S., Litov A.G., Klimentov A.S., Belova O.A., Polienko A.E., Nikitin N.A., et al. Isolation and characterisation of Alongshan virus in Russia. Viruses. 2020; 12(4): 362. https://doi.org/10.3390/v12040362
- Kholodilov I.S., Belova O.A., Morozkin E.S., Litov A.G., Ivannikova A.Y., Makenov M.T., et al. Geographical and tick-dependent distribution of flavi-like Alongshan and Yanggou tick viruses in Russia. Viruses. 2021; 13(3): 458. https://doi.org/10.3390/v13030458
- Kholodilov I.S., Belova O.A., Ivannikova A.Y., Gadzhikurbanov M.N., Makenov M.T., Yakovlev A.S., et al. Distribution and characterisation of tick-borne flavi-, flavi-like, and phenuiviruses in the Chelyabinsk region of Russia. Viruses. 2022; 14(12): 2699. https://doi.org/10.3390/v14122699
- Wang Z.D., Wang B., Wei F., Han S.Z., Zhang L., Yang Z.T., et al. A new segmented virus associated with human febrile illness in China. N. Engl. J. Med. 2019; 380(22): 2116–25. https://doi.org/10.1056/NEJMoa1805068
- Kuivanen S., Levanov L., Kareinen L., Sironen T., Jääskeläinen A.J., Plyusnin I., et al. Detection of novel tick-borne pathogen, Alongshan virus, in Ixodes ricinus ticks, south-eastern Finland, 2019. Euro Surveill. 2019; 24(27): 1900394. https://doi.org/10.2807/1560-7917.ES.2019.24.27.1900394
- Temmam S., Bigot T., Chrétien D., Gondard M., Pérot P., Pommelet V., et al. Insights into the host range, genetic diversity, and geographical distribution of Jingmenviruses. mSphere. 2019; 4(6): e00645–19. https://doi.org/10.1128/mSphere.00645-19
- Stanojević M., Li K., Stamenković G., Ilić B., Paunović M., Pešić B., et al. Depicting the RNA virome of hematophagous arthropods from Belgrade, Serbia. Viruses. 2020; 12(9): 975. https://doi.org/10.3390/v12090975
- Ebert C.L., Söder L., Kubinski M., Glanz J., Gregersen E., Dümmer K., et al. Detection and characterization of Alongshan virus in ticks and tick saliva from Lower Saxony, Germany with serological evidence for viral transmission to game and domestic animals. Microorganisms. 2023; 11(3): 543. https://doi.org/10.3390/microorganisms11030543
- Stegmüller S., Fraefel C., Kubacki J. Genome Sequence of Alongshan virus from Ixodes ricinus ticks collected in Switzerland. Microbiol. Resour. Announc. 2023; 12(3): e0128722. https://doi.org/10.1128/mra.01287-22
- Bugmyrin S.V., Romanova L.Y., Belova O.A., Kholodilov I.S., Bespyatova L.A., Chernokhaeva L.L., et al. Pathogens in Ixodes persulcatus and Ixodes ricinus ticks (Acari, Ixodidae) in Karelia (Russia). Ticks Tick Borne Dis. 2022; 13(6): 102045. https://doi.org/10.1016/j.ttbdis.2022.102045
- Wang Z.D., Wang W., Wang N.N., Qiu K., Zhang X., Tana G., et al. Prevalence of the emerging novel Alongshan virus infection in sheep and cattle in Inner Mongolia, northeastern China. Parasit Vectors. 2019; 12(1): 450. https://doi.org/10.1186/s13071-019-3707-1
- Ladner J.T., Wiley M.R., Beitzel B., Auguste A.J., Dupuis A.P. 2nd., Lindquist M.E., et al. A multicomponent animal virus isolated from mosquitoes. Cell Host Microbe. 2016; 20(3): 357–67. https://doi.org/10.1016/j.chom.2016.07.011
- Souza W.M., Fumagalli M.J., Torres Carrasco A.O., Romeiro M.F., Modha S., Seki M.C., et al. Viral diversity of Rhipicephalus microplus parasitizing cattle in southern Brazil. Sci. Rep. 2018; 8(1): 16315. https://doi.org/10.1038/s41598-018-34630-1
- Emmerich P., Jakupi X., von Possel R., Berisha L., Halili B., Günther S., et al. Viral metagenomics, genetic and evolutionary characteristics of Crimean-Congo hemorrhagic fever orthonairovirus in humans, Kosovo. Infect. Genet. Evol. 2018; 65: 6–11. https://doi.org/10.1016/j.meegid.2018.07.010
- Danchinova G.A., Khasnatinov M.A., Zlobin V.I., Kozlova I.V., Verkhozina M.M., Suntsova O.V., et al. Ixodid ticks in Southern part of Eastern Siberia and Mongolia and their spontaneous infectiveness by infectious agents. Byulleten’ sibirskoi meditsiny. 2006; 5(S1): 137–43. https://doi.org/10.20538/1682-0363-2006-137-143 https://elibrary.ru/rtnbop (in Russian)
- Morozkin E.S., Makenov M.T., Zhurenkova O.B., Kholodilov I.S., Belova O.A., Radyuk E.V., et al. Integrated Jingmenvirus polymerase gene in Ixodes ricinus genome. Viruses. 2022; 14(9): 1908. https://doi.org/10.3390/v14091908
- Kartashov M.Yu., Krivosheina E.I., Svirin K.A., Tupota N.L., Ternovoi V.A., Loktev V.B. Genotyping of tick-borne pathogens and determination of human attacking tick species in Novosibirsk and its suburbs. Infektsiya i immunitet. 2022; 12(6): 1103–12. https://doi.org/10.15789/2220-7619-GOT-1979 (in Russian)
- Litov A.G., Okhezin E.V., Kholodilov I.S., Polienko A.E., Karganova G.G. Quantitative polymerase chain reaction system for Alongshan virus detection. Methods Protoc. 2023; 6(5): 79. https://doi.org/10.3390/mps6050079
Supplementary files