Генетическая характеристика изолята аденовируса человека 55-го генотипа (Adenoviridae: Mastadenovirus), выделенного в Москве в 2022 г.
- Авторы: Шеин Д.А.1, Рыжова Н.Н.1, Кунда М.С.1, Ермолова Е.И.1, Ожаровская Т.А.1, Попова О.1, Никитенко Н.А.1, Краснослободцев К.Г.1, Бурцева Е.И.1, Зубкова О.В.1, Воронина О.Л.1, Гинцбург А.Л.1
-
Учреждения:
- ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
- Выпуск: Том 70, № 5 (2025)
- Страницы: 431-443
- Раздел: ОРИГИНАЛЬНЫЕ ИССЛЕДОВАНИЯ
- URL: https://virusjour.crie.ru/jour/article/view/16721
- DOI: https://doi.org/10.36233/0507-4088-297
- EDN: https://elibrary.ru/aatdbs
- ID: 16721
Цитировать
Аннотация
Введение. Аденовирусная инфекция встречается повсеместно в виде спорадических случаев и отдельных вспышек. Аденовирус человека 55-го генотипа (HAdV-55), эндемичный для территорий Китая и Южной Кореи, вызывает острые респираторные вирусные инфекции (ОРВИ) разной степени тяжести как среди гражданского населения, так и в воинских коллективах в разных странах мира. Геномные исследования способствуют достоверной идентификации HAdV-55.
Цель данной работы состояла в идентификации HAdV, выделенного в Москве в 2022 г., проведении полногеномного секвенирования и сравнительного геномного исследования.
Материалы и методы. HAdV-55 выделили из образца пациента, госпитализированного с пневмонией, исследовали методами анализа полиморфизма длины рестрикционных фрагментов и полногеномного секвенирования. Биоинформационный сравнительный анализ выполняли для выборки геномов 83 изолятов.
Результаты. Проведено полногеномное секвенирование изолята HAdV-55, впервые выделенного в РФ. Последовательность генома изолята SCV3008:Ad55 депонировали в GenBank (регистрационный номер PQ641625). Выявлены уникальные мутации в геноме SCV3008:Ad55, одна из которых приводила к консервативной замене T29A в пентоне, не влияющей на его функции. Филогенетический анализ показал кластеризацию SCV3008:Ad55 с изолятами клады II, включившей представителей 7 стран разных континентов, что свидетельствует о широком распространении HAdV-55. Изоляты эндемичных регионов Китая и Южной Кореи формировали отдельные клады. Исследование полиморфизма длин микросателлитов в нетранслируемых областях генома стало дополнительным инструментов различения близкородственных геномов.
Заключение. Сравнительное геномное исследование изолятов HAdV-55, появившегося в результате рекомбинации HAdV-14 и HAdV-11, показало медленное накопление мутаций с 1969 г. как в транслируемых, так и в нетранслируемых областях, позволило выявить уникальные замены нового изолята SVC3008:Ad55. Полученная геномная информация заложила основу для дальнейшего мониторинга HAdV-55 в России и продемонстрировала информативность и значимость полногеномных исследований для наблюдения за аденовирусами. Разработка и внедрение в практику методов генотипирования, нацеленных на выявление HAdV-55 и других клинически значимых генотипов, позволит значительно повысить эффективность диагностики аденовирусных инфекций с угрозой развития бронхопневмонии.
Полный текст
Введение
Аденовирусы (семейство Adenoviridae) – безоболочечные, содержащие двухцепочечную ДНК вирусы, подразделяют на 6 родов: Aviadenovirus, Barthadenovirus, Ichtadenovirus, Mastadenovirus, Siadenovirus и Testadenovirus. Аденовирусы млекопитающих входят в род Mastadenovirus, включающий более 50 видов. Аденовирусы человека (HAdV) относятся к 7 видам: Mastadenovirus adami, M. blackbeardi, M. caesari, M. dominans, M. exoticum, M. faecale и M. russelli1. Различные виды HAdV обладают разным тканевым тропизмом, что зачастую коррелирует с конкретными клиническими симптомами инфекции [1]. HAdV вызывают в основном острые респираторные вирусные инфекции (ОРВИ), но могут также поражать органы зрения, кишечник, мочевыводящие пути и нервную систему. Тяжесть заболевания зависит от типа вируса и иммунного статуса хозяина [1–5]. Наиболее серьезные респираторные инфекции вызывают HAdV 8 генотипов из 10, принадлежащих виду M. blackbeardi: 3, 7, 11, 14, 16, 21, 50, 55 [6].
История выделения HAdV-55 в отдельный генотип показывает роль и развитие методической базы вирусологии, способствующей более точной классификации вирусов. Впервые атипичный вирус HAdV-11 как возбудитель ОРВИ был выявлен с помощью иммунохимических методов в 1974 г. [7]. В 1991 г. сравнение полиморфизма длин фрагментов рестрикции ДНК изолятов позволило выделить генотип HAdV-11a и показать, что вирусы этого генотипа ассоциированы с инфекцией верхних дыхательных путей и бронхопневмонией [8]. В 2009 г. был опубликован первый полный геном HAdV-11a изолята HAdV11-QS (регистрационный номер FJ643676), полученный сборкой перекрывающихся ампликонов, секвенированных по Сэнгеру. Сравнение данных секвенирования способствовало доказательству происхождения HAdV-11a посредством рекомбинации: геном HAdV-11a имеет в основе геном HAdV-14 и часть гена гексона HAdV-11 [9]. В 2011 г. рабочая группа по аденовирусам человека (Human Adenovirus Working Group) рекомендовала использовать сиквенсы полных геномов для типирования и характеристики HAdV и классифицировать рекомбинанты в новые генотипы при отличии нуклеотидной последовательности и биологических свойств [10]. На основании этих рекомендаций в 2013 г. рекомбинант HAdV-11a был назван HAdV-55 с типовым изолятом HAdV11-QS [11]. Ретроспективные исследования коллекционных изолятов показали, что HAdV-55 является эндемичным для Китая и Южной Кореи и доминировал среди вирусов, выделенных при ОРВИ в Пекине с 1965 по 1985 г. [8]. Вспышки HAdV-55-ОРВИ в организованных коллективах фиксируют с 1969 г. среди воинского контингента в Испании [7], в центре профессиональной подготовки в США [12], среди детей в Аргентине, Чили и Уругвае [13], в психиатрических институтах Израиля [14], в семейных коллективах в Китае [15]. HAdV-55 по сравнению с респираторными аденовирусами других генотипов вызывает более тяжелые заболевания и представляет значительную угрозу здоровью населения [6]. В России, по данным Центра экологии и эпидемиологии гриппа Института вирусологии им. Д.И. Ивановского ФГБУ «НИЦЭМ им. Н.Ф. Гамалеи» Минздрава России в сотрудничестве с 10 опорными базами, в эпидемическом сезоне 2021–2022 гг. на исследованных территориях частота положительных проб на аденовирусы составила 7,7% (1793 образца) из числа тестированных на ОРВИ [16]. HAdV, выделенный из одного из 1793 образцов, стал объектом подробного исследования.
Цель работы – идентификация HAdV, выделенного в Москве в 2022 г., проведение полногеномного секвенирования и сравнительного геномного исследования.
Материалы и методы
Материалы. Бронхоальвеолярный лаваж (БАЛ) от госпитализированного мужчины 34 лет с диагнозом «пневмония неуточненная».
Исследование проводили при добровольном информированном согласии пациента. Протокол исследования одобрен Этическим комитетом ГБУЗ г. Москвы «Инфекционная клиническая больница № 1 Департамента здравоохранения города Москвы» (Протокол № 8 от 28.12.2022)
Методы. Идентификацию вирусов проводили посредством экстракции РНК/ДНК из клинического материала с помощью набора «РИБО-ПРЕП» («Интерлабсервис», Россия) с последующей детекцией РНК/ДНК возбудителей респираторных инфекций в полимеразной цепной реакции с обратной транскрипцией в реальном времени (ПЦР-РВ) с использованием коммерческих тест-систем «АмплиСенс ОРВИ-скрин-FL» («Интерлабсервис», Россия) согласно инструкции производителя на детектирующем амплификаторе Bio-Rad CFX-96 (Bio-Rad, США).
Выделение HAdV. Вирус накапливали в клетках НЕК293 (эмбриональной почки человека): 100 мкл БАЛ заражали клетки (0,5 × 106 клеток/3 см2, инкубировали в стандартных условиях (+37 °C, 5% CO2) до наступления цитопатического действия (ЦПД). Для препаративного наращивания вируса использовали культуральные чашки диаметром 15 см. Инфицированные клетки после наступления 100% ЦПД собирали, концентрировали низкоскоростным центрифугированием (2000 об/10 мин), ресуспендировали в буфере (0,01 М трис-HCl pH 8,0, 0,01 M NaCl, 5 мМ ЭДТА), подвергали трем циклам замораживания–оттаивания и центрифугировали при 5000 об/10 мин, осадок удаляли. Аденовирус из супернатанта очищали методом ультрацентрифугирования в градиенте плотности хлористого цезия (в ступенчатом (CsСl с показателем преломления 1,355, 1,365 и 1,375) и равновесном градиенте (CsСl с показателем преломления 1,365).
Анализ полиморфизма длин рестрикционных фрагментов (ПДРФ). Геномную ДНК очищенного вируса выделяли с использованием набора Wizard Genomic DNA Purification Kit (Promega, США). ДНК (1 мкг) гидролизовали рестриктазами Cfr41I, XagI и XhoI (Thermo, США) и анализировали методом электрофореза в агарозном геле, используя более длительную выдержку для детектирования низкомолекулярных фрагментов. Анализ ПДРФ in silico проводили с помощью программы Geneious Prime (Biomatters, Новая Зеландия).
Секвенирование и сборка генома. Приготовление библиотеки выполняли с применением набора KAPA HyperPlus Kit (F. Hoffmann-La Roche Ltd., Швейцария) согласно протоколам производителя, проверку качества и размера библиотек проводили с помощью электрофореза на чипах High Sensitivity DNA Chips 2100 Bioanalyzer System (Agilent, США), секвенирование – на приборе NextSeq 500/550 (Illumina, США), используя картриджи Mid Output 300 cycles. Для сборки прочтений de novo и по референсным последовательностям использовали пакет программ CLC Genomic Workbench v. 21 (Qiagen, США). Для уточнения последовательностей гомополимеров применяли секвенирование по Сэнгеру, используя наборы BDT UltraSeq HP Kit (SenseCare Bio, Китай), электрофорез проводили в капиллярах 50 см, в геле POP-7 на приборе 3500 Genetic Analyzer (Applied Biosystems, США).
Сравнительный анализ. В сравнительный анализ включили 83 полных генома HAdV-55 (табл. 1) и геном HAdV-14 (MF062484). Выравнивание геномных последовательностей выборки изолятов, построение филогенетического древа Neighbor-joining, расчет ANI (average nucleotide identity, средней нуклеотидной идентичности) выполняли с помощью модуля Whole Genome Alignment пакета программ CLC Genomic Workbench v. 21 (Qiagen, США). Для визуализации древа использовали программу MEGA11 [17]. Трансляцию рамок считывания и выравнивание аминокислотных последовательностей выполняли в программе MEGA11 [17].
Таблица 1. Штаммы HAdV-55, используемые для геномного анализа с целью изучения распространения вируса, региональной персистенции и генетической изменчивости
Table 1. HAdV-55 strains used for genomic analysis in order to study the spread of the virus, regional persistence and genetic variability
Номер в базе NCBI GenBank Accession Number | Место выделения Country | Год выделения Year of isolation | Номер в базе NCBI GenBank Accession Number | Место выделения Country | Год выделения Year of isolation |
MN654381.1 | Египет / Egypt | 2000 | PP002035.1 | Китай / China | 2018 |
MN654383.1 | Египет / Egypt | 2000 | PP002036.1 | Китай / China | 2018 |
MN654385.1 | Египет / Egypt | 2000 | PP002037.1 | Китай / China | 2018 |
MN654380.1 | Египет / Egypt | 2000 | PP002043.1 | Китай / China | 2018 |
MN654382.1 | Египет / Egypt | 2002 | PP002044.1 | Китай / China | 2018 |
MN654390.1 | Египет / Egypt | 2005 | PP002045.1 | Китай / China | 2018 |
MN654391.1 | Египет / Egypt | 2005 | PP002046.1 | Китай / China | 2018 |
MN654386.1 | Египет / Egypt | 2007 | MH256653.1 | Китай / China | 2018 |
MN654384.1 | Египет / Egypt | 2008 | MH256655.1 | Китай / China | 2018 |
MN654387.1 | Египет / Egypt | 2009 | MH256657.1 | Китай / China | 2018 |
MG905110.1 | Испания / Spain | 1969 | MH256654.1 | Китай / China | 2018 |
FJ643676.1 | Китай / China | 2006 | MH256656.1 | Китай / China | 2018 |
JX123027.1 | Китай / China | 2010 | PP002040.1 | Китай / China | 2018 |
JX491639.1 | Китай / China | 2011 | MT806174.1 | Китай / China | 2019 |
JX123028.1 | Китай / China | 2011 | MT806175.1 | Китай / China | 2019 |
MK123979.1 | Китай / China | 2011 | MT806170.1 | Китай / China | 2019 |
KJ883522.1 | Китай / China | 2011 | MT806172.1 | Китай / China | 2019 |
KP279748.1 | Китай / China | 2012 | MT806173.1 | Китай / China | 2019 |
KP896478.1 | Китай / China | 2012 | MT806171.1 | Китай / China | 2019 |
JX123029.1 | Китай / China | 2012 | OM714808.1 | Китай / China | 2020 |
KC857701.1 | Китай / China | 2012 | OP375144.1 | Китай / China | 2021 |
KP896483.1 | Китай / China | 2013 | MN654388.1 | Сингапур / Singapore | 2005 |
KJ883520.1 | Китай / China | 2013 | MN654389.1 | Сингапур / Singapore | 2005 |
KJ883521.1 | Китай / China | 2013 | MN654394.1 | США / USA | 1976 |
KP896484.1 | Китай / China | 2013 | MN654392.1 | США / USA | 1997 |
MK123980.1 | Китай / China | 2013 | MT513753.1 | США / USA | 2006 |
MK123981.1 | Китай / China | 2013 | MN654395.1 | США / USA | 2020 |
KF908851.1 | Китай / China | 2013 | MN654375.1 | Южная Корея / South Korea | 2009 |
MK886831.1 | Китай / China | 2015 | MN654376.1 | Южная Корея / South Korea | 2009 |
KX289874.1 | Китай / China | 2015 | MN654377.1 | Южная Корея / South Korea | 2009 |
KY070248.1 | Китай / China | 2016 | MN654378.1 | Южная Корея / South Korea | 2009 |
KY780931.1 | Китай / China | 2016 | MN654379.1 | Южная Корея / South Korea | 2009 |
KY780932.1 | Китай / China | 2016 | KX494979.1 | Южная Корея / South Korea | 2016 |
KY780933.1 | Китай / China | 2016 | KY471318.1 | Южная Корея / South Korea | 2017 |
PP002039.1 | Китай / China | 2018 | KY471322.1 | Южная Корея / South Korea | 2017 |
PP002041.1 | Китай / China | 2018 | KY471319.1 | Южная Корея / South Korea | 2017 |
MN052861.1 | Китай / China | 2018 | KY471320.1 | Южная Корея / South Korea | 2017 |
MK123978.1 | Китай / China | 2018 | KY471321.1 | Южная Корея / South Korea | 2017 |
PP002033.1 | Китай / China | 2018 | KY471323.1 | Южная Корея / South Korea | 2017 |
PP002034.1 | Китай / China | 2018 | MW053454.1 | Южная Корея / South Korea | 2019 |
PP002038.1 | Китай / China | 2018 | MN654393.1 | Япония / Japan | 2012 |
PP002032.1 | Китай / China | 2018 |
Результаты
В выборке респираторных образцов, поступивших из стационаров Москвы в сезоне 2021–2022 гг., 12 содержали HAdV по данным ПЦР-РВ. Образцы проанализировали по значениям вирусной нагрузки и множественности инфекции. Один из образцов (БАЛ пациента, госпитализированного с пневмонией), отличавшийся высоким показателем нагрузки ДНК HAdV (значение порогового цикла, Ct = 12,3) и отсутствием сопутствующей инфекции другими респираторными вирусами, использовали для изоляции HAdV.
Выделенный аденовирус идентифицировали как HAdV-55 на основании данных секвенирования, депонировали в Государственной коллекции вирусов Института вирусологии им. Д.И. Ивановского ФГБУ «НИЦЭМ им. Н.Ф. Гамалеи» Минздрава России под номером SCV3008:Ad55, геномные данные зарегистрировали в GenBank (регистрационный номер PQ641625).
Анализ ПДРФ ДНК изолята выявил следующие фрагменты: Cfr41I (12894, 8674, 7768, 2664, 1194, 1181 и 403 п.н.), XhoI (10335, 8005, 6544, 5761, 2628, 1355 и 150 п.н.) и XagI (20615, 5219, 3789, 2944, 1359 и 852 п.н.). На рис. 1 (а) представлены наиболее яркие из них. Сопоставление с данными in silico для изолятов HAdV-55 (MG905110), HAdV-11 (AY163756) и HAdV-14 (MF062484) (рис. 1 б) подтверждает сходство рестрикционных фрагментов изолятов SCV3008:Ad55 и HAdV-55 (MG905110) и подчеркивает, что в рекомбинанте HAdV-55 основу генома составляют гены HAdV-14.
Рис. 1. Полиморфизм длин рестрикционных фрагментов, полученных с рестриктазами Cfr41I, XagI и XhoI. a ‒ для ДНК изолята SCV3008:Ad55 in vitro; б ‒ для ДНК штаммов HAd-55, HAd-11 и HAd-14 in silico.
Fig. 1. Restriction fragment length polymorphisms (RFLPs) using Cfr41I, XagI, and XhoI restriction enzymes. a – DNA of the SCV3008 isolate in vitro; b – DNA of the HAd-55, HAd-11 and HAd-14 strains in silico.
Сходство с HAdV-14 показал и расчет ANI между геномами HAdV-55 выборки, представленными в табл. 1, SCV3008:Ad55 и HAdV-14 (MF062484). Значение ANI между HAdV-55 и HAdV-14 составило 98,7478–98,9411, а между геномами HAdV-55 – 99,6546–100,000.
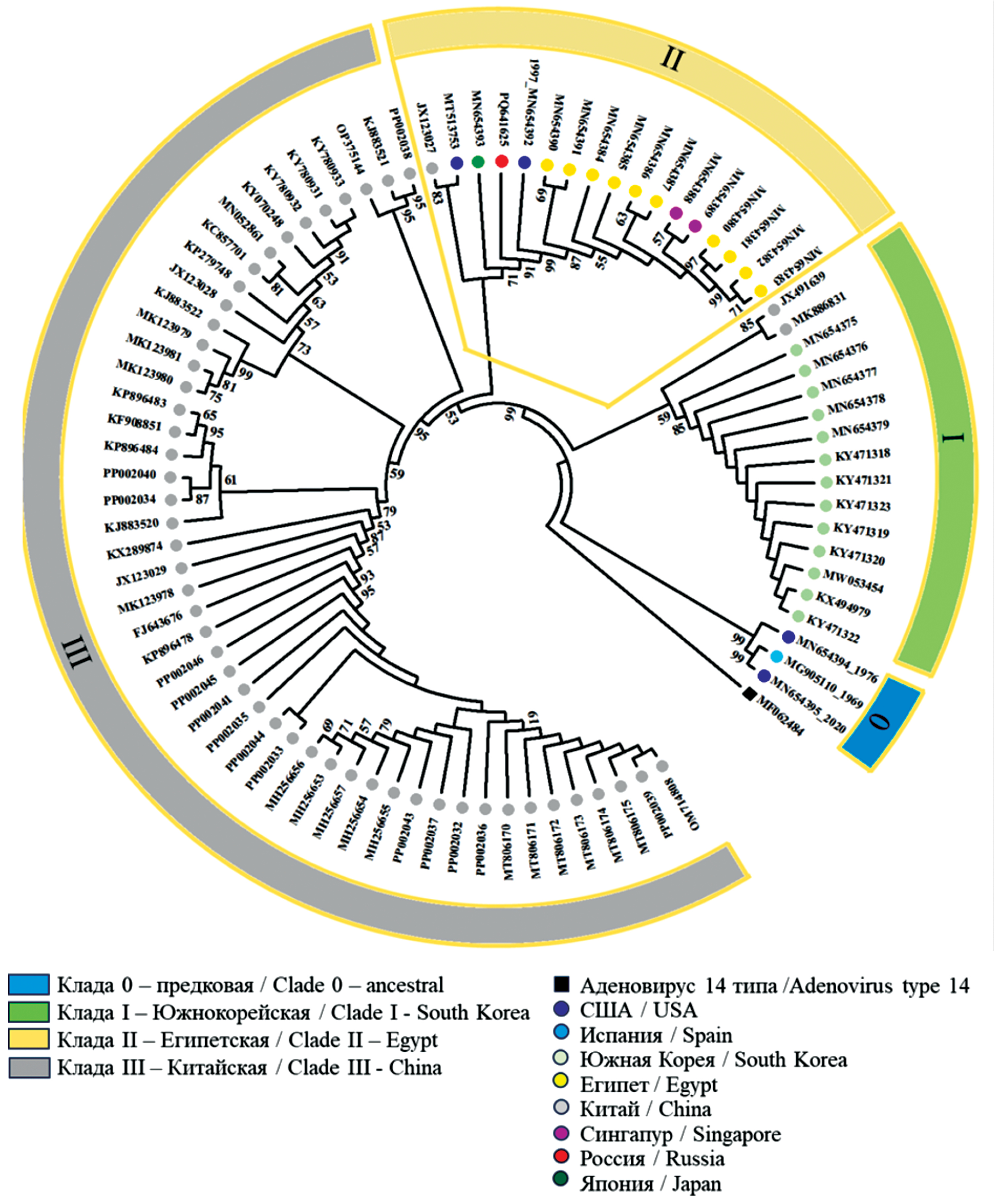
В филогенетический анализ включили изоляты HAdV-55, выделенные в 8 странах в 1969–2022 гг. (табл. 1). Древо гомологии представлено на рис. 2. Как видно из рисунка, геномы сформировали 4 клады. В базовую кладу «0» вошли наиболее ранние изоляты MG905110 (Испания, 1969), MN654394 (США, 1976) и изолят MN654395 (США, 2020). Кладу «I» образовали 13 изолятов из Южной Кореи и два изолята из Китая (2011 и 2015 гг.). Клада «II», основу которой составили изоляты из Египта (12 изолятов), была наиболее разнообразна по представленности стран. В нее вошли изоляты из Сингапура (MN654388 и MN654389, 2005), Японии (MN654393, 2012), США (MN654392, 1997; MT513753, 2006), Китая (JX123027, 2010) и выделенный нами изолят SCV3008:Ad55. Самую многочисленную кладу «III» образовали изоляты Китая 2006–2021 гг.
Рис. 2. Филогенетическое древо Neighbor-joining, построенное на основе полных геномов 83 изолятов HAd-55, представленных в табл. 1, и генома изолята SCV3008:Ad55 (PQ641625). Клады 0–III охарактеризованы в легенде. MF062484 – изолят HAd-14, представляет внешнюю группу.
Fig. 2. Neighbor-joining phylogenetic tree constructed based on the complete genomes of 83 HAd-55 isolates presented in Table 1, and the genome of the SCV3008:Ad55 isolate (PQ641625). Clades 0–III are characterized in the legend. MF062484 – HAd-14 isolate, represents an outgroup.
В кладе «II» максимально близким изоляту SCV3008:Ad55 по значению ANI (99,9396) был изолят из Японии (MN654393). Отличия между геномами SCV3008:Ad55 и MN654393 выявили как в нетранслируемых областях генома, так и в генах структурных и неструктурных белков (табл. 2). Выявленные замены сопоставили с последовательностями других геномов выборки. Отметим, что сравнение областей ITR и ближайших к ним было возможно не для всех геномов. Из выявленных замен 4 были характерны для большинства изолятов клады «II», 4 являлись уникальными для генома японского изолята, 12 замен отличали изолят SCV3008:Ad55.
Таблица 2. Характеристика замен в геноме изолята SVC308:Ad55
Table 2. Characterization of substitutions in the genome of isolate SVC308-Ad55
Название области генома/CDS Name of the region of the genome/CDS | Замена в геноме SVC308:Ad55 относительно генома WPAFB415 Substitution in the SVC308:Ad55 genome relative to the WPAFB415 | Встречаемость мутации Mutation frequency | Замены в последовательности белка Substitutions in amino acid sequence |
Нетранслируемые области / Untranslated regions | C134T** | Clade Egypt* | |
A445G | SVC308:Ad55 | ||
T3437C | SVC308:Ad55 | ||
A3924G | SVC308:Ad55 | ||
G34619T*** | SVC308:Ad55 | ||
pIX 14,2 кДа / 14.2 kDa | C3536T | SVC308:Ad55 | |
pIVa2 50,9 кДа / 50.9 kDa | T4656C | WPAFB415 | |
128,9 кДа ДНК-полимераза / 128.9 kDa DNA polymerase | C6707T | SVC308:Ad55 | |
T6764A | WPAFB415 | Lys→Asp | |
pTP 73,4 кДа / 73.4 kDa | G8779A | SVC308:Ad55 | |
C8815T | Clade Egypt* | ||
L1 52/55K 43,9 кДа / 43,9 kDa | C11734T | SVC308:Ad55 | |
L1 pIIIa 65,6 кДа / 65.6 kDa | G13349A | Clade Egypt* | |
L2 пентон 62,5 кДа / 62,5 kDa penton protein | T13787A | SVC308:Ad55 | Thr→Ala |
G14203A | SVC308:Ad55 | ||
L2 pV 40,1 кДа / 40.1 kDa | T16315C | WPAFB415 | Glu→Leu |
G16530C | WPAFB415 | ||
E2A 58,3 кДа ДНК-связывающий белок / 58.3 kDa DNA-binding protein | G22776A | SVC308:Ad55 | |
L4 22K 21,6 кДа / 21.6 kDa | C26083T | Clade Egypt* | His→Tyr |
L4 pVIII 25 кДа / 25 kDa | A26918G | WPAFB415 | |
E3 18,5 kDa / 18.5 kDa | T28096G | SVC308:Ad55 |
Примечание. * – кроме MT513753, MN654393, JX123027; ** – область левого ITR есть только в 65 геномах из 85; *** – область генома перед правым ITR присутствует только в 75 геномах из 85.
Note. * – except for MT513753, MN654393, JX123027; ** – the left ITR region is found in only 65 of the 85 genomes; *** – the region of the genome in front of the right ITR is present in only 75 out of 85 genomes.
Замены в 4 рамках считывания были несинонимичными. Мутация в пептоне (Thr29Ala) была уникальна для изолята SCV3008:Ad55. Замены в ДНК-полимеразе (Asp566Lys) и в белке pV (Leu105Glu) отличали японский изолят MN654393. Мутация в белке L4 22К (His162Tyr) встречалась у 14 изолятов клады «II», включая SCV3008:Ad55 (табл. 2).
При анализе геномов мы обратили внимание на неоднородность размеров поли-А/поли-Т-последовательностей в межгенных областях (табл. 3). Размеры областей 2–6, отмеченные в геноме SCV3008:Ad55, были характерны для многих геномов выборки HAdV-55. Область 1 с заменой A6G являлась уникальной для SCV3008:Ad55. Последовательности гомополимеров в геномах аденовирусов, называемые также микросателлитами, обратили на себя внимание исследователей при расследовании вспышек аденовирусной инфекции со смертельными исходами в воинских коллективах США в 2006–2007 гг. Полиморфизм длин локусов микросателлитов стал маркером высокого разрешения для отнесения HAdV-14 к одной вспышке [18]. Мы провели сравнение локусов микросателлитов у изолятов клады II «Egypt», включившей 17 изолятов разных континентов. Из данных табл. 4 следует, что большинство изолятов клады было сходно по размеру микросателлитов всех 6 локусов. Максимальное количество локусов (4) отличало геномы изолятов из Японии (MN654393) и России (SCV3008:Ad55), по 3 локусам отличался геном единственного изолята из Китая в кладе II (JX123027), по двум разным локусам – изоляты из США (MN654392, 1997; MT513753, 2006), по одному – изоляты из Сингапура (MN654388 и MN654389). Из 10 египетских изолятов в кладе II три имели по одному локусу отличий. Таким образом, при высоком консерватизме геномов HAdV-55 локусы микросателлитов действительно позволяют различить геномы вирусов в пределах одной клады.
Таблица 3. Области повторов в геномах выборки аденовирусов человека 55-го типа
Table 3. Regions of repeats in the genomes of a sample of adenoviruses belonging to genotype 55
N | Положение по геному PQ641625 / Соседние ОРС The position according to PQ641625 genome / Neighboring ORS | Изолят Isolate | Число нуклеотидов в повторе The number of nucleotides in the repeat | Количество изолятов Number of isolates |
1 | 3918–3933 bp / pIX; pIVa2 | MF062484/China/2010* | 1 | |
OP375144/China/2021 | 3 | |||
MW053454/China/2023 | 1 | |||
PQ641625/Russia/2022 | 1 | |||
MN654393/Japan/2012 | 1 | |||
MN654378/S.Korea/2009 | 24 | |||
MN654392/USA/1997 | 5 | |||
MN654394/USA/1976 | 1 | |||
FJ643676/China/2011 | 48 | |||
2 | 10651–10664 bp / pTP; L1 | MF062484/China/2010* | ||
MN654395/USA/2020 | 1 | |||
MW053454/China/2023 | 3 | |||
KY471322/S.Korea/2017 | 9 | |||
PP002032/China/2018 | 21 | |||
PQ641625/Russia/2022 | 7 | |||
MN654394/USA/1976 | 37 | |||
MK123980/China/2013 | 6 | |||
KP896484/China/2013 | 1 | |||
3 | 13620–13630 bp/L1 pIIIa; L2 penton [polyA_signal_sequence (aaataaa) 13627–13633 bp] | MF062484/China/2010* | 1 | |
PP002034/China/2018 | 1 | |||
MN654388/Singapore/2005 | 16 | |||
PQ641625/Russia/2022 | 66 | |||
PP002040/China/2018 | 1 | |||
4a | 17323–17334 bp / L2 pX; L3 pVI | MF062484/China/2010* | ||
MK123978/China/2018 | 1 | |||
PQ641625/Russia/2022 | 51 | |||
KY070248/China/2016 | 20 | |||
MN654394/USA/1976 | 12 | |||
KP896484/China/2013 | 1 | |||
4b | 17341–17352 bp / L2 pX; L3 pVI [polyA_signal_sequence (aataaa) 17339–17344 bp] | MF062484/China/2010* | ||
KX494979/S.Korea/2016 | 5 | |||
PQ641625/Russia/2022 | 75 | |||
KH289874/China/2015 | 5 | |||
5 | 29474–29486 bp / E3 20.2 kDa; E3 10.3 kDa | MF062484/China/2010* | ||
MT513753/USA/2006 | 1 | |||
MK123981/China/2013 | 3 | |||
OM714808/China/2020 | 1 | |||
PQ641625/Russia/2022 | 17 | |||
KP896483/China/2013 | 37 | |||
JX123029/China/2012 | 25 | |||
KP896484/China/2013 | 1 | |||
6 | 34006 – 34016 bp / E4 ORF2; E4 ORF1 | MF062484/China/2010* | 1 | |
MT513753/USA/2006 | 3 | |||
PQ641625/Russia/2022 | 28 | |||
JX123029/China/2012 | 53 |
Примечание. * – предковый геном Human adenovirus 14.
Note. * – the ancestral genome of Human adenovirus 14.
Таблица 4. Размер локусов микросателлитов в геномах изолятов клады II «Egypt»
Table 4. Size of microsatellite loci in the genomes of clade II «Egypt» isolates
Локус Locus | Изолят Isolate | Размер гомополимера (нт) Homopolymer size (nt) | Локус Locus | Изолят Isolate | Размер гомополимера (нт) Homopolymer size (nt) |
1 | Большинство* / Most | A (13) | 4a, 4b | Большинство / Most | A (10); A (10) |
MN654380 (Egypt 2000) | A (12) | JX123027 (China 2010) | A (10); A (11) | ||
MN654392 (USA 1997) | A (12) | MN654392 (USA 1997) | A (9); A (10) | ||
MN654393 (Japan 2012) | A (14) | 5 | Большинство / Most | T (9) | |
PQ641625 (Russia 2022) | A (13) G (1) | MN654393 (Japan 2012) | T (11) | ||
2 | Большинство / Most | T (11) | PQ641625 (Russia 2022) | T (11) | |
MN654393 (Japan 2012) | T (12) | JX123027 (China 2010) | T (10) | ||
PQ641625 (Russia 2022) | T (12) | MT513753 (USA 2006) | T (14) | ||
JX123027 (China 2010) | T (12) | 6 | Большинство / Most | A (8) | |
3 | Большинство / Most | A (10) | MN654393 (Japan 2012) | A (10) | |
MN654386 (Egypt 2007) | A (11) | PQ641625 (Russia 2022) | A (10) | ||
MN654387 (Egypt 2009) | A (11) | MT513753 (USA 2006) | A (11) | ||
MN654388 (Singapore 2005) | A (11) | ||||
MN654389 (Singapore 2005) | A (11) |
Примечание. * ‒ в кладе II «Egypt» 17 изолятов.
Note. * ‒ Clade II «Egypt» contains 17 isolates.
Обсуждение
В представленной работе впервые описан геном изолята HAdV-55 SCV3008:Ad55, выделенного на территории РФ. Отметим, что молекулярно-эпидемиологические геномные исследования аденовирусов в РФ, выполненные ранее, являются единичными и посвящены изучению M. caesari HAdV, возбудителей респираторных инфекций у детей [19]. Сбор сравнительной информации для геномных исследований затруднен в связи с низкой степенью внедрения методов генотипирования в лабораторную диагностику аденовирусной инфекции.
В России молекулярно-генетический подход, одобренный с 2010 г., применяют для эпидемиологического мониторинга аденовирусной инфекции и идентификации возбудителя до семейства Adenoviridae в Референс-центре по диагностике гриппа и ОРВИ на базе НИИ гриппа им. А.А. Смородинцева, в Центре экологии и эпидемиологии гриппа НИЦЭМ им. Н.Ф. Гамалеи и в опорных базах Роспотребнадзора. На особом контроле находятся аденовирусные инфекции у детей, которые также подлежат молекулярной диагностике согласно клиническим рекомендациям2. Однако генотипирование HAdV не входит в перечень методов лабораторной диагностики.
ECDC (European Centre for Disease Prevention and Control) не проводит планового наблюдения аденовирусной инфекции и фиксирует только вспышки заболевания, тогда как CDC (Centers for Disease Control and Prevention, США) разработал рекомендации по идентификации HAdV на основе амплификации нуклеиновых кислот и создал Национальную систему отчетности по типам аденовирусов (The National Adenovirus Type Reporting System, NATRS). По сведениям NATRS, в 2017–2023 гг. HAdV 6 генотипов являлись наиболее распространенными в США, среди которых HAdV-7 и HAdV-14 вида M. blackbeardi составили 13,4 и 7,8% соответственно (https://www.cdc.gov/adenovirus/hcp/outbreaks/index.html). Японская национальная система эпидемиологического надзора за инфекционными заболеваниями также проводит генотипирование аденовирусов, отмечая среди преобладающих M. blackbeardi HAdV-3, 7, 11, 34, 35, а среди минорных 14, 16, 55, 66, 68, 79 [20]. CDC Китая контролирует грипп и ОРВИ, но не публикует в открытой печати отчеты по генотипированию вирусов [21]. Таким образом, из национальных систем контроля учет HAdV-55 проводит только система эпиднадзора Японии.
Анализ научных публикаций за 2012–2025 гг., размещенных в PubMed, показал, что из 48 статей, упоминающих в ключевых словах HAdV-55, 39 (81%) опубликовано исследователями из Китая, 7 – из Южной Кореи, по одной – из США и Сенегала. Такое соотношение публикаций подтверждает эндемичность HAdV-55 для Китая и Южной Кореи. Следует отметить, что из публикаций Южной Кореи только в двух представлено исследование инфекций HAdV-55 среди гражданского населения, в остальных описаны вспышки ОРВИ, вызванной HAdV-55, среди воинского контингента [22]. Тему HAdV-55-инфекции среди военных продолжает публикация из США [23], посвященная анализу изолята вируса MW053454, выделенного от американского военнослужащего, находившегося в Южной Корее в 2019 г. Изолят MW053454 отличался от южнокорейского изолята KX494979 2016 г. одной синонимичной заменой. В нашем исследовании оба изолята вошли в кладу I «South Korea». В Сенегале с 2012 по 2015 г. у пациентов с ОРВИ M. blackbeardi HAdV выявили в 9 случаях, среди которых отмечены HAdV-7, HAdV-55 и HAdV-11 [24]. Приведенные данные свидетельствуют о том, что генотипирование HAdV постепенно входит в лабораторную практику.
Учитывая вышесказанное, для проведения сравнительного исследования генома изолята SCV3008:Ad55 мы воспользовались данными GenBank, собрав выборку из 83 изолятов 1969–2022 гг. из 7 стран. Анализ выборки показал высокое сходство геномов HAdV-55, достигающее по показателю ANI 99,7–100%, что согласуется с данными других исследований, выполненных на меньшем количестве изолятов [25]. Вместе с тем филогенетический анализ позволил разделить геномы выборки на клады, что свидетельствует о наличии гетерогенности даже при высокой гомологии. Клады I и III соответствовали географической принадлежности изолятов и показали эпидемическую связь как между изолятами из Китая, так и изолятами из Южной Кореи. Изоляты из Египта 2000–2009 гг., страны, удаленной от эндемичных по HAdV-55 территорий, кластеризовались с изолятами из 5 государств, в том числе Китая, что свидетельствует о распространении HAdV-55, чему способствуют процессы глобализации. Следует отметить, что в кладу II вошли преимущественно изоляты от гражданского населения. Исключение составили изоляты из Сингапура и Японии, полученные из образцов военных, заболевших ОРВИ [25].
Сравнительный геномный анализ выявил отличия изолята SVC3008:Ad55, а именно 12 точковых мутаций, распределенных по всему геному, из которых несинонимичной была замена в рамке считывания пентона L2 62,5 кДа, приводящая к замене T29A в N-конце белковой последовательности. Поскольку замена консервативна, она не сказывается на амфипатических свойствах N-концевой спирали белка и на способности мотива PPRY (42–45 а.о.) взаимодействовать с доменами WW клеточных убиквитинлигаз, что обеспечивает проникновение вируса в эукариотическую клетку, определяющее его инфекционность [26].
Дополнительную информацию о разнообразии близкородственных геномов изолятов клады II дал анализ полиморфизма длин микросателлитов (гомополимеров) 6 локусов в нетранслируемых областях. Из 17 изолятов клады 10 имели отличие хотя бы по одному локусу. Изоляты из Японии и России отличались от других изолятов клады по 4 локусам микросателлитов. Такой подход позволил различить даже изоляты из Египта внутри двух регионов: Александрии (2000–2002) и Каира (2005–2009).
Заключение
Сравнительное геномное исследование изолятов HAdV-55, появившегося в результате рекомбинации HAdV-14 и HAdV-11, показало медленное накопление мутаций с 1969 г. как в транслируемых, так и в нетранслируемых областях, позволило выявить уникальные замены нового изолята SVC3008:Ad55. Полученная геномная информация заложила основу для разработки диагностических наборов и дальнейшего мониторинга HAdV-55, вызывающего инфекции, осложненные бронхопневмонией. В то же время, поскольку аденовирусы подвержены рекомбинационной изменчивости, а горячие точки рекомбинации множественны (гены пентона, гексона, филамента (фибера), E1, E3 и E4) [27], именно полногеномное секвенирование эффективно в мониторинге и молекулярно-эпидемиологическом анализе возбудителей аденовирусной инфекции.
1 ICTV. Family: Adenoviridae. Доступно по: https://ictv.global/report/chapter/adenoviridae/adenoviridae
2 Клинические рекомендации (протокол лечения) оказания медицинской помощи детям больным аденовирусной инфекцией; 2013. Доступно по: http://niidi.ru/dotAsset/69f7f879-9765-4634-a621-8792acf587b7.pdf
Об авторах
Даниил Алексеевич Шеин
ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
Email: daniil.schein@yandex.ru
ORCID iD: 0009-0003-3768-9817
аспирант лаборатории иммунобиотехнологии
Россия, 123098, МоскваНаталья Николаевна Рыжова
ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
Email: rynatalia@yandex.ru
ORCID iD: 0000-0001-5361-870X
канд. биол. наук, старший научный сотрудник лаборатории анализа геномов
Россия, 123098, МоскваМарина Сергеевна Кунда
ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
Email: markunda99@gmail.com
ORCID iD: 0000-0003-1945-0397
канд. биол. наук, старший научный сотрудник лаборатории анализа геномов
Россия, 123098, МоскваЕкатерина Ивановна Ермолова
ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
Email: aksenova16@yandex.ru
ORCID iD: 0000-0002-0437-9404
канд. биол. наук, старший научный сотрудник лаборатории анализа геномов
Россия, 123098, МоскваТатьяна Андреевна Ожаровская
ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
Email: t.ozh@yandex.ru
ORCID iD: 0000-0001-7147-1553
канд. биол. наук, старший научный сотрудник лаборатории иммунобиотехнологии
Россия, 123098, МоскваОльга Попова
ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
Email: olga.popova31@yandex.ru
ORCID iD: 0000-0003-3248-1227
младший научный сотрудник лаборатории иммунобиотехнологии
Россия, 123098, МоскваНаталья Анатольевна Никитенко
ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
Email: nan-nikitenko@yandex.ru
ORCID iD: 0000-0001-5829-744X
канд. биол. наук, старший научный сотрудник лаборатории клеточной микробиологии, заведующая медицинским отделом
Россия, 123098, МоскваКирилл Геннадьевич Краснослободцев
ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
Email: kkg_87@mail.ru
ORCID iD: 0000-0003-1745-9128
научный сотрудник этиологии и эпидемиологии гриппа
Россия, 123098, МоскваЕлена Ивановна Бурцева
ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
Email: elena-burtseva@yandex.ru
ORCID iD: 0000-0003-2518-6801
д-р мед. наук, заведующая лабораторией этиологии и эпидемиологии гриппа
Россия, 123098, МоскваОльга Вадимовна Зубкова
ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
Автор, ответственный за переписку.
Email: olga-zubkova@yandex.ru
ORCID iD: 0000-0001-7893-8419
канд. биол. наук, ведущий научный сотрудник лаборатории иммунобиотехнологии
Россия, 123098, МоскваОльга Львовна Воронина
ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
Email: olv550@gmail.com
ORCID iD: 0000-0001-7206-3594
канд. биол. наук, доцент, ведущий научный сотрудник – заведующая лабораторией анализа геномов
Россия, 123098, МоскваАлександр Леонидович Гинцбург
ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
Email: gintsburg@gamaleya.org
ORCID iD: 0000-0003-1769-5059
д-р биол. наук, академик РАН, профессор, директор
Россия, 123098, МоскваСписок литературы
- Lynch J.P. 3rd, Kajon A.E. Adenovirus: epidemiology, global spread of novel serotypes, and advances in treatment and prevention. Semin. Respir. Crit. Care Med. 2016; 37(4): 586–602. https://doi.org/10.1055/s-0036-1584923
- Coleman K.K., Wong C.C., Jayakumar J., Nguyen T.T., Wong A.W.L., Yadana S., et al. Adenoviral infections in Singapore: Should new antiviral therapies and vaccines be adopted? J. Infect. Dis. 2020; 221(4): 566–77. https://doi.org/10.1093/infdis/jiz489
- Xu W., Xu Z., Huang L., Qin E.Q., Zhang J.L., Zhao P., et al. Transcriptome sequencing identifies novel immune response genes highly related to the severity of human adenovirus type 55 infection. Front. Microbiol. 2019; 10: 130. https://doi.org/10.3389/fmicb.2019.00130
- Kajon A.E., Lamson D.M., St. George K. Emergence and re-emergence of respiratory adenoviruses in the United States. Curr. Opin. Virol. 2019; 34: 63–9. https://doi.org/10.1016/j.coviro.2018.12.004
- Dhingra A., Hage E., Ganzenmueller T., Böttcher S., Hofmann J., Hamprecht K., et al. Molecular Evolution of Human Adenovirus (HAdV) Species C. Sci Rep. 2019; 9(1): 1039. https://doi.org/10.1038/s41598-018-37249-4
- Scott M.K., Chommanard C., Lu X., Appelgate D., Grenz L., Schneider E., et al. Human adenovirus associated with severe respiratory infection, Oregon, USA, 2013–2014. Emerg. Infect. Dis. 2016; 22(6): 1044–51. https://doi.org/10.3201/eid2206.151898
- Hierholzer J.C., Pumarola A., Rodriguez-Torres A., Beltran M. Occurrence of respiratory illness due to an atypical strain of adenovirus type 11 during a large outbreak in Spanish military recruits. Am. J. Epidemiol. 1974; 99(6): 434–42. https://doi.org/10.1093/oxfordjournals.aje.a121632
- Li Q.G., Hambraeus J., Wadell G. Genetic relationship between thirteen genome types of adenovirus 11, 34, and 35 with different tropisms. Intervirology. 1991; 32(6): 338–50. https://doi.org/10.1159/000150218
- Yang Z., Zhu Z., Tang L., Wang L., Tan X., Yu P., et al. Genomic analyses of recombinant adenovirus type 11a in China. J. Clin. Microbiol. 2009; 47(10): 3082–90. https://doi.org/10.1128/JCM.00282-09
- Seto D., Chodosh J., Brister J.R., Jones M.S. Using the whole-genome sequence to characterize and name human adenoviruses. J. Virol. 2011; 85(11): 5701–2. https://doi.org/10.1128/JVI.00354-11
- Seto D., Jones M.S., Dyer D.W., Chodosh J. Characterizing, typing, and naming human adenovirus type 55 in the era of whole genome data. J. Clin. Virol. 2013; 58(4): 741–2. https://doi.org/10.1016/j.jcv.2013.09.025
- Centers for Disease Control and Prevention (CDC). Civilian outbreak of adenovirus acute respiratory disease – South Dakota, 1997. MMWR Morb. Mortal. Wkly Rep. 1998; 47(27): 567–70.
- Kajon A.E., Mistchenko A.S., Videla C., Hortal M., Wadell G., Avendaño L.F. Molecular epidemiology of adenovirus acute lower respiratory infections of children in the south cone of South America (1991–1994). J. Med. Virol. 1996; 48(2): 151–6. https://doi.org/10.1002/(sici)1096-9071(199602)48:2%3C151::aid-jmv6%3E3.0.co;2-8
- Salama M., Amitai Z., Nutman A., Gottesman-Yekutieli T., Sherbany H., Drori Y., et al. Outbreak of adenovirus type 55 infection in Israel. J. Clin. Virol. 2016; 78: 31–5. https://doi.org/10.1016/j.jcv.2016.03.002
- Jing S., Zhang J., Cao M., Liu M., Yan Y., Zhao S., et al. Household transmission of human adenovirus type 55 in case of fatal acute respiratory disease. Emerg. Infect. Dis. 2019; 25(9): 1756–8. https://doi.org/10.3201/eid2509.181937
- Бурцева Е.И., Панова А.Д., Колобухина Л.В., Игнатьева А.В., Кириллова Е.С., Бреслав Н.В. и др. Эпидемический сезон 2021–2022 годов. Частота ко-инфекции респираторными вирусными патогенами. Эпидемиология и инфекционные болезни. 2023; 28(2): 67–77. https://doi.org/10.17816/EID321873
- Tamura K., Stecher G., Kumar S. MEGA11: Molecular Evolutionary Genetics Analysis Version 11. Mol. Biol. Evol. 2021; 38(7): 3022–7. https://doi.org/10.1093/molbev/msab120
- Houng H.S., Lott L., Gong H., Kuschner R.A., Lynch J.A., Metzgar D. Adenovirus microsatellite reveals dynamics of transmission during a recent epidemic of human adenovirus serotype 14 infection. J. Clin. Microbiol. 2009; 47(7): 2243–8. https://doi.org/10.1128/JCM.01659-08
- Kurskaya O.G., Prokopyeva E.A., Dubovitskiy N.A., Solomatina M.V., Sobolev I.A., Derko A.A., et al. Genetic Diversity of the Human Adenovirus C Isolated from Hospitalized Children in Russia (2019-2022). Viruses. 2024; 16(3): 386. https://doi.org/10.3390/v16030386
- Adenovirus infections, 2008 to 2020, Japan. IASR. 2021; 42(4): 67–9. Available at: https://id-info.jihs.go.jp/niid/en/iasr/12459-494te.html
- Sun H., Hu W., Wei Y., Hao Y. Review: Drawing on the development experiences of infectious disease surveillance systems around the world. China CDC Wkly. 2024; 6(41): 1065–74. https://doi.org/10.46234/ccdcw2024.220
- Ko J.H., Woo H.T., Oh H.S., Moon S.M., Choi J.Y., Lim J.U., et al. Ongoing outbreak of human adenovirus-associated acute respiratory illness in the Republic of Korea military, 2013 to 2018. Korean J. Intern. Med. 2021; 36(1): 205–13. https://doi.org/10.3904/kjim.2019.092
- Hughes J.J., Yang Y., Fries A.C., Maljkovic Berry I., Pollio A.R., Fung C.K., et al. Complete genome sequences of two human adenovirus type 55 isolates from South Korea and the United States. Microbiol. Resour. Announc. 2021; 10(5): e01347-20. https://doi.org/10.1128/MRA.01347-20
- Niang M.N., Diop N.S., Fall A., Kiori D.E., Sarr F.D., Sy S., et al. Respiratory viruses in patients with influenza-like illness in Senegal: Focus on human respiratory adenoviruses. PLoS One. 2017; 12(3): e0174287. https://doi.org/10.1371/journal.pone.0174287
- Hang J., Kajon A.E., Graf P.C.F., Berry I.M., Yang Y., Sanborn M.A., et al. Human adenovirus type 55 distribution, regional persistence, and genetic variability. Emerg. Infect. Dis. 2020; 26(7): 1497–505. https://doi.org/10.3201/eid2607.191707
- Wodrich H., Henaff D., Jammart B., Segura-Morales C., Seelmeir S., Coux O., et al. A capsid-encoded PPxY-motif facilitates adenovirus entry. PLoS Pathog. 2010; 6(3): e1000808. https://doi.org/10.1371/journal.ppat.1000808
- Wang F., De R., Han Z., Xu Y., Zhu R., Sun Y., et al. High-frequency recombination of human adenovirus in children with acute respiratory tract infections in Beijing, China. Viruses. 2024; 16(6): 828. https://doi.org/10.3390/v16060828
Дополнительные файлы