Identification of orthohantaviruses detected for the first time in the Republic of Belarus
- Authors: Semizhon P.A.1, Scheslenok E.P.1, Dubkov N.A.1, Sukhotskaya E.A.1, Stolbunova K.A.2, Popov I.V.3, Popov I.V.3, Alekseev A.Y.2, Kabwe E.4, Davidyuk Y.N.4
-
Affiliations:
- Republican Center for Hygiene, Epidemiology and Public Health of the Ministry of Health of the Republic of Belarus
- Research Institute of Virology, Federal Research Center for Fundamental and Translational Medicine
- Don State Technical University
- Institute of Fundamental Medicine and Biology, Kazan Federal University
- Issue: Vol 70, No 1 (2025)
- Pages: 87-98
- Section: ORIGINAL RESEARCHES
- URL: https://virusjour.crie.ru/jour/article/view/16719
- DOI: https://doi.org/10.36233/0507-4088-292
- EDN: https://elibrary.ru/rzopww
- ID: 16719
Cite item
Full Text
Abstract
Introduction. Monitoring of hemorrhagic fever with renal syndrome (HFRS) pathogens in the Republic of Belarus is necessary and relevant, since the number of HFRS cases in the population has increased in recent years, and genetic characteristics of the pathogens remain unidentified.
Aim of the study. Identification of orthohantaviruses circulating in the territory of the Republic of Belarus and defining of their genetic characteristics.
Materials and methods. Screening of 613 samples from small mammals caught in the territory of the Republic of Belarus was carried out by the real time PCR method using the test system «Belar-GLPS-PCR/RV». Positive samples were sequenced by the Sanger method. Comparative and phylogenetic analysis was carried out using the MegAlign programs from the Lasergene package (DNASTAR, USA) and MEGA 11.
Results. The primary screening yielded 32 PCR-positive samples (5.2%), of which 24 belonged to Puumala virus (PUUV) and 8 to Dobrava-Belgrade virus (DOBV). Three nucleotide sequences of the M-segment region of PUUV, two sequences of the 291-base pair (bp) M-segment region and one sequence of the 348-bp S-segment region of DOBV were sequenced. Comparative and phylogenetic analysis showed that the identified PUUV sequences belong to the Russian genetic lineage, to the same sublineage as the strains common in the Moscow and Kursk regions. The identified DOBV ssequences demonstrated the closest relationship to the strains from the central region of the European part of Russia.
Conclusion. The results of molecular biological analysis showed that PUUV circulates in the territory of the Republic of Belarus and is widespread. At the same time, DOBV was detected in four regions of the republic, which indicates an expansion of the range of this HFRS pathogen. In the Republic of Belarus, nucleotide sequences of orthohantaviruses were obtained for the first time and their molecular genetic analysis was carried out.
Keywords
Full Text
Введение
Геморрагическая лихорадка с почечным синдромом (ГЛПС) – острая вирусная природно-очаговая зоонозная инфекция человека, характеризующаяся интоксикацией, лихорадкой, почечными и геморрагическими проявлениями [1]. ГЛПС занимает ведущее место в мире по числу регистрируемых случаев среди природно-очаговых инфекций. Возбудители ГЛПС относятся к отряду Bunyavirales, семейству Hantaviridae и принадлежат к роду Orthohantavirus. Наиболее значимыми, вызывающими у людей заболевания разной степени тяжести, являются 5 видов вирусов рода Orthohantavirus Старого Света – это вирусы Хантаан/HNTV (Orthohantavirus hantanense); Добрава-Белград/DOBV (Orthohantavirus dobravaense); Сеул/SEOV (Orthohantavirus seoulense); Пуумала/PUUV (Orthohantavirus puumalaense) и Тула/TULV (Orthohantavirus tulaense) [2–5]. HNTV и SEOV наиболее характерны для азиатских стран, таких как Китай и Корея. DOBV и PUUV циркулируют в Скандинавских странах, на западе России, в Балканском регионе, а также в некоторых странах Центральной и Восточной Европы, включая Республику Беларусь (РБ). TULV долгое время считался непатогенным для человека, но в Европейской части России с 2015 г. фиксируются случаи ГЛПС, вызванные именно этим вирусом [5]. В 2015 г. во Франции и в 2019 г. в Германии были выявлены нуклеотидные последовательности (НП) TULV в сыворотке крови человека (номера в GenBank: KU297981 и MT993951).
К настоящему времени у людей выделяют две основные клинические формы хантавирусной инфекции: ГЛПС (возбудители HNTV; DOBV; SEOV; PUUV) и хантавирусный пульмональный синдром (возбудители – ортохантавирусы Нового Света Sin-Nombre; Black Creek; New York; Bayou; Andes; Laguna Negra).
В Европе DOBV является наиболее опасным для человека: он вызывает ГЛПС с летальностью до 12% [6, 7]. В соответствии с географическими ареалами его естественных хозяев, мышей рода Apodemus, DOBV образует ряд генетических линий [8]. Линия DOBV-Af, представленная исходным изолятом DOBV из Словении (Slo/Af), связанная с A. flavicollis (Af), ассоциирована с тяжелыми случаями ГЛПС в Балканском регионе. У A. agrarius (Aa) были обнаружены две линии хантавируса: линия DOBV-Aa, представленная изолятами клеточной культуры SK/Aa из Словакии, и линия Lipetsk/Aa из России, типичная для Центральной Европы и центрально-европейской части России [3, 9], где она ассоциирована с ГЛПС легкой/умеренной тяжести. Для DOBV-подобного вируса Сааремаа (SAAV), представленного изолятом клеточной культуры Saa/160V из Эстонии, Северо-Восточной Европы [10], до сих пор окончательно не выявлена связь с клиническими случаями заболевания. Случаи ГЛПС средней и тяжелой степени течения заболевания на территории Краснодарского края РФ были связаны с линией DOBV-Ap, представленной штаммом Sochi/Ap. Резервуаром этого штамма являлась кавказская лесная мышь A. ponticus (Ap) [11].
У PUUV в настоящее время различают 8 линий: центрально-европейскую (CE), датскую (DAN), южно-скандинавскую (S-SCA), северо-скандинавскую (N-SCA), финскую (FIN), латвийскую (LAT), альпийско-адриатическую (ALAD) и русскую (RUS) [4]. В Российской Федерации идентифицированы две генетические линии PUUV: RUS – на большинстве территорий европейской части России (ЕЧР), и FIN – на севере ЕЧР и в западной Сибири [12].
Возбудитель ГЛПС является оболочечным вирусом с геномом, представленным одноцепочечной «−»РНК, состоящей из 3 сегментов: малого (S), среднего (M) и большого (L). Каждый геномный сегмент имеет единственную открытую рамку считывания (ORF) и кодирует нуклеокапсидный белок (N-белок), предшественника гликопротеинов мембраны Gn и Gc и РНК-зависимую РНК-полимеразу соответственно [13].
Резервуаром вирусов семейства Hantaviridae в природной среде служат мышевидные грызуны, насекомоядные и представители отряда рукокрылых [14‒16]. Природные очаги ГЛПС расположены чаще всего в определенных ландшафтно-географических зонах, таких как пойменные леса, овраги, луга, которые в значительной степени представлены на территории РБ. В РБ доминирующим видом грызунов ‒ резервуаров хантавирусов является рыжая полевка [17]. Грызуны переносят инфицирование в виде латентного вирусоносительства. Возбудитель выделяется во внешнюю среду с калом, мочой, слюной. Передача возбудителя между грызунами осуществляется в основном воздушным или воздушно-пылевым путем [1].
Заболеваемость ГЛПС характеризуется выраженной сезонностью: с апреля по декабрь. По многолетним данным, в РБ пик заболеваемости приходится на сентябрь–ноябрь. С января по март заболеваемость прекращает фиксироваться, что связано с сокращением численности мышевидных грызунов в зимнее время. Помимо сезонных, существуют еще и годовые колебания заболеваемости, которые составляют 3–4 года. По данным ведомственной отчетности, по состоянию на апрель 2024 г. в РБ зарегистрировано 483 природных очага, из них: в Брестской области – 34 (7% от всех зарегистрированных), Витебской области – 298 (61,7% соответственно), Гомельской области – 59 (12,2%), Гродненской области – 8 (1,7%), Минской области – 42 (8,7%), Могилевской области – 42 (8,7%).
Особенностью ГЛПС является 100% восприимчивость человека независимо от пола и возраста, а также отсутствие контагиозности (больной не является источником инфекции и не опасен для окружающих). Люди заражаются при соприкосновении с объектами внешней среды, которые инфицированы грызунами. Заражение человека происходит преимущественно воздушно-пылевым путем при вдыхании. Инкубационный период продолжается от 4 до 49 сут.
Случаи заболевания ГЛПС в РБ ежегодно регистрируются с 1991 г. В течение первого десятилетия 1991–2001 гг. отмечались единичные случаи ГЛПС: всего 24 случая за весь период времени: 1991 г. – 3 случая; 1992 г. – 1; 1993 г. – 1; 1994 г. – 1; 1995 г. – 0; 1996 г. – 0; 1997 г. – 0; 1998 г. – 5 (из них один случай летальный); 1999 г. – 7; 2000 г. – 5 (из них один случай летальный); 2001 г. – 2. Однако в последующие годы в РБ регистрировался значительный рост заболеваемости ГЛПС: с 2009 г. по сентябрь 2016 г. выявлено 390 случаев инфекции, из них только в 2012 г. 59 случаев (показатель заболеваемости – 0,6 на 100 тыс. населения); значительный вклад в формирование среднереспубликанского показателя заболеваемости ГЛПС вносила Могилевская область, в которой в 2015 г. наблюдалось превышение республиканского уровня заболеваемости ГЛПС в 4,9 раза (показатели заболеваемости на 100 тыс. населения составили 3,55 для Могилевской области и 0,72 для РБ), что в целом свидетельствует об эпидемическом подъеме ГЛПС [18]. С 2018 по 2020 г. на территории РБ зафиксировано 274 случая заболевания ГЛПС, из которых 54,3% пришлось на 2019 г. – 1,58 на 100 тыс. населения республики (149 случаев заболевания) [17]. В Минске в 2020 г. зарегистрировано 5 случаев заболевания ГЛПС (0,25 на 100 тыс. населения); в 2021 г. – 6 случаев (0,29 на 100 тыс.); в 2022 г. – 7 случаев (0,35 на 100 тыс.) [19].
Таким образом, проведение мониторинга возбудителей ГЛПС на территориях РБ является необходимым и актуальным.
Цель работы ‒ выявление ортохантавирусов, циркулирующих на территории РБ, и определение их генетических характеристик.
Материалы и методы
Для выявления вирусов – возбудителей ГЛПС был проведен отлов и идентификация мелких млекопитающих в природных и антропургических очагах на территориях Могилевской, Минской, Витебской, Гродненской, Гомельской и Брестской областей РБ за период с 2016 по 2020 г. (рис. 1). Всего было получено 613 проб биологического материала от представителей 9 видов отряда грызунов (Rodentia) и 2 видов отряда насекомоядных (Eulipotyphla). Видовой состав грызунов и количество обследованных особей составило: рыжая полевка (Myodes glareolus) – 93; желтогорлая мышь (A. flavicollis) – 146; лесная мышь (A. sylvaticus) – 39; полевка обыкновенная (Microtus arvalis) – 40; домовая мышь (Mus musculus) – 189; полевая мышь (A. agrarius) – 54; крыса серая (Rattus norvegicus) – 25; полевка-экономка (M. oeconomus) – 21; крыса черная (R. rattus) – 2. Видовой состав насекомоядных и количество обследованных особей составило: бурозубка обыкновенная (Sorex araneus) – 3; кутора обыкновенная (Neomys fodiens) – 1.
Рис. 1. Локализация точек отлова мелких млекопитающих на территории Республики Беларусь.
Fig. 1. Locations of small mammal capture points on the territory of the Republic of Belarus.
Авторы подтверждают соблюдение институциональных и национальных стандартов по использованию лабораторных животных в соответствии с «Consensus author guidelines for animal use» (IAVES 23 July 2010). Протокол исследования одобрен Комитетом по биомедицинской этике ФГБНУ «Научно-исследовательский институт экспериментальной и клинической медицины» (Протокол № 31 от 10.12.2015).
В качестве материала для исследования использовали фрагменты легочной ткани грызунов и насекомоядных. В пробирку помещали фрагмент органа массой 20–30 мг, добавляли фосфатно-буферный физиологический раствор (PBS) в количестве 1 мл. Обработку содержимого пробирок проводили дезинтегратором модели Xenox D-54518 Niersbach в течение 30 с, после чего полученную взвесь центрифугировали при 1700 rcf (g) в течение 20 мин при температуре +4 °С. Надосадочную жидкость отбирали для экстракции РНК целевых вирусов.
Экстракцию общей РНК проводили с использованием набора реагентов «Рибо-преп» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия). Вирусную комплементарную ДНК синтезировали с использованием обратной транскриптазы «Реверта-L» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия). Индикацию вируса в биологическом материале осуществляли методом полимеразной цепной реакции (ПЦР) с помощью тест-системы с детекцией результатов в реальном времени – «Белар-ГЛПС-ПЦР/РВ» (ГУ «Республиканский центр гигиены, эпидемиологии и общественного здоровья» Минздрава Республики Беларусь, РБ). Реакцию, анализ и учет результатов проводили с использованием термоциклера IQ5 (Bio-Rad, США). Для регистрации накопления продуктов амплификации анализировали кривые накопления флуоресцентного сигнала по каналам FAM и ROX. Далее для идентификации фрагментов генов, обусловливающих видовую специфичность возбудителей ГЛПС, положительные образцы секвенировали по методу Сэнгера.
Для ПЦР-амплификации и секвенирования фрагментов генома положительных образцов участка M-сегмента PUUV и DOBV использовали праймеры, последовательности которых приведены ранее [20]. Для выявления участка S-сегмента DOBV были использованы праймеры Primer Dob-F: CGCGAAGCTTGCAACACTAGAGGAA и Primer Dob-R: GCGCCTCGAGAGCAGTTTGCCCAGT, разработанные нами на основе последовательностей из базы данных GenBank: AJ131673.1, AJ131672.1, EU188452.1, KC848500.1, AY961618.1, AJ616854.1, KC848499.1, GU904027.1, KC848501.1, AY533120.2, AY961615.1.
Для валидации специфичности, используемых в рамках исследования разработанной пары праймеров Dob-F и Dob-R, был проведен биоинформатический анализ методами in silico ПЦР. Так как область генома, кодирующая фрагмент нуклеокапсидного белка, к которому комплементарна эта пара праймеров, находится на S-сегменте генома хантавирусов [21‒23], в анализ были включены 45 записей НП S-сегмента вирусов семейства Hantaviridae из базы данных RefSeq [24] и дополнительно 4 записи S-сегментов ортохантавирусов Добрава-Белград, в том числе штаммов вируса Куркино из базы данных GenBank [25] (дата обращения к базам данных: 28.01.2025). Для проведения in silico ПЦР использовали инструмент «primesearch» из программного обеспечения EMBOSS (версия 6.6.0) [26]. Максимальное количество несовпадений нуклеотидов было установлено равным 2. Результаты были визуализированы в виде тепловой карты с использованием пакета ggplot2 (версия 3.5.1) [27] для языка программирования R (версия 4.4.2).
Очистку продуктов ПЦР-амплификации проводили с использованием набора QIAquick Gel Extraction Kit (Qiagen, США). Секвенирование очищенных продуктов проводили с использованием набора BigDye Terminator v. 3.1 Cycle Sequencing Kit (Applied Biosystems, США) на автоматическом анализаторе 3100 Genetic Analyzer (Applied Biosystems, США). Выравнивание НП выполняли в программном обеспечении Mega 11 с использованием алгоритма ClustalW [28].
Для сравнительного анализа и построения филогенетического дерева в качестве референсных были задействованы НП PUUV и DOBV, депонированные в базе данных GenBank NCBI.
Сравнительный анализ нуклеотидного и аминокислотного профиля был проведен в программе MegAlign из пакета Lasergene (DNASTAR, США).
Филогенетический анализ выполняли с помощью программы MEGA 11. Филогенетические деревья были рассчитаны по методу максимального правдоподобия с использованием моделей Tamura 3-parameter и Tamura-Nei для M- и S-сегментов соответственно. Индексы поддержки были рассчитаны для 1000 повторов. В качестве внешней группы для анализа последовательностей PUUV использовали TULV изолят «MarDNk29»; для анализа последовательностей DOBV ‒ HNTV изолят «HV004».
Результаты
Вирусная РНК возбудителей ГЛПС была выявлена в 32 образцах, что составило 5,2% инфицированности особей. РНК PUUV была обнаружена у 24 особей из 32 ПЦР-положительных, что составило 3,9% инфицированности от общего количества анализируемых образцов и 75% среди положительных. РНК DOBV была выявлена у 8 особей, процент зараженности которых составил 1,3% среди всех анализируемых и 25% среди положительных. РНК PUUV выявлена в легких грызунов из всех точек отлова (таблица).
Таблица. Результаты выявления вирусной РНК возбудителей ГЛПС в точках отлова на территории областей Республики Беларусь
Table. Results of detection of viral RNA of HFRS pathogens at capture points in regions of the Republic of Belarus
Область Region | Общее число образцов, шт. Total number of samples | Положительные образцы, шт. (% от общего числа) Positive samples, number (% of the total) | |
PUUV | DOBV | ||
Минская | Minsk | 221 | 9 (4,1) | 2 (0,9) |
Могилевская | Mogilev | 225 | 6 (2,7) | 4 (1,8) |
Гродненская | Grodno | 64 | 4 (6,3) | 1 (1,6) |
Гомельская | Gomel | 50 | 2 (4) | 1 (2) |
Брестская | Brest | 30 | 2 (6,7) | 0 |
Витебская | Vitebsk | 23 | 1 (4,4) | 0 |
∑ | 613 | 24 (3,9) | 8 (1,3) |
Результаты анализа специфичности посредством проведения in silico ПЦР продемонстрировали, что используемая пара праймеров способна детектировать область генома, кодирующую фрагмент нуклеокапсидного белка хантавирусов, принадлежащих в основном роду Ortohantavirus (NC_038273.1), в том числе DOBV (в частности, к штаммам Куркино), которые послужили основой для дизайна праймеров [29] (AJ131673.1, AJ131672.1, KP878312.1, KP878313.1), а также родам Actinovirus (NC_055639.1) и Mobatvirus (NC_078483.1) (рис. 2). Таким образом, была валидирована возможность используемых в исследовании пары праймеров детектирования НП, кодирующих нуклеокапсидный белок хантавирусов, в основном DOBV.
Рис. 2. Результаты анализа специфичности пары праймеров Dob-F и Dob-R посредством проведения in silico ПЦР с использованием записей нуклеотидных последовательностей S-сегментов геномов вирусов семейства Hantaviridae из баз данных RefSeq и GenBank.
*Отряд Primates представлен только человеком (Homo sapiens).
Fig. 2. Results of the specificity analysis of the primer pair Dob-F and Dob-R using in silico PCR based on S segment nucleotide sequences of viruses of family Hantaviridae from the RefSeq and GenBank databases.
*The order Primates is represented only by humans (Homo sapiens).
В результате прямого секвенирования было получено 6 сиквенсов (PUUV – 3; DOBV – 3). Для идентификации обнаруженных ортохантавирусов проводили сравнительный анализ полученных нуклеотидных и аминокислотных последовательностей (НП и АП соответственно).
В результате сравнительного анализа было установлено, что идентичность НП секвенированного участка генома длиной 325 пар нуклеотидов (п.н.) штаммов ортохантавируса из Могилевской области (Могилевский, Славгородский, Горецкий районы) между собой составила 99,4% и от 99,1 до 100% при сравнении АП. Сравнительный анализ НП выявленных и референсных штаммов продемонстрировал 79,1–91,4% идентичность с участком М-сегмента PUUV и 90,7–100% идентичность при сравнении соответствующих АП (Приложение, табл. 1). Таким образом, полученные НП были идентифицированы как фрагмент М-сегмента генома PUUV и депонированы в международную базу данных GenBank под номерами MH844481.1–MH844483.1.
Анализ НП длиной 348 п.н. одного штамма (из Шкловского района Могилевской области) выявил 84,8–94,9 и 95,7–99,1% идентичность для НП и АП соответственно, при сравнении с референсными последовательностями S-сегмента DOBV (Приложение, табл. 2). В то же время НП длиной 291 п.н. двух штаммов (из Могилевского района Могилевской области и Пуховичского района Минской области) продемонстрировали 91,4 и 96,9% идентичность НП и АП соответственно при сравнении между собой, а также 82,5–90,7% идентичность при сравнении с референсными НП М-сегмента DOBV и 90,6–97,9% при сравнении соответствующих АП (Приложение, табл. 3). Таким образом, три полученных НП были идентифицированы как фрагменты генома DOBV.
Наибольшее значение идентичности анализируемых последовательностей PUUV из Могилевской области было выявлено при сравнении со штаммом «Puumala orthohantavirus isolate Klishino» (91,4% идентичность при сравнении НП и 98,1–99,1% при сравнении АП), обнаруженным в Курской области (Россия) в 2019 г. (Приложение, табл. 1). Анализ последовательности S-сегмента обнаруженного изолята DOBV_N_25_621 из Шкловского района Могилевской области показал его максимальное сходство с изолятом «Kurkino/53Aa/98» из России 1998 г. (94,0 и 99,1% идентичность для НП и АП соответственно) (Приложение, табл. 2). Наибольший процент идентичности НП S-сегмента анализируемых образцов DOBV_N_2141 и DOBV_N_2201 из Могилевского района Могилевской области и Пуховичского района Минской области соответственно (89,3–90,7%) составил с изолятом вируса «Dobrava-Belgrade orthohantavirus isolate LT18/LK_11», выявленным в Литве в 2018 г. (Приложение, табл. 3).
По результатам филогенетического анализа установлено, что анализируемые изоляты PUUV группируются в отдельной субкладе, близкородственной к субкладе штаммов PUUV, циркулирующих в Курской и Московской областях, что может свидетельствовать об их принадлежности к одной сублинии в русской линии PUUV (рис. 3).
Рис. 3. Филогенетическое дерево, построенное для нуклеотидных последовательностей М-сегмента генома PUUV (позиции 1256–1580).
Фиолетовым цветом выделены последовательности выявленных штаммов PUUV.
Fig. 3. Phylogenetic tree constructed for the nucleotide sequences of the M-segment of the PUUV genome (positions 1256–1580).
The sequences of the identified PUUV strains are highlighted in purple.
По результатам филогенетического анализа, проведенного для S-сегмента последовательности штамма DOBV_N_25_621, было выявлено его близкое родство к субкладе с изолятами «Kurkino/53Aa/98» и «Aa1854/Lipetsk-02» из России (рис. 4).
Рис. 4. Филогенетическое дерево, построенное для нуклеотидных последовательностей S-сегмента генома DOBV (позиции 4–351).
Оранжевым цветом выделена последовательность выявленного штамма DOBV из Могилевской области (Шкловский район).
Fig. 4. Phylogenetic tree constructed for the nucleotide sequences of the S-segment of the DOBV genome (positions 4–351).
The sequence of the DOBV strain identified from the Mogilev region (Shklov district) is highlighted in orange.
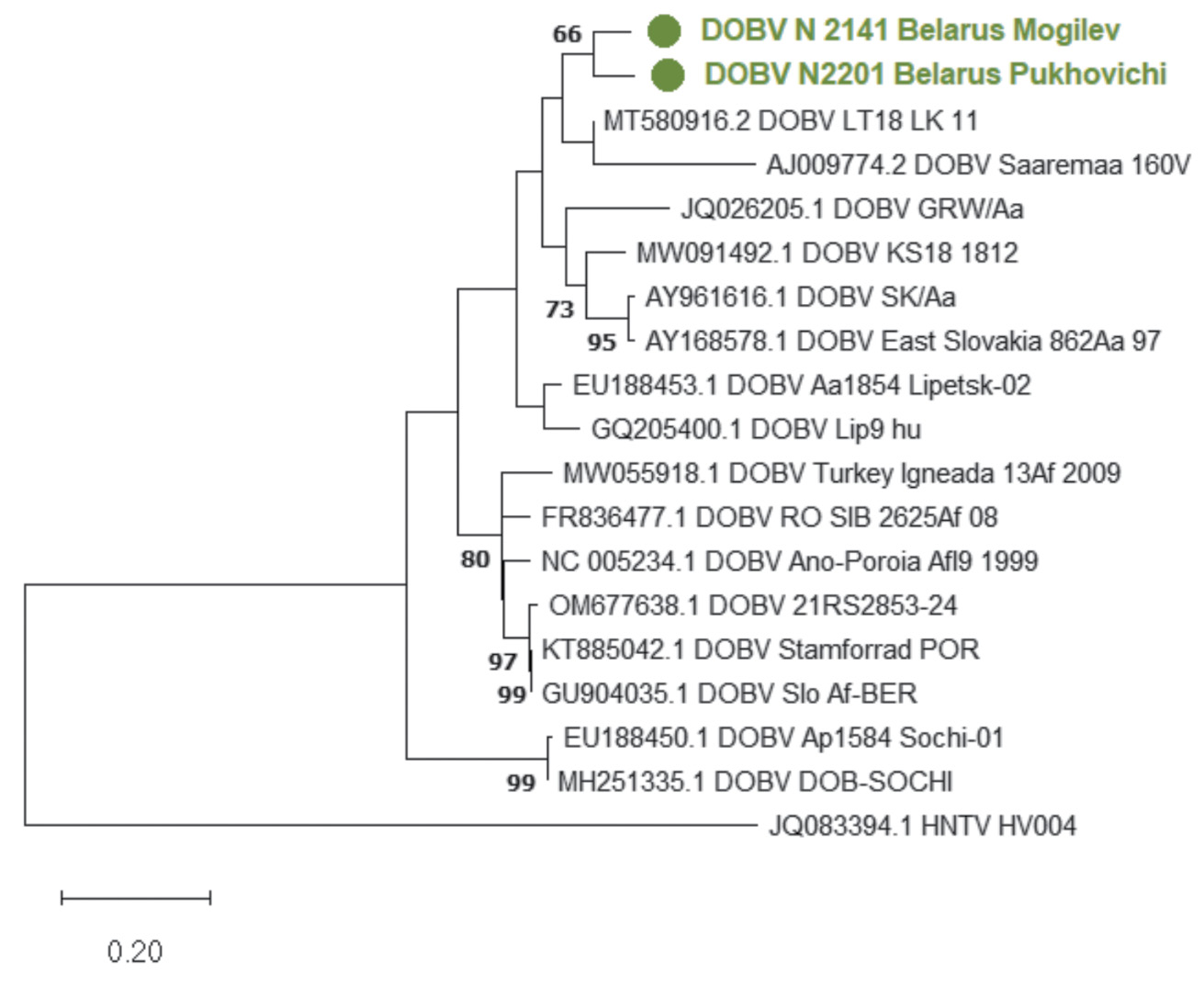
В результате филогенетического анализа, проведенного для М-сегмента последовательностей выявленных штаммов DOBV_N_2141 и DOBV_N_2201 из Могилевской и Минской областей соответственно, было установлено, что анализируемые последовательности группируются в отдельной субкладе на соседней ветке с субкладой, включающей изоляты «Dobrava-Belgrade orthohantavirus isolate LT18/LK_11» и «DOB/Saaremaa/160V» из Литвы и Эстонии соответственно (рис. 5).
Рис. 5. Филогенетическое дерево, построенное для нуклеотидных последовательностей М-сегмента генома DOBV (позиции 1269–1559).
Зеленым цветом выделены последовательности штамма DOBV, выявленные в Могилевской (Могилевский район) и в Минской области (Пуховичский район).
Fig. 5. Phylogenetic tree constructed for the nucleotide sequences of the M-segment of the DOBV genome (positions 1269–1559).
The sequences of the DOBV strain identified in Mogilev (Mogilev district) and Minsk region (Pukhovichi district) are highlighted in green.
Обсуждение
РБ относится к странам Европы, в которых ежегодно регистрируется значительное число заболеваний ГЛПС, вызываемого хантавирусами. Случаи заболевания ГЛПС в РБ ежегодно регистрируются с 1991 г., причем с 2009 г. в республике отмечается значительный рост заболеваемости. В 2012 г. было выявлено 59 случаев (показатель заболеваемости – 0,6 на 100 тыс. населения); значительный вклад в формирование среднереспубликанского показателя заболеваемости ГЛПС вносила Могилевская область, в которой в 2015 г. наблюдалось превышение республиканского уровня заболеваемости ГЛПС в 4,9 раза (показатели заболеваемости на 100 тыс. населения составили 3,55 для Могилевской области и 0,72 для республики), что в целом свидетельствует об эпидемическом подъеме ГЛПС. Источником заражения людей являются только грызуны – резервуарные хозяева патогенных хантавирусов, к которым на европейской территории относятся: рыжая полевка (вирус Пуумала) и полевая мышь (вирус Куркино). Таким образом, одной из актуальных задач для обеспечения профилактики и лечения ГЛПС является выявление циркуляции патогенов на территории и изучение их эволюции и особенностей строения геномов хантавирусов.
Вирус PUUV был выявлен на административных территориях всех областей республики, что вероятнее всего свидетельствует о его повсеместном распространении. Отмечается циркуляция вируса DOBV в Могилевской области, впервые выявленного в РБ и на территории данного региона в 2016 г. [30]. В настоящем исследовании вирус DOBV был впервые зарегистрирован на территориях Минской (Солигорский район), Гродненской (Островецкий район) и Гомельской (Жлобинский район) областей.
Результаты молекулярно-биологического анализа образцов из точек отлова, расположенных на территориях административных районов всех областей РБ, показали, что изоляты PUUV группируются в отдельной субкладе, близкородственной к субкладе вирусов, циркулирующих в Курской и Московской областях, что может свидетельствовать об их принадлежности к одной сублинии в русской линии PUUV. Вероятнее всего, это связано с генетической гомогенностью популяций вирусов, ассоциированных с определенной территорией и основным резервуаром вируса – рыжей полевкой.
В результате филогенетического анализа выявленных штаммов DOBV было установлено, что анализируемые последовательности группируются в отдельной субкладе с изолятами из России, Литвы и Эстонии, причем линия DOBV-Aa типична для Центральной Европы и центрально-европейской части России. Помимо этого, изоляты, выявленные в настоящем исследовании, имеют значительное генетическое подобие со штаммами, характерными для Эстонии, у которых отмечено значительное сходство с DOBV-подобным вирусом Сааремаа. Эти факты позволяют предположить, что выявленные штаммы, вероятнее всего, могут вызывать исключительно заболевания легкой/умеренной степени тяжести. Безусловно, антропогенное и техногенное воздействие на окружающую среду приводит к изменению биоценотической структуры очагов распространения инфекций и, как следствие, повышается участие грызунов в эпидемическом процессе. Однако на текущий момент на территории РБ не установлено тенденции к ухудшению эпидемиологической обстановки в отношении ГЛПС. При этом динамическое наблюдение за патогеном должно быть продолжено и усилено в целях контроля и предотвращения будущих угроз.
Заключение
Изучение структурно-функциональной организации геномов вирусов является важной фундаментальной и прикладной задачей современной генетики, особенно учитывая широкую распространенность заболеваний, вызываемых вирусами. Несмотря на то что случаи ГЛПС регулярно регистрируются среди населения на территории РБ, данные о генетических характеристиках ортохантавирусов с этой территории были впервые представлены авторами в настоящем исследовании.
Полученные данные свидетельствуют о коциркуляции двух видов ортохантавирусов – PUUV и DOBV – на территории РБ. Соотношение выявленных видов патогенов указывает на более широкую циркуляцию PUUV, нежели DOBV. По данным проведенного исследования наиболее эпидемически значимым на территории РБ является PUUV, так же как и на территории Европейской части России, где около 98% случаев ГЛПС ассоциировано с этим вирусом [31].
Таким образом, в целях предотвращения возможного ухудшения эпидемиологической ситуации в отношении природно-очаговых инфекций, в частности ГЛПС, необходимо продолжать совершенствование молекулярно-генетического мониторинга в РБ и на прилегающих союзных территориях. Данные о генетических последовательностях вирусов играют важную роль в различных аспектах науки, медицины и здравоохранения. Эти последовательности представляют собой уникальную информацию о строении и функциональности вирусов, которая может быть использована на практике для разработки диагностических систем, лекарств и вакцин.
About the authors
Pavel A. Semizhon
Republican Center for Hygiene, Epidemiology and Public Health of the Ministry of Health of the Republic of Belarus
Author for correspondence.
Email: pavel5555@tut.by
ORCID iD: 0000-0001-7986-8299
PhD, Head of the Laboratory of Biotechnology and Immunodiagnostics of Particularly Dangerous Infections
Belarus, 220099, MinskElena P. Scheslenok
Republican Center for Hygiene, Epidemiology and Public Health of the Ministry of Health of the Republic of Belarus
Email: elena_pavlovna@tut.by
ORCID iD: 0000-0001-8143-684X
PhD, Leading Researcher, Laboratory of Biotechnology and Immunodiagnostics of Particularly Dangerous Infections
Belarus, 220099, MinskNikita A. Dubkov
Republican Center for Hygiene, Epidemiology and Public Health of the Ministry of Health of the Republic of Belarus
Email: dubkov.nikita@gmail.com
ORCID iD: 0009-0007-8488-4307
Researcher at the Laboratory of Biotechnology and Immunodiagnostics of Particularly Dangerous Infections
Russian Federation, 220099, MinskElizaveta A. Sukhotskaya
Republican Center for Hygiene, Epidemiology and Public Health of the Ministry of Health of the Republic of Belarus
Email: elissuchozkaja5@gmail.com
ORCID iD: 0009-0007-0439-6363
Junior Researcher, Laboratory of Biotechnology and Immunodiagnostics of Particularly Dangerous Infections
Belarus, 220099, MinskKristina A. Stolbunova
Research Institute of Virology, Federal Research Center for Fundamental and Translational Medicine
Email: kristina.sunwo@yandex.ru
ORCID iD: 0000-0003-3376-945X
Junior Researcher, Laboratory of Virus Genomics and Evolution, Research Institute of Virology
Russian Federation, 630060, NovosibirskIgor V. Popov
Don State Technical University
Email: ipopov@donstu.ru
ORCID iD: 0000-0002-9223-8731
Junior Researcher, Faculty of Bioengineering and Veterinary Medicine
Russian Federation, 344003, Rostov-on-DonIlia V. Popov
Don State Technical University
Email: ivpopov@donstu.ru
ORCID iD: 0000-0001-7947-1654
Junior Researcher, Faculty of Bioengineering and Veterinary Medicine
Russian Federation, 344003, Rostov-on-DonAlexander Y. Alekseev
Research Institute of Virology, Federal Research Center for Fundamental and Translational Medicine
Email: ayalekseev@frcftm.ru
ORCID iD: 0000-0003-0015-9305
PhD, Senior Researcher at the Research Institute of Virology
Russian Federation, 630060, NovosibirskEmmanuel Kabwe
Institute of Fundamental Medicine and Biology, Kazan Federal University
Email: emmanuelkabwe@ymail.com
ORCID iD: 0000-0003-4328-8190
PhD, Senior Research Fellow at Laboratory OpenLab «Gene and Cell Technologies», Scientific and Clinical Center for Precision and Regenerative Medicine
Russian Federation, 420008, KazanYuri N. Davidyuk
Institute of Fundamental Medicine and Biology, Kazan Federal University
Email: davi.djuk@mail.ru
ORCID iD: 0000-0002-4409-2942
PhD, Senior Research Fellow at Laboratory OpenLab «Gene and Cell Technologies», Scientific and Clinical Center for Precision and Regenerative Medicine
Russian Federation, 420008, KazanReferences
- Morozov V.G., Ishmukhametov A.A., Dzagurova T.K., Tkachenko E.A. Clinical manifestations of hemorrhagic fever with renal syndrome in Russia. Meditsinskiy sovet. 2017; (5): 156–61. https://doi.org/10.21518/2079-701X-2017-5-156-161 https://elibrary.ru/yorunb (in Russian)
- Yashina L.N., Smetannikova N.A., Kompanets G.G., Zdanovskaya N.I., Ivanov L.I. Molecular epidemiology of pathogenic hantaviruses in the far east of Russia, 2015–2018. Problemy osobo opasnykh infektsii. 2019; (4): 102–8. https://doi.org/10.21055/0370-1069-2019-4-102-108 https://elibrary.ru/cnooaj (in Russian)
- Klempa B, Tkachenko E.A., Dzagurova T.K., Yunicheva Y.V., Morozov V.G., Okulova N.M., et al. Hemorrhagic fever with renal syndrome caused by 2 lineages of Dobrava hantavirus, Russia. Emerg. Infect. Dis. 2008; 14(4): 617–25. https://doi.org/10.3201/eid1404.071310
- Castel G., Chevenet F., Razzauti M., Murri S., Marianneau P., Cosson J.F., et al. Phylogeography of Puumala orthohantavirus in Europe. Viruses. 2019; 11(8): 679. https://doi.org/10.3390/v11080679
- Schlegel M., Kindler E., Essbauer S.S., Wolf R., Thiel J., Groschup M.H., et al. Tula virus infections in the Eurasian water vole in Central Europe. Vector Borne Zoonotic Dis. 2012; 12(6): 503–13. https://doi.org/10.1089/vbz.2011.0784
- Avsic-Zupanc T., Petrovec M., Furlan P., Kaps R., Elgh F., Lundkvist A. Hemorrhagic fever with renal syndrome in the Dolenjska region of Slovenia-a 10-year survey. Clin. Infect. Dis. 1999; 28(4): 860–5. https://doi.org/10.1086/515185
- Papa A., Antoniadis A. Hantavirus infections in Greece-an update. Eur. J. Epidemiol. 2001; 17(2): 189–94. https://doi.org/10.1023/a:1017987104363
- Kruger D.H., Klempa B. Dobrava-Belgrade virus. In: Molecular Detection of Human Viral Pathogens. Boca Raton: CRC Press; 2011: 631–6.
- Klempa B., Stanko M., Labuda M., Ulrich R., Meisel H., Krüger D.H. Central European Dobrava hantavirus isolate from a striped field mouse (Apodemus agrarius). J. Clin. Microbiol. 2005; 43(6): 2756–63. https://doi.org/10.1128/JCM.43.6.2756-2763.2005
- Nemirov K., Vapalahti O., Lundkvist A., Vasilenko V., Golovljova I., Plyusnina A., et al. Isolation and characterization of Dobrava hantavirus carried by the striped field mouse (Apodemus agrarius) in Estonia. J. Gen. Virol. 1999: 80(2): 371–9. https://doi.org/10.1099/0022-1317-80-2-371
- Tkachenko E.A., Okulova N.M., Yunicheva YU.V., Morzunov S.P., Khaibulina S.F., Ryabova T.E., et al. The epizootological and virological characteristics of a natural hantavirus infection focus in the subtropic zone of the Krasnodarsk Territory. Voprosy virusologii. 2005; 50(3): 14–9. https://elibrary.ru/hsghsn (in Russian)
- Blinova E., Deviatkin A., Makenov M., Popova Y., Dzagurova T. Evolutionary Formation and Distribution of Puumala Virus Genome Variants, Russia. Emerg. Infect. Dis. 2023; 29(7): 1420–4. https://doi.org/10.3201/eid2907.221731
- Plyusnin A., Vapalahti O., Vaheri A. Hantaviruses: Genome structure, expression and evolution. J. Gen. Virol. 1996; 77(11): 2677–87. https://doi.org/10.1099/0022-1317-77-11-2677
- Tunik T.V.1, Arbatskaya E.V.1, Lyapunov A.V., Khasnatinov M.A., Petrova I.V., Manzarova E.L., et al. To hantavirus infection in people and small mammals in Baikal area. Byulleten’ Vostochno-Sibirskogo nauchnogo tsentra Sibirskogo otdeleniya Rossiiskoi akademii meditsinskikh nauk. 2014; (2): 71–6. https://elibrary.ru/sizjln (in Russian)
- Yashina L.N., Abramov S.A., Dupal T.A., Yakimenko V.V., Tantsev A.K., Malyshev B.S., et al. Hantaviruses in insectivore populations in Siberia. Problemy osobo opasnykh infektsii. 2018; (4): 89–93. https://doi.org/10.21055/0370-1069-2018-4-89-93 https://elibrary.ru/yteidj (in Russian)
- Ohlopkova O.V., Stolbunova K.A., Popov I.V., Popov I.V., Kabwe E., Davidyuk Y.N., et al. Detection of Brno loanvirus (Loanvirus brunaense) in common noctule bats (Nyctalus noctula) in Southern Russia. Braz. J. Microbiol. 2024. https://doi.org/10.1007/s42770-024-01587-5
- Dorozhenkova T.E. Hemorrhagic fever with renal syndrome and its epidemiological characteristics in the republic of Belarus and Minsk. Medicus. 2020; (3): 71–5. https://elibrary.ru/klhcld (in Russian)
- State report «On the sanitary and epidemiological situation in the Republic of Belarus in 2015». Minsk; 2016. (in Russian)
- State report «Public health and the environment of Minsk in 2022: achieving the Sustainable Development Goal» Minsk; 2023. (in Russian)
- Lee H.W., Calisher C., Schmaljohn C. Manual of hemorrhagic fever with renal syndrome and hantavirus pulmonary syndrome. Seoul; 1998.
- Alfadhli A., Love Z., Arvidson B., Seeds J., Willey J., Barklis E. Hantavirus nucleocapsid protein oligomerization J. Virol. 2001; 75(4): 2019–23. https://doi.org/10.1128/JVI.75.4.2019-2023.2001
- Severson W.E., Xu X., Jonsson C.B. Cis-acting signals in encapsidation of Hantaan virus S-segment viral genomic RNA by its N protein. J. Virol. 2001; 75(6): 2646–52. https://doi.org/10.1128/JVI.75.6.2646-2652.2001
- Welke R.W., Sperber H.S., Bergmann R., Koikkarah A., Menke L., Sieben C., et al. Characterization of Hantavirus N protein intracellular dynamics and localization. Viruses. 2022; 14(3): 457. https://doi.org/ 45710.3390/v14030457
- Pruitt K.D., Tatusova T., Maglott D.R. NCBI reference sequences (RefSeq): a curated non-redundant sequence database of genomes, transcripts and proteins. Nucleic Acids Res. 2005; 33: D501–4. https://doi.org/10.1093/nar/gki025
- Sayers E.W., Cavanaugh M., Clark K., Ostell J., Pruitt K.D., Karsch-Mizrachi I. GenBank. Nucleic Acids Res. 2020; 48(D1): D84–6. https://doi.org/10.1093/nar/gkz956
- Rice P., Longden I., Bleasby A. EMBOSS: the European molecular biology open software suite. Trends Genet. 2000; 16(6): 276–7. https://doi.org/10.1016/s0168-9525(00)02024-2
- Wickham H., Wickham H. Data Analysis. Springer; 2016: 189–201. https://doi.org/10.1007/978-3-319-24277-4_9
- Tamura K., Stecher G., Kumar S. MEGA11: Molecular evolutionary genetics analysis, version 11. Mol. Biol. Evol. 2021; 38(7): 3022–7. https://doi.org/10.1093/molbev/msab120
- Scheslenok E.P., Semizhon P.A., Kasnitskaya T.N., Omel’yanovich O.G., Voitenko N.T., Chaika A.V., et al. Hantaviruses circulating in the territory of the Republic of Belarus. In: Proceedings of the IX All-Russian Scientific and Practical Conference with International Participation «Molecular Diagnostics – 2017». Volume 2. Moscow; 2017: 350–1. (in Russian)
- Scheslenok E.P., Fomina E.G., Semizhon P.A., Grigorieva E.E., Shkolina T.V., Vladyko A.S., et al. Preparation and description of recombinant polypeptides representing antigenically active regions of hantavirus nucleocapsid proteins. Zdravookhranenie (Minsk). 2018; (4): 16–21. https://elibrary.ru/xtcsrv (in Russian)
- Tkachenko E.A., Ishmukhametov A.A., Dzagurova T.K., Bernshtein A.D., Morozov V.G., Siniugina A.A., et al. Hemorrhagic fever with renal syndrome, Russia. Emerg. Infect. Dis. 2019; 25(12): 2325–8. https://doi.org/10.3201/eid2512.181649
Supplementary files