Prevalence and phylogenetic analysis of cytomegalovirus (Orthoherpesviridae: Cytomegalovirus: Cytomegalovirus humanbeta5) genetic variants from children and immunocompromised patients in central Russia
- Authors: Van’kova O.E.1, Brusnigina N.F.1, Novikova N.A.1
-
Affiliations:
- Nizhny Novgorod Research Institute of Epidemiology and Microbiology named after Academician I.N. Blokhina
- Issue: Vol 69, No 6 (2024)
- Pages: 535-545
- Section: ORIGINAL RESEARCHES
- URL: https://virusjour.crie.ru/jour/article/view/16696
- DOI: https://doi.org/10.36233/0507-4088-277
- EDN: https://elibrary.ru/epnrav
- ID: 16696
Cite item
Full Text
Abstract
Introduction. Cytomegalovirus (CMV) is a DNA-containing virus that is widespread worldwide and is of great importance in infectious pathology of children and adults.
The aim of this study is to evaluate the prevalence of CMV among children and immunocompromised patients in the Nizhny Novgorod region (central Russia) and to perform a phylogenetic analysis of the identified strains.
Materials and methods. DNA samples of CMV detected in frequently ill children and adult recipients of solid organs were studied. The genetic diversity of CMV was assessed for two variable genes: UL55(gB) and UL73(gN), using NGS technology on the Illumina platform. Phylogenetic trees were constructed in the MEGA X program, the reliability of the cluster topology on the trees was confirmed using the rapid bootstrap method based on the generation of 500 pseudo-replicas.
Results. Circulation of 5 CMV genotypes by gene UL55(gB) and 5 genotypes by gene UL73(gN) was established in the territory of the Nizhny Novgorod region. While genotypes gB1 and gB2 dominated both in children and in adults, genotype gB5 was detected only in children. The spectrum of gN genotypes was more diverse: genotypes gN4a and gN3b prevailed in children, and gN1 and gN4b genotypes were predominant in adults. The obtained results allowed us to establish the similarity of the landscape of CMV genotypes circulating in Russia (Nizhny Novgorod region), Brazil, China and the USA.
Conclusion. The obtained data indicate the similarity of the landscape of CMV genotypes circulating in Russia (Nizhny Novgorod region), Brazil, China and the USA: in children, the predominant genotypes are gB1 (40.0%), gB2 (33.3%), gN4a (42.8%), and gN3b (28.6%), while in adults (recipients of solid organs) genotypes gB1 (37.5%), gB2 (37.5%), gN1 (26.3%), and gN4b (26.3%) are prevailed.
Full Text
Введение
Цитомегаловирус (ЦМВ) (Orthoherpesviridae: Cytomegalovirus: Cytomegalovirus humanbeta5) относится к крупным цитоплазматическим ДНК-вирусам и имеет сферический/плейоморфный вирион диаметром 150‒200 нм. Вирион имеет сложную структур ‒ икосаэдрический капсид (Т = 16, 162 капсомера), содержащий линейную двунитевую ДНК, который окружен липидной оболочкой, включающей множество гликопротеиновых комплексов и элементы аморфного белкового матрикса (тегумента) [1].
ЦМВ широко распространен по всему миру. Инфекция, вызываемая ЦМВ, принадлежит к категории социально значимых и представляет серьезную проблему для современного здравоохранения. Это связано с высокой долей инфицированности населения (50‒90% в большинстве стран мира), полиморфизмом клинических проявлений, многообразием путей и факторов передачи инфекции [2]. У здоровых людей первичная цитомегаловирусная инфекция (ЦМВИ) протекает бессимптомно, но у иммунокомпрометированных пациентов (реципиентов солидных органов, ВИЧ-инфицированных, онкобольных) ЦМВИ имеет зачастую тяжелое течение, приводящее к различного рода осложнениям, включая пневмонию, гепатит, энцефалит, колит, увеит, ретинит, слепоту, и даже к летальному исходу. Серьезной проблемой является заражение ЦМВ реципиентов крови и солидных органов, поскольку фактором передачи инфекции может быть не только перелитая кровь, но и орган [3, 4].
ЦМВИ является основной причиной врожденных пороков развития у детей раннего возраста. В разных странах мира частота внутриутробного заражения плода ЦМВ колеблется от 0,3 до 3%, в России ‒ от 0,1 до 2,8% [5, 6]. В Центральной части России (Нижегородская область) частота выявления ДНК ЦМВ у детей (580 человек) варьирует от 3,8 до 18,9%, в зависимости от клинической формы заболевания, при этом установлены высокие показатели инфицированности детей в возрасте от 1 года до 3 лет (56,1‒58,5%) [7]. Частота обнаружения ДНК ЦМВ у 160 пациентов, перенесших трансплантацию солидных органов (печени и почек), составила 37,7% [8].
Значимость ЦМВИ в инфекционной патологии детей и взрослых определяет необходимость молекулярно-генетического изучения возбудителя. Геном ЦМВ значительно больше, чем геномы других герпесвирусов, и представлен линейной двухцепочечной ДНК длиной 220‒240 кб, кодирующей около 165 генов (открытых рамок считывания, ОРС) [1]. Геном ЦМВ имеет два уникальных участка ДНК ‒ длинный регион (UL) и короткий регион (US), фланкированые инвертированными повторами b (TRL/IRL) и с (IRS/TRS) [9]. Следует отметить, что бо́льшая часть генома разных штаммов ЦМВ достаточно консервативна, но отдельные гены могут демонстрировать высокую вариабельность [10].
Несмотря на отсутствие в настоящее время общепринятой системы генотипирования ЦМВ, для дифференциации штаммов часто используются вариабельные гены: UL55(gB), UL73(gN), UL74(gO), UL144-TNRF.
Ген UL55 кодирует гликопротеин gB (906 a.o), который относится к гликопротеиновому комплексу gC-I, играет значительную роль в присоединении и проникновении вируса в клетку хозяина и является основной мишенью для нейтрализующих антител. Кроме того, он участвует в процессе вирусной репликации, отвечает за слияние клеток и распространение вируса от клетки к клетке. В настоящее время известно 7 основных gB-генотипов ЦМВ: gB-1, gB-2, gB-3, gB-4, gB-5, gB-6, gB-7. Область основной вариабельности расположена в позиции 448‒481 а.о. [11].
Ген UL73 кодирует гликопротеин gN (138 а.о.), который индуцирует образование нейтрализующих антител и участвует в присоединении вируса к клетке хозяина. Гликопротеин gN является самым полиморфным белком среди белков ЦМВ человека [12]. Показано, что аминокислотные замены присутствуют в N-концевой области белка и захватывают участок первых 87 а.о. Данный участок расположен на поверхности вирусной оболочки. Различают 7 gN-генотипов ЦМВ: gN-1, gN-2, gN3a, gN3b и gN4a, gN4b, gN4c [12, 13].
В России исследования ЦМВ в основном направлены на изучение эпидемиологических и клинических аспектов инфекции среди разных групп населения. Знания о пейзаже генотипов ЦМВ и региональных особенностях их циркуляции необходимы для эффективного эпидемиологического надзора за ЦМВИ и разработки отечественных вакцин.
Цель работы – оценить филогенетические взаимоотношения штаммов ЦМВ, обнаруженных у детей и иммунокомпрометированного населения Нижегородского региона, на основе анализа нуклеотидных последовательностей генов UL55 и UL73.
Материалы и методы
Материалом для исследования служили 52 образца ДНК, которая была выделена из биологических субстратов, полученных от пациентов, перенесших трансплантацию солидных органов и находящихся на стационарном лечении во ФБУЗ «Приволжский окружной медицинский центр» ФМБА России, а также часто болеющих детей, обратившихся в медицинские организации Нижнего Новгорода и Нижегородской области. Оценку генетического разнообразия ЦМВ проводили по двум вариабельным генам: UL55(gB) и UL73(gN).
Исследование проводилось при добровольном информированном согласии пациентов и законных представителей несовершеннолетних пациентов. Протокол исследования одобрен Локальным этическим комитетом ФБУН «Нижегородский НИИ эпидемиологии и микробиологии им. академика И.Н. Блохиной» Федеральной службы по надзору в сфере защиты прав потребителей и благополучия человека (Роспотребнадзор) (Протокол № 2 от 19.09.2024).
Выделение и очистку ДНК осуществляли сорбционным методом с применением наборов «ДНК-сорб В» (ФСР 2012/14019) и «ДНК-сорб АМ» (ФСР 2007/00183) согласно инструкции производителя (ЦНИИЭ, Россия). Выделенную и очищенную ДНК хранили при −70 °С до момента анализа.
Амплификацию выделенной ДНК проводили в термоциклере «Терцик МС-2» («ДНК-технология», Россия) и Rotor Gene 6000 (Corbett Research, Австралия).
Продукты амплификации детектировали путем горизонтального электрофореза в 1,8% агарозном геле с использованием трис-боратной-ЭДТА (TBE) буферной системы (pH 8,2). Визуализацию и учет результатов реакции осуществляли с помощью гель-документирующей видеосистемы Geldoc EZ и программы Image Lab 5.0 (Bio-Rad).
Секвенирование фрагментов выполняли с применением технологии секвенирования нового поколения (NGS) на платформе MiSeq и iSeq (Illumina, США). Концентрацию ДНК в образцах определяли на флуориметре Qubit (Invitrogen, Австрия) с использованием набора Qubit DNA HS Assay Kit (Invitrogen, США). Библиотеки ДНК для секвенирования готовили с помощью набора Nextera XT DNA Sample Preparation Kit (Illumina, США), ShotGun SG GM (ООО «Сесана», Россия) и комплекта индексированных праймеров для двойного баркодирования для секвенаторов Illumina (Raissol, Россия), в соответствии с инструкцией производителя. Качество подготовленной библиотеки ДНК для секвенирования оценивали на флуориметре Qubit (Thеrmofisher, США) и автоматизированной системе капиллярного гель-электрофореза QIAxcel Advanced System (QIAGEN, Германия) с применением набора реагентов для быстрого разделения фрагментов ДНК QIAxcel DNA Fast Analysis Kit (3000) (QIAGEN, Германия) и программного обеспечения QIAxcel ScreenGel (QIAGEN, Германия). Для секвенирования использовали набор MiSeq reagent kit v2 на 500 циклов и iSeq 100 i1 Reagent v2 (300-cycle) (Illumina, США).
Выравнивание и сборку полученных коротких чтений относительно референс-генома осуществляли с использованием встроенного в секвенатор программного обеспечения и программы CLC Genomics Workbench 5.5 (CLC bio, США). Анализ последовательности генов (UL55(gB) и UL73(gN)) ЦМВ проводили с использованием алгоритма BLAST и пакета программ, представленных на сервере NCBI (http://www.ncbi.nlm.nih.gov/blast). Выравнивание нуклеотидных последовательностей выполняли с помощью программы CLUSTALX 2.0 (http://bips.ustrasbg.fr/fr/Documentation/ClustalX/).
Генетическое разнообразие исследуемых изолятов ЦМВ оценивали с использованием анализа нуклеотидных последовательностей генов UL55 и UL73 ЦМВ, основанного на биоинформатических методах. Для этого были амплифицированы и секвенированы фрагменты генов ЦМВ UL55(gB) и UL73(gN) длиной 356 и 406 п.н. соответственно. Для проведения филогенетического анализа из международной базы данных GenBank были отобраны нуклеотидные последовательности гена UL55(gB) 12 референс-штаммов и 37 клинических изолятов ЦМВ, полученных в разных странах Европы (Италии, Испании, Бельгии, Великобритании), США, Китае, Мексике, Индии, Египте, а также гена UL73(gN) – 7 референс-штаммов и 39 клинических изолятов ЦМВ из стран Европы (Италии, Испании, Великобритании), США, Китая, Индии. Дифференциацию кластеров проводили на основе сравнения уровня сходства нуклеотидных последовательностей, рассчитанного методом попарных дистанций (p-distans), как внутри, так и между отдельными кластерами. На основе выбранных последовательностей в программе MEGA-X (версия 10.2.6) были построены филогенетические деревья с использованием метода максимального правдоподобия (Мaximum likelihood). Достоверность топологии кластеров на деревьях подтверждали с помощью rapid bootstrаp-анализа на основе генерации 500 псевдореплик [12]. Принадлежность изучаемых штаммов к филогенетическим линиям и сублиниям определяли на основе кластеризации изолятов на филогенетических деревьях с индексом поддержки узлов более 75 и высокого уровня сходства нуклеотидных последовательностей (98,5–100%).
Статистический анализ проводили с помощью общепринятых алгоритмов в программах Microsoft office (Excel), пакета статистических программ Statz, Statistica 6.0, Вiostat. Достоверным считали различие между сравниваемыми величинами с уровнем доверительной вероятности 95 и 99% при р < 0,05 и р < 0,001.
Результаты
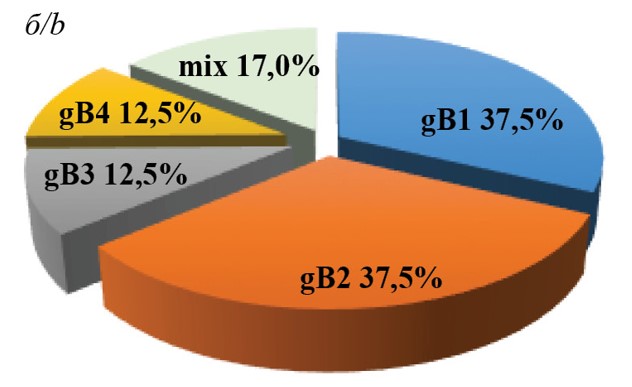
С использованием ранее предложенного нами алгоритма генотипирования были определены спектры и долевое распределение gB- и gN-генотипов ЦМВ, циркулирующего среди детей и взрослых Нижегородского региона. Результаты генотипирования, проведенного по двум генам (UL55 и UL73), показали, что штаммы ЦМВ характеризовались высоким генетическим разнообразием. Спектр идентифицированных gB-генотипов ЦМВ у детей состоял из 5 кластеров (gB1, gB2, gB3, gB4 и gB5), а у взрослых – из 4 (gB1, gB2, gB3, gB4) (рис. 1).
Рис. 1. Частота встречаемости gB-генотипов ЦМВ у детей (а) и взрослых реципиентов органов (б).
Fig. 1. Frequency of occurrence of CMV gB genotypes in children (a) and adult organ recipients (b).
Пейзаж gN-генотипов включал 4 кластера (gN4c, gN4a, gN3b, gN1) у детей и 5 кластеров у реципиентов органов (gN1, gN3a, gN3b, gN4a, gN4b, gN4c) (рис. 2).
Рис. 2. Частота встречаемости gN-генотипов ЦМВ у детей (а) и взрослых реципиентов органов (б).
Fig. 2. Frequency of occurrence of CMV gN genotypes in children (a) and adult organ recipients (b).
Сравнительны анализ распространенности gB- и gN-генотипов показал, что у детей доминировали генотипы gB1 (40,0%), gB2 (33,3%), gN4a (42,8%) и gN3b (28,6%), а у взрослых (реципиентов солидных органов) превалировали генотипы gB1 (37,5%), gB2 (37,5%), gN1 (26,3%) и gN4b (26,3%). Смешанная инфекция, обусловленная сочетанием двух и трех генотипов ЦМВ, была выявлена в 6 случаях у детей и 5 случаях у взрослых. Установлены следующие сочетания генотипов: gB4 и gB2; gB1 и gB2; gN3b и gN4a; gN1 и gN4a; gN4a, gN1 и gN3b у детей, и gB4, и gB3; gN4c, gN4b и gN3b; gN4a и gN1; gN4с и gN1; gN4c и gN4a у взрослых.
Филогенетический анализ цитомегаловируса
Для изучения филогенетических взаимоотношений штаммов выявленных ЦМВ были использованы нуклеотидные последовательности двух генов (UL55 и UL73). Филогенетический анализ позволил изучить внутригенотиповое разнообразие исследуемых штаммов на уровне линий и сублиний. Филогенетические деревья, построенные на основе нуклеотидных последовательностей генов UL55(gB) и UL73(gN) анализируемых и депонированных в базе данных GenBank/NCBI изолятов ЦМВ, выделенных у детей и взрослых реципиентов органов, с использованием метода Maximum Likelihood, представлены на рис. 3–6.
Рис. 3. Филогенетическое дерево, построенное по алгоритму Maximum Likelihood на основе анализа нуклеотидных последовательностей фрагмента (366 н.о.) гена UL55(gB) ЦМВ, обнаруженного у детей.
Fig. 3. Phylogenetic tree based on partial nucleotide sequences of CMV UL55(gB) gene (366 nt) isolated from children, built using the Maximum Likelihood algorithm.
Рис. 4. Филогенетическое дерево, построенное по алгоритму Maximum Likelihood на основе анализа нуклеотидных последовательностей фрагмента гена UL73(gN) (420 н.о.) ЦМВ, обнаруженного у детей.
Fig. 4. Phylogenetic tree based on nucleotide sequences of CMV UL73(gN) gene (420 nt) isolated from children, built using the Maximum Likelihood algorithm.
Рис. 5. Филогенетическое дерево, построенное по алгоритму Maximum Likelihood на основе анализа нуклеотидных последовательностей фрагмента гена UL55(gB) (366 н.о.) ЦМВ, выявленного у взрослых реципиентов солидных органов.
Fig. 5. Phylogenetic tree based on partial nucleotide sequences CMV UL55(gB) gene (366 nt) isolated from adults solid organ recipients, built using the Maximum Likelihood algorithm.
Рис. 6. Филогенетическое дерево, построенное по алгоритму Maximum Likelihood на основе анализа нуклеотидных последовательностей фрагмента гена UL73(gN) (420 н.о.) ЦМВ, выявленного у взрослых реципиентов солидных органов.
Fig. 6. Phylogenetic tree based on nucleotide sequences of CMV UL73(gN) gene (420 nt) isolated from adults solid organ recipients, built using the Maximum Likelihood algorithm.
Уровень сходства между отдельными кластерами gB-генотипов, рассчитанного на основании P-distans, варьировал от 76,5 до 87,2% н.о.
Кластер, объединивший российские изоляты ЦМВ генотипа gB1, выявленного у детей и взрослых, составил общую группу с изолятами ЦМВ, циркулирующего на территории Великобритании, США и Германии. Идентичность внутри группы варьировала от 97,8 до 100% н.о., а с другими кластерами – от 81,9 до 96,5% н.о.
Исследуемые штаммы ЦМВ генотипа gB2, обнаруженные у детей и взрослых реципиентов органов, вошли в общую группу с изолятами, выделенными на территории США, Мексики, Великобритании, Египта и стран Европы, уровень гомологии внутри группы составил 98,8–100% н.о. Следует отметить, что изоляты ЦМВ, выявленные у реципиентов солидных органов, заняли отдельную ветвь и отличались от изолятов ЦМВ, взятых из GenBank, при их сравнении с другими кластерами, сходство варьировало в пределах от 83,0 до 96,5% н.о.
Штаммы ЦМВ, имеющие генотип gB4, вошли в одну группу с изолятами, выделенными в США и Мексике, уровень идентичности нуклеотидных последовательностей внутри группы варьировал от 98,0 до 100,0% н.о, при этом процент схожести с другими кластерами составил 81,9–96,0% н.о.
Установлен высокий (96,0–99,4% н.о) уровень гомологии внутри группы, соответствующей генотипу gB3, сходство с другими кластерами варьировало от 82,0 до 88,7% н.о.
Особый интерес представляли обнаруженные у детей штаммы ЦМВ, принадлежащие генотипу gB5. Показан высокий уровень (98,5–99,1% н.о) их идентичности внутри группы. Сравнительный анализ нуклеотидных последовательностей гена UL55 российских и представленных в международной базе GenBank изолятов ЦМВ показал, что наиболее близкими оказались кластеры gB4 и gB1, которые образовали две отдельные группы с самым высоким процентом сходства между собой (от 93,9 до 97,5% н.о.).
Филогенетический анализ нуклеотидных последовательностей гена UL73(gN) показал, что уровень идентичности между отдельными кластерами варьировал от 74,8 до 87,7% н.о. Нижегородские штаммы ЦМВ генотипа gN4c образовали общую группу с изолятами, выявленными на территории Великобритании и Италии, уровень гомологии внутри группы составил 99,8–100% н.о. Штаммы ЦМВ генотипов gN3b и gN1 вошли в общую группу со штаммами ЦМВ, циркулирующего на территории Китая, Италии и Великобритании, уровень идентичности нуклеотидных последовательностей внутри каждой группы составил 99,8–100% н.о. Установлен высокий уровень гомологии (99,5–100% н.о.) штаммов вируса генотипа gN4a с ЦМВ, выделенным в Италии.
Определено близкое родство нуклеотидных последовательностей гена UL73 штаммов ЦМВ генотипа gN4c с нуклеотидными последовательностями вируса, выявленного на территории Италии и Великобритании. Уровень идентичности последовательностей внутри группы варьировал от 99,75 до 100% н.о. Штаммы ЦМВ генотипа gN4a, обнаруженные у реципиентов органов, так же, как и у детей, образовали общую группу с ЦМВ, циркулирующим на территории Италии. Степень их сходства внутри группы варьировал от 99,5 до 100% н.о.
Для штаммов ЦМВ генотипа gN4b, по сравнению с другими группами вируса, установлен более низкий уровень сходства, который варьировал в пределах 93,4–100% н.о. В эту группу анализируемые штаммы вошли вместе с вариантами, выделенными в Италии и Китае. Штаммы ЦМВ генотипа gN1 характеризовались высоким уровнем идентичности (99,7–100%) нуклеотидных последовательностей гена UL73 и образовали общую группу со штаммами, выявленными на территории Китая. Представители кластера gN3b объединились с вариантами ЦМВ, выявленными в Италии, Великобритании и Китае. Уровень идентичности между отдельными кластерами варьировал от 73,8 до 81,6% н.о.
Обсуждение
В настоящее время уделяется большое внимание изучению генотипового разнообразия ЦМВ в группах риска среди детей и взрослых. Сравнительный анализ распространенности различных генотипов ЦМВ у детей и взрослых пациентов, перенесших пересадку органов, в разных странах мира и России по результатам собственных исследований выявил сходство пейзажа генотипов ЦМВ, циркулирующего в России, Бразилии, Китае и США. Установленные нуклеотидные последовательности распределились по кластерам, соответствующим определенным генотипам ЦМВ, новые генотипы не были обнаружены. Уровень идентичности для каждого генотипа внутри группы был порядка 96,0–100% н.о., а между разными кластерами варьировал от 73,8 до 87,6% н.о.
Следует отметить сходство спектров gB-генотипов в исследуемых нами группах пациентов, исключение составил генотип gB5, который был выявлен у детей и отсутствовал у взрослых реципиентов органов. Генотипы gB1 и gB2 доминировали как у детей, так и у взрослых, частота встречаемости других вариантов gB-генотипов не имела существенных отличий. По данным литературы, генотип gB1 превалирует у детей с врожденной ЦМВИ в большинстве стран мира, а среди пациентов, перенесших пересадку органов, ‒ в Канаде, Польше и Бразилии [8, 12, 15, 16]. Кроме того, генотип gB5 у детей с врожденной ЦМВИ был обнаружен у взрослых реципиентов органов в США, в минорных количествах он был выявлен в Китае [17, 18].
Работы, посвященные типированию ЦМВ по гену UL73(gN), встречаются значительно реже, чем по гену UL55(gB), поскольку ген UL73 является наиболее полиморфным среди генов, кодирующих поверхностные гликопротеины ЦМВ. Спектр gN-генотипов в исследуемых нами группах был более разнообразным. Показано, что у детей доминировали генотипы gN4a и gN3b, а у взрослых реципиентов органов ‒ генотипы gN1 и gN4b. Следует отметить, что в публикациях зарубежных исследователей имеются противоречивые данные о распространенности разных gN-генотипов у детей и взрослых из групп риска. Отмечены также различия в пейзаже генотипов. Так, по данным S. Pignatelly и соавт., в Италии у детей доминировали генотипы gN4c, gN1 и gN4a, а, по данным китайских исследователей, ‒ генотипы gN1 и gN3a; генотип gN3b были обнаружен у детей в США [12, 17, 19]. Таким образом, спектр gN-генотипов российских изолятов ЦМВ идентичен спектру генотипов ЦМВ, циркулирующего в Италии и США. У пациентов – реципиентов органов, согласно данным литературы, в Китае и Сербии доминируют генотипы gN1 и gN3а [8, 20]. Генотип gN4b циркулировал только в Сербии. Следует отметить, что пейзаж gN-генотипов ЦМВ, выявленного у реципиентов солидных органов в России, близок к спектру gN-генотипов, циркулирующих в Сербии. Штаммы генотипа gN3a встречались только у реципиентов солидных органов и не доминировали в популяции ЦМВ.
Известно, что у пациентов с иммунодефицитными состояниями различного генеза часто регистрируется ЦМВИ, обусловленная двумя и более генотипами [21]. Аналогичные результаты получены в настоящем исследовании. Так, у детей смешанная ЦМВИ выявлена в 12,5% случаев, у реципиентов органов – в 22,0%. Следует отметить, что современные молекулярные технологии, такие как NGS, являются перспективными и высокоточными методами для оценки эволюционно-генетического разнообразия широкого спектра возбудителей инфекционных болезней, включая ЦМВ. Кроме того, использование высокопроизводительного секвенирования позволяет в одной реакции определить у пациента наличие нескольких штаммов вируса, в том числе и тех, которые присутствуют в минорных количествах. Проведение таких исследований необходимо при обследовании лиц из групп риска по ЦМВ, т.к. ЦМВИ, обусловленная ассоциацией нескольких генотипов ЦМВ, может протекать более тяжело, и для элиминации вируса требуется больше времени.
Проведенный анализ пейзажа gB- и gN-генотипов ЦМВ показал, что предпочтительным является способ генотипирования ЦМВ по гену UL55. Кроме того, поверхностный гликопротеин gB является основным компонентом при разработке вакцин, т.к. он является мишенью для нейтрализующих антител и цитотоксических Т-клеток. Как эпидемиологический маркер он интересен еще и тем, что участвует не только в процессе проникновения вируса в клетку, но и в распространении вируса от клетки к клетке [9].
Заключение
Результаты проведенного исследования позволили охарактеризовать генотиповую структуру территориальной (нижегородский регион) популяции ЦМВ, различающуюся в разных группах иммунокопрометированных пациентов. Установлены филогенетические связи со штаммами вируса, циркулирующего на территории Бразилии, Китая и США. Полученная информация необходима для понимания эволюционных процессов в популяции ЦМВ.
About the authors
Ol’ga E. Van’kova
Nizhny Novgorod Research Institute of Epidemiology and Microbiology named after Academician I.N. Blokhina
Author for correspondence.
Email: voe0@mail.ru
ORCID iD: 0000-0002-9838-1133
art. researcher, laboratory of metagenomics and molecular indication of pathogens
Russian Federation, 603950, Nizhny NovgorodNina F. Brusnigina
Nizhny Novgorod Research Institute of Epidemiology and Microbiology named after Academician I.N. Blokhina
Email: nfbrusnigina@yandex.ru
ORCID iD: 0000-0003-4582-5623
PhD, leading researcher, head of the laboratory of metagenomics and molecular Indication of pathogens
Russian Federation, 603950, Nizhny NovgorodNadezhda A. Novikova
Nizhny Novgorod Research Institute of Epidemiology and Microbiology named after Academician I.N. Blokhina
Email: novikova_na@mail.ru
ORCID iD: 0000-0002-3710-6648
DrSci (Biology), professor, leading researcher, head of the laboratory of molecular epidemiology of viral infections
Russian Federation, 603950, Nizhny NovgorodReferences
- Connoly S.A., Jardetzky T.S., Longnecker R. The structural basis of herpesvirus entry. Nat. Rev. Microbiol. 2021; 19(2): 110–21. https://doi.org/10.1038/s41579-020-00448-w
- Ssentongo P., Hehnly C., Birungi P., Roach M.A., Spady J., Fronterre C., et al. Congenital cytomegalovirus infection burden and epidemiologic risk factors in countries with universal screening. JAMA Netw. Open. 2021; 4(8): e2120736. https://doi.org/10.1001/jamanetworkopen.2021.20736
- Gorbachev V.V., Dmitrachenko T.I., Semenov V.M., Egorov S.K. Aspects of cytomegalovirus reactivation in critically ill patients. Zhurnal infektologii. 2022; 14(4): 61–8. https://doi.org/10.22625/2072-6732-2022-14-4-61-68 https://elibrary.ru/davphd (in Russian)
- Hayden R.T., Caliendo A.M. Persistent challenges of interassay variability in transplant viral load testing. J. Clin. Microbiol. 2020; 58(10): e00782-20. https://doi.org/10.1128/JCM.00782-20
- Mu H., Qiao W., Zou J., Zhang H. Human cytomegalovirus glycoprotein B genotypic distributions and viral load in symptomatic infants. J. Infect. Dev. Ctries. 2023; 17(12): 1806-1813. https://doi.org/10.3855/jidc.18291
- Shakhgildyan V.I. Congenital cytomegalovirus infection: current challenges and possible solutions. Neonatologiya: novosti, mneniya, obuchenie. 2020; 8(4): 61–72. https://doi.org/10.33029/2308-2402-2020-8-4-61-72 https://elibrary.ru/xsnbrk (in Russian)
- Vankova O.E., Brusnigina N.F. Molecular and phylogenetic characteristics of cytomegaloviruses isolated from children in Nizhny Novgorod. Zdorov‘e naseleniya i sreda obitaniya – ZNiSO. 2021; (4): 25–30. https://doi.org/10.35627/2219-5238/2021-337-4-25-30 https://elibrary.ru/rfedkt (in Russian)
- Vankova O.E., Brusnigina N.F. Genotyping clinical cytomegalovirus isolates in solid-organs-transplant recipients. Infektsiya i immunitet. 2022; 12(1): 59–68. https://doi.org/10.15789/2220-7619-GCC-1653 https://elibrary.ru/thvdbq (in Russian)
- Ye L., Qian Y., Yu W., Guo G., Wang H., Xue X. Functional profile of human cytomegalovirus genes and their associated diseases: a review. Front. Microbiol. 2020; 11: 2104. https://doi.org/10.3389/fmicb.2020.02104
- Marti-Carreras J., Maes P. Human cytomegalovirus genomics and transcriptomics through the lens of next-generation sequencing: revision and future challenges. Virus Genes. 2019; 55(2): 138–64. https://doi.org/10.1007/s11262-018-1627-3
- Sijmons S., Thys K., Ngwese M.M., Damme V.E., Dvorak J., Loock M.V., et al. High-throughput analysis of human cytomegalovirus genome diversity highlights the widespread occurrence of gene-disrupting mutations and pervasive recombination. J. Virol. 2015; 89(15): 7673–95. https://doi.org/10.1128/jvi.00578-15
- Dong N., Cao L., Zheng D., Su L., Lu L., Dong Z., et al. Distribution of CMV envelop glycoprotein B, H and N genotypes in infants with congenital cytomegalovirus symptomatic infection. Front. Pediatr. 2023; 11: 1112645. https://doi.org/10.3389/fped.2023.1112645
- Vankova O.E., Brusnigina N.F., Novikova N.A. NGS technology in monitoring the genetic diversity of cytomegalovirus strains. Sovremennye tekhnologii v meditsine. 2023; 15(2): 41–7. https://doi.org/10.17691/stm2023.15.2.04 https://elibrary.ru/gwepec (in Russian)
- Kumar S., Stecher G., Li M., Knyaz C., Tamura K. MEGA X: Molecular Evolutionary Genetics Analysis across computing platforms. Mol. Biol. Evol. 2018; 35(6): 1547–9. https://doi.org/10.1093/molbev/msy096
- Sarkar A., Das D., Ansari S., Chatterjee R. P., Mishra L., Basu B., et al. Genotypes of glycoprotein B gene among the Indian symptomatic neonates with congenital CMV infection. BMC Pediatr. 2019; 19(1): 291. https://doi.org/10.1186/s12887-019-1666-5
- Puhakka L., Pati S., Lappalainen M., Lonnqvist T., Niemensivu R., Lindahl P., et al. Viral shedding and distribution of cytomegalovirus glycoprotein H(UL75), glycoprotein B (UL55), and glycoprotein N(UL73) genotypes in congenital cytomegalovirus infection. J. Clin. Virol. 2020; 125: 104287. https://doi.org/10.1016/j.jcv.2020.104287
- Pati S., Pinninti S., Novak Z., Chowdhury N., Patro R., Fowler K., et al. Genotypic diversity and mixed infection in newborn disease and hearing loss in congenital cytomegalovirus infection. J. Pediatr. Infect. Dis. 2014; 32(10): 1050–4. https://doi.org/10.1097/inf.0b013e31829bb0b9
- Wu X.J., Wang Y., Zhu Z.L., Xu Y., He G.S., Han Y., et al. The correlation of cytomegalovirus gB genotype with viral DNA load and treatment time in patients with CMV infection after hematopoietic stem cell transplantation. Zhonghua Xue Ye Xue Za Zhi. 2013; 34(2): 109–12. (in Chinese)
- Pignatelli S., Lazzarotto Т., Gatto M.R. Cytomegalovirus gN genotypes distribution among congenitally infected newborns and their relationship with symptoms at birth and sequelae. Clin. Infect. Dis. 2010; 51(1): 33–41. https://doi.org/10.1086/653423
- Jankovića M., Ćupića M., Kneževića A., Vujićb D., Soldatovićc I., Zečevićb Ž., et al. Cytomegalovirus glycoprotein B and N genotypes in pediatric recipients of the hematopoietic stem cell transplant. J. Virology. 2020; 548: 168–73. https://doi.org/10.1016/j.virol.2020.07.010
- Wang H., Valencia S.M., Preifer S.P., Jensen J.D., Kowalik T.F., Permar S.R. Common polymorphisms in the glycoproteins of human cytomegalovirus and associated strain-specific immunity. Viruses. 2021; 13(6): 1106. https://doi.org/10.3390/v13061106
- Suárez N.M., Wilkie G.S., Hage E., Camiolo S., Holton M., Hughes J., et al. Human cytomegalovirus genomes sequenced directly from clinical material: variation, multiple-strain infection, recombination, and gene loss. J. Infect. Dis. 2019; 220(5): 781–91. https://doi.org/10.1093/infdis/jiz208
Supplementary files