Генетическое разнообразие белка Vif у вариантов вируса иммунодефицита человека 1-го типа (Retroviridae: Orthoretrovirinae: Lentivirus: Human immunodeficiency virus-1), циркулировавших в Московской области в 2019‒2020 гг.
- Авторы: Антонова А.А.1, Протасова Л.А.1, Ким К.В.1, Мунчак Я.М.1, Меженская Е.Н.1, Орлова-Морозова Е.А.2, Пронин А.Ю.2, Прилипов А.Г.1, Кузнецова А.И.1
-
Учреждения:
- Институт вирусологии им. Д.И. Ивановского ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
- ГБУЗ Московской области «Центр профилактики и борьбы со СПИД»
- Выпуск: Том 70, № 2 (2025)
- Страницы: 117-132
- Раздел: ОРИГИНАЛЬНЫЕ ИССЛЕДОВАНИЯ
- URL: https://virusjour.crie.ru/jour/article/view/16744
- DOI: https://doi.org/10.36233/0507-4088-281
- EDN: https://elibrary.ru/qoqqce
- ID: 16744
Цитировать
Аннотация
Введение. Белок Vif вируса иммунодефицита человека 1-го типа (ВИЧ-1) является фактором вирусной инфекционности и противодействует клеточным дезаминазам семейства APOBEC3, препятствующим репликации вируса. На основе белка Vif ведутся разработки для создания терапевтических средств. Природные замены в белке Vif могут влиять на его функциональность и ассоциироваться с ускоренным переходом ВИЧ-инфекции в стадию СПИДа. Изучение особенностей белка Vif у вариантов ВИЧ-1, циркулирующих в России, ранее не проводилось.
Цель работы: изучить генетическое разнообразие белка Vif вариантов ВИЧ-1, циркулировавших в Московской области в 2019‒2020 гг.
Материалы и методы. Проанализировано 234 образца цельной крови ВИЧ-инфицированных пациентов без опыта приема антиретровирусной терапии. Дизайн исследования включал следующие стадии: экстракция провирусной ДНК, амплификация гена vif, секвенирование продуктов амплификации, определение генетических вариантов полученных нуклеотидных последовательностей; затем проводили исследование консенсусных последовательностей белка Vif наиболее распространенных генетических вариантов ВИЧ-1, анализ консервативности и генетического разнообразия белка Vif-А6 (белок Vif вариантов ВИЧ-1 суб-субтипа А6) у пациентов с разными стадиями заболевания, оценку генетического разнобразия белка Vif-А6 в Московской области.
Результаты. Проведена оценка генетического разнообразия ВИЧ-1 в области генома, кодирующего белок Vif, – выявлено большое генетическое разнообразие, включающее в себя ВИЧ-1 чистых субтипов (A6, B и G), а также его рекомбинантных форм (CRF63_02A6). Впервые получены консенсусные последовательности белка Vif генетических вариантов ВИЧ-1 – B и CRF63_02A6, циркулирующих на территории Российской Федерации. Для наиболее распространенных в России вариантов ВИЧ-1 определены характерные замены в консенсусных последовательностях.
Заключение. Ограничением исследования является небольшая выборка последовательностей, принадлежащих генетическим вариантам B и CRF63_02A6 ВИЧ-1. Полученные результаты могут быть интересны и учтены при разработке терапевтических средств на основе белка Vif, а также при изучении вопросов патогенности ВИЧ-1 суб-субтипа А6.
Ключевые слова
Полный текст
Введение
Белок Vif вируса иммунодефицита человека 1-го типа (Retroviridae: Orthoretrovirinae: Lentivirus: Human immunodeficiency virus-1, HIV-1, ВИЧ-1) является фактором вирусной инфекционности, который специфически противодействует клеточным дезаминазам семейства APOBEC3, или A3 [1, 2]. Члены семейства APOBEC3 являются эффекторами врожденного иммунитета, противодействующими многим экзогенным вирусам, включая ВИЧ-1. При этом геном человека кодирует 7 генов A3 (A3A, B, C, D, F, G и H) [2]. A3G (APOBEC3G) является наиболее сильным ингибитором репликации ВИЧ-1 в отсутствие белка Vif [2, 3]. Благодаря своей цитидиндезаминазной активности, A3G преобразует цитидин (C) в уридин (U) в одноцепочечной некодирующей вирусной ДНК, образующейся в процессе обратной транскрипции вирусной РНК. На этапе синтеза второй, кодирующей, цепи провирусной ДНК происходит замена гуанина (G) на аденин (A), что в конечном итоге приводит к нарушению синтеза белка и прерыванию репликации ВИЧ-1 [2, 4]. Другие белки семейства APOBEC3: A3D, A3F и A3H, проявляют разную степень противовирусной активности [5, 6]. При этом белки семейства А3 могут проявлять противовирусную активность также дезаминазно-независимым способом на разных стадиях цикла репликации вируса (обратная транскрипция, интеграция, созревание) [2, 7, 8]. Vif нейтрализует белки семейства A3 через присоединение этих белков к E3-убиквитин-лигазному комплексу, включающему белки Cullin 5, Elongin C, Elongin B и CBF-β, для поли-убиквитинилирования с последующей протеасомной деградацией. Кроме того, Vif способен ингибировать транскрипцию A3G через захват транскрипционного кофактора (CBF-β), блокировать трансляцию A3G, ингибировать включение A3G во вновь образованные вирусные частицы [2].
Белок Vif является высоко основным полипептидом, состоящим из 192 аминокислот, массой около 23 кДа, синтезирующимся на поздней стадии репликации вируса [9, 10]. На протяжении многих лет с момента открытия белка Vif (1986 г.) изучаются его свойства и особенности взаимодействия с белками клетки-хозяина, однако полностью его роль так и не определена [2, 9–11]. На рис. 1 обозначены описанные функциональные мотивы белка Vif.
Рис. 1. Схематическое изображение функциональных мотивов в белке Vif.
11WQVDRMR17, 74TGERxWH80 и 171EDRW174 – сайты, картирующиеся с A3F; K22, K26, Y30, 40YRHHYE45, S52, W70 и 161PPLP164 – с A3G; F39, H48 и 60GDAK63 – с A3H; 69YxxL72 – с A3G и A3F; 90RKKR93 – сигнал, ингибирующий ядерную локализацию; 108Hx5Cx17−18Cx3−5H139(HCCH мотив) – с Cullin 5, входит в состав E3-убиквитин-лигазного комплекса; 144SLQYLA149 – с Elongin C, входит в состав E3-убиквитин-лигазного комплекса; 5WQVMIVW11, W89, T96, L106 – с CBF-β [2, 9–11].
Fig. 1. Schematic representation of functional motifs in the Vif protein.
11WQVDRMR17, 74TGERxWH80 and 171EDRW174 – sites mapping to A3F; K22, K26, Y30, 40YRHHYE45, S52, W70 and 161PPLP164 – to A3G; F39, H48 and 60GDAK63 – to A3H; 69YxxL72 – to A3G и A3F; 90RKKR93 – a signal inhibiting nuclear localisation; 108Hx5Cx17−18Cx3−5H139 (HCCH motif) – to Cullin 5, is part of the E3 ubiquitin ligase complex; 144SLQYLA149 – to Elongin C, is a member of the E3-ubiquitin ligase complex; 5WQVMIVW11, W89, T96, L106 – to CBF-β [2, 9–11].
В более ранних исследованиях показано, что натуральные вариации в белке Vif могут вызывать дефекты A3G- и A3F-нейтрализующей активности [12]. Последующие работы продемонстрировали, что между разными вариантами ВИЧ-1 присутствуют различия в функциональной активности белка Vif [13, 14]. Например, в области генома ВИЧ-1, кодирующего ген vif, суб-субтипа A6, широко распространенного на территории Российской Федерации, были обнаружены 12 нуклеотидных замен, 4 из которых являлись несинонимичными и приводили к заменам аминокислот K91Q, E134N, Q136P и I159E, по сравнению с ВИЧ-1 HXB2 [15]. При этом в другом исследовании была обнаружена ассоциация изменений Vif с ускоренным переходом заболевания в стадию СПИДа, мутация Q136P являлась одним из таких изменений наряду со вставкой одной аминокислоты в 61-м положении и заменами A62D/N/S [16].
В процессе исследований генетического разнообразия белка Vif были определены пары аминокислот и единичные замены, которые могут быть ассоциированы как со сниженным уровнем CD4-клеток, так и с высокой вирусной нагрузкой (ВН) [17, 18]. Таким образом, естественные вариации в белке Vif могут влиять на функциональность белка и течение заболевания.
Более того, в настоящее время разрабатываются молекулы, являющиеся антагонистами белка Vif, которые в дальнейшем могут стать основой для создания антиретровирусных препаратов нового класса [19–21]. В дизайн многих разрабатываемых мультиэпитопных вакцинных конструкций входят антигены, содержащиеся в белке Vif [22]. Таким образом, белок Vif является привлекательной мишенью для разработки терапевтических препаратов и вакцин, что делает исследование его генетического разнообразия еще более актуальным. Высокое генетическое разнообразие ВИЧ-1 и неравномерное распределение существующих вариантов вируса в мире определяют актуальность изучения особенностей белка Vif у локально циркулирующих вариантов вируса [14, 18, 23–25].
В России состав циркулирующих генетических вариантов ВИЧ-1 уникален: доминирующим вариантом является суб-субтип А6 (82,9%), на втором месте – субтип В (7,14%), затем быстро распространяющаяся рекомбинантная форма CRF63_02A6 (3,59%), и около 1% приходится на каждую из рекомбинантных форм CRF02_AG и CRF03_AB [26]. В ранее проведенных нами исследованиях были изучены особенности регуляторных белков Tat, Rev и вспомогательного белка Vpu ВИЧ-1 среди вирусов, принадлежащих суб-субтипу A6, который является наиболее широко распространенным генетическим вариантом на территории Российской Федерации; в процессе исследований проводили сравнение генетической вариабельности данных белков среди пациентов с разными стадиями заболевания [27–30]. При сравнении консенсусных последовательностей белка Vif у вариантов вируса суб-субтипа А6, циркулирующих в разных регионах России, были отмечены характерные особенности, которые могут быть связаны с так называемым «эффектом основателя» [31].
Цель исследования – изучение генетического разнообразия белка Vif вариантов ВИЧ-1, циркулирующих в Московской области: определение генетических вариантов, сравнение консенсусных последовательностей наиболее распространенных из них; анализ консервативности и генетического разнообразия белка Vif-А6 (белок Vif вариантов ВИЧ-1 суб-субтипа А6) у пациентов с разными стадиями заболевания, оценка генетического разнобразия белка Vif-А6 в Московской области.
Материалы и методы
В работе были использованы клинические образцы цельной крови, полученные от 234 наивных (без опыта приема антиретровирусной терапии (АРТ) в анамнезе) ВИЧ-инфицированных пациентов, проходивших лечение в ГКУЗ МО «Центр профилактики и борьбы со СПИД» (далее – Центр СПИД). Процедуру забора крови осуществляли сотрудники Центра СПИД. Забор крови производили единоразово в период с августа 2019 г. по июль 2020 г. в рамках реализации проекта CARE (https://www.careresearch.eu/, доступ на 29 июня 2024 г.). Весь полученный клинический материал использовали с информированного согласия пациентов на основании одобрения Комитета по биомедицинской этике ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России (протокол № 16 от 08.02.2019). Выявление факторов риска инфицирования проводили путем опроса пациентов при сборе эпидемиологического анамнеза. Также регистрировали следующую дополнительную информацию: пол и возраст пациента, дату забора клинического образца, стадию заболевания, показатели ВН и иммунный статус пациента (количество CD4+-клеток).
Пациенты, включенные в исследование, находились на 2, 3 и 4-й стадиях заболевания (табл. 1). Таким образом, для дальнейшего анализа были сформированы 3 группы пациентов в зависимости от их стадии заболевания.
Таблица 1. Характеристика включенных в исследование ВИЧ-инфицированных пациентов, классифицированных по стадии ВИЧ-инфекции*
Table 1. Characteristics of HIV-infected patients included in the study, classified by stage of HIV infection*
Характеристики Characteristics | 2-я стадия/стадия начальных проявлений Stage 2/stage of initial manifestations | 3-я стадия/субклиническая стадия Stage 3/subclinical stage | 4-я стадия/стадия вторичных проявлений Stage 4/stage of secondary manifestations |
Всего пациентов, абс. Total patients, abs. | 47 | 82 | 105 |
Пол, абс. | Gender, abs. | |||
мужской | male | 29 | 46 | 75 |
женский | female | 18 | 36 | 30 |
Возраст, медиана лет (диапазон) Age, median years (range) | 38 (19–62) | 38 (21–70) | 39 (24–64) |
Путь инфицирования, абс. | infection route, abs. | |||
гетеро | hetero | 25 | 58 | 64 |
ПИН | IDU | 5 | 11 | 36 |
МСМ | MSM | 16 | 10 | 4 |
нозокомиальный nosocomial | 0 | 0 | 1 |
неизвестно unknown | 1 | 3 | 0 |
CD4, кл/мкл (диапазон) CD4, cells/μL (range) | 642,96 (290–2022) | 474,10 (110–1658) | 236,78 (8–1062) |
Вирусная нагрузка, lg РНК, копий/мл (диапазон) Viral load, log10 RNA, copies/mL (range) | 4,9 (3,4–7,0) | 4,6 (3,3–6,2) | 5,1 (3,1–6,4) |
Примечание. В скобках представлены диапазоны абсолютных значений. ПИН – потребители инъекционных наркотиков; МСМ – мужчины, имеющие секс с мужчинами.
Note. Ranges of absolute values are presented in brackets. IDU – injecting drug users; MSM – men having sex with other men
В табл. 1 показаны основные демографические и клинико-эпидемиологические характеристики пациентов, включенных в исследование, в зависимости от стадии ВИЧ-инфекции, согласно клиническим рекомендациям Минздрава России.
Экстракцию провирусной ДНК в составе геномной ДНК из клеток крови (лимфоцитов) осуществляли методом высаливания [32]. На следующем этапе проводили амплификацию гена vif с помощью «гнездовой (Nested)» двухраундовой полимеразной цепной реакции (ПЦР) с последующим секвенированием продуктов амплификации. Последовательности использованных в работе праймеров и условия амплификации:
- 1-й раунд: праймеры: Vif1р (GCAGGTAAGAGAGCAAGCTGAACA) и Vif1о (GTCTCCGCTTCTTCCTGCCATAGGA), программа: 1) 95 °C – 5 мин; 2) 35 циклов: 95 °C – 30 с, 57 °C – 1 мин, 72 °C – 1 мин 20 с; 3) 72 °C – 7 мин; 4) 4 °C – ∞ (хранение);
- 2-й раунд: праймеры; Vif2р (GCTaCTCTGGAAAGGTGAAGG) и Vif2о (TACAAGGAGTCTTGGGCTGAC), программа: 1) 95 °C – 5 мин; 2) 35 циклов: 95 °C – 30 с, 53 °C – 1 мин, 72 °C – 1 мин 20 с; 3) 72 °C – 7 мин; 4) 4 °C – ∞ (хранение).
Амплифицированный фрагмент содержал 948 нуклеотидных оснований и включал ген vif. Очистку продуктов амплификации проводили с использованием коммерческого набора для очистки ПЦР-фрагментов – Clean S-Cap («Евроген», Россия) в соответствии с протоколом фирмы-производителя.
Определение нуклеотидных последовательностей осуществляли дидезокси-методом по Сэнгеру с использованием набора BigDye Terminator v3.1 Cycle Sequencing Kit (Applied Biosystems, США) и праймеров Vif2р и Vif2о согласно протоколу фирмы-производителя. Проведение оценки реакции секвенирования выполнялось автоматически в генетическом анализаторе ABI Prism 3130 Genetic Analyzer (Applied Biosystems, США).
Сборку и редактирование полученных нуклеотидных последовательностей проводили с использованием приложения SeqMan II 6.1. (DNASTAR Inc., США). Попарное и множественное выравнивание было выполнено с помощью модуля ClustalW, интегрированного в программный пакет AliView [33]. В случае, если в полученных алайментах были выявлены проблемные участки, проводили их дополнительное выравнивание «вручную».
Предварительное определение генетических вариантов ВИЧ-1 осуществляли с применением специализированных программ: COMET HIV-1 (https://comet.lih.lu/) – использует контекстноориентированное моделирование для быстрого типирования вирусов ВИЧ-1 [34]; jpHMM – определяет рекомбинантные формы вируса, используя скрытую марковскую модель [35]; RIP 3.0 (https://www.hiv.lanl.gov/content/sequence/RIP/RIP) – служит для идентификации рекомбинантных форм вируса; размер окна при анализе составил 50 н.о., что позволило детектировать рекомбинацию в последовательностях короткой длины. Для уточнения полученных результатов был проведен филогенетический анализ. Его осуществляли методом максимального правдоподобия (Maximum Likelihood, ML) с использованием программы IQ-TREE [36]. Источником эталонных последовательностей служила база данных Лос-Аламосской лаборатории США (https://www.hiv.lanl). Модель замещения нуклеотидов определяли при помощи программы jModelTest v. 2.1.7 на основании информационного критерия Акаике (Akaike information criterion, AIC) [37]. Достоверность выведенных филогений оценивали с помощью бутстрэп-теста (bootstrap) и критерия приблизительного отношения правдоподобия Шимодайры–Хасегавы (SH-aLRT) с 1000 послестартовых итераций. Кластеры с поддержкой SH-aLRT > 0,9 считали достоверно установленными. Визуализацию и графическую обработку результатов филогенетического анализа осуществляли в программе iTOL [38].
Затем были отобраны наиболее распространенные генетические варианты ВИЧ-1, для каждого из них были сформированы консенсусные аминокислотные последовательности для их дальнейшего сравнения. Для этого первоначально полученные нуклеотидные последовательности ВИЧ-1 наиболее распространенных генетических вариантов были переведены в аминокислотные последовательности с помощью онлайн-инструмента для трансляции, представленного на сайте: https://www.bioinformatics.org/sms2/translate.html. После этого с помощью инструмента Simple Consensus Maker (https://www.hiv.lanl.gov/content/sequence/CONSENSUS/SimpCon.html) на основе полученных аминокислотных последовательностей для каждого анализируемого генетического варианта ВИЧ-1 была сформирована общая консенсусная аминокислотная последовательность. Далее полученные консенсусные последовательности сравнивали между собой и относительно референсного штамма HXB2 ВИЧ-1 (GenBank Accession No: K03455) с применением программы MEGA v. 10.2.2.
На следующем этапе исследования оценивали консервативность и генетическую вариабельность белка Vif ВИЧ-1 суб-субтипа А6 у пациентов с разными стадиями заболевания. Для этого полученные аминокислотные последовательности Vif-А6 были сгруппированы в соответствии со стадией ВИЧ-инфекции пациента (3 группы), от которого они были получены. Затем для каждой группы дополнительно формировали консенсусные последовательности, как описано ранее. Таким образом, с помощью инструмента Simple Consensus Maker (https://www.hiv.lanl.gov/content/sequence/CONSENSUS/SimpCon.html) на основе полученных аминокислотных последовательностей для каждой стадии заболевания (2, 3 и 4-я) была сформирована общая консенсусная аминокислотная последовательность, характерная для каждой стадии. Затем оценивали уровень консервативности аминокислот в каждой позиции в каждой группе, между группами и по отношению к общему консенсусу для вариантов суб-субтипа А6, полученных ранее для всей группы исследуемых последовательностей (см. абзац выше). Для каждой позиции аминокислоты проводилась оценка консервативности по следующей шкале: 100, 90–99, 76–89, 51–75, ≤ 50%. Далее с применением программного модуля Nonparametric Statistics из пакета Statistica 8.0 (StatSoft Inc., США) оценивали статистически значимые различия в консервативности последовательностей белка Vif у пациентов с разными стадиями заболевания (точный двусторонний тест Фишера с коррекцией множественного теста Бонферрони, p < 0,0033).
Для оценки генетической вариабельности белка Vif ВИЧ-1 суб-субтипа А6 аминокислотные последовательности Vif-А6 также были поделены на группы в зависимости от стадии заболевания пациента (3 группы). Затем определяли аминокислотные замены (с применением программы MEGA v. 10.2.2) в каждой группе пациентов, используя в качестве референсной последовательности общую консенсусную аминокислотную последовательность, полученную для Vif ВИЧ-1 суб-субтипа A6. Сайты со статистически достоверными различиями в частоте встречаемости у пациентов с разными стадиями заболевания выявляли с с применением программного модуля Nonparametric Statistics из пакета Statistica 8.0 (StatSoft Inc., США), допустимым считали уровень значимости при использовании критерия χ2 с поправкой Бонферрони (p < 0,0005).
На завершающем этапе исследования в рамках изучения генетического разнообразия белка Vif суб-субтипа А6 в группе вариантов вируса, циркулирующих в Московской области, проводили их сравнение с референс-группой. Для формирования референсной группы из международной базы данных Лос-Аламос (www.hiv.lanl.gov, дата обращения 17 октября 2024 г.) были выгружены полногеномные нуклеотидные последовательности ВИЧ-1 суб-субтипа А6. Последовательности, полученные от одного и того же пациента, были исключены из анализа. Общее число последовательностей для дальнейшего анализа составило 166. Затем из загруженных полногеномных нуклеотидных последовательностей в программе MEGA v. 10.2.2 были вырезаны последовательности гена vif; полученные нуклеотидные последовательности гена vif переведены в аминокислотные, как описано ранее. Также на основе полученных аминокислотных последовательностей (исследуемых и загруженных) была сформирована общая консенсусная последовательность, которая использовалась в качестве референсной последовательности. Под генетическим разнообразием понимали частоту встречаемости аминокислоты, отличной от представленной в референсной последовательности, в каждой группе (последовательностей Vif-A6 из Московской области и последовательностей Vif-A6 из не Московской области). С помощью программного модуля Nonparametric Statistics из пакета Statistica 8.0 (StatSoft Inc., США) были выявлены участки со статистически значимыми различиями (p < 0,0013 при использовании критерия χ2 с поправкой Бонферрони).
Результаты
По результатам первичного анализа нуклеотидных последовательностей было установлено, что два образца (0,86%, 2/234), полученные от пациентов с 3-й стадией заболевания, 1311000563 и 1311001125, относились к уникальным рекомбинантам (URFs) ВИЧ-1 и были образованы фрагментами ВИЧ-1 генетических вариантов A6 и B. Данные последовательности были исключены из дальнейшего филогенетического анализа.
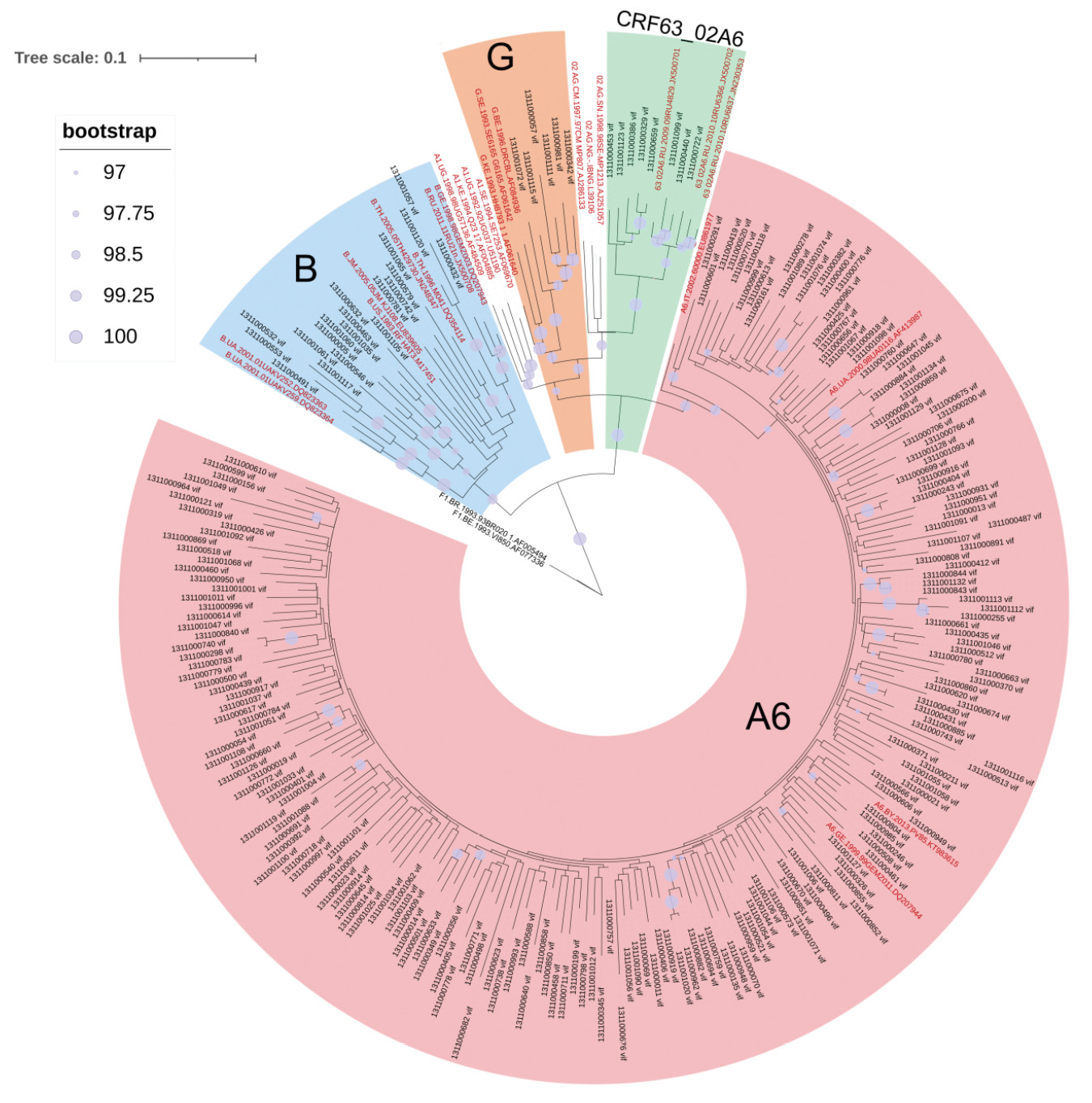
По результатам филогенетического анализа 6 (2,56%) нуклеотидных последовательностей (н.п.) были включены в кластер, образованный нуклеотидными последовательностями ВИЧ-1 субтипа G, 8 (3,42%) н.п. – циркулирующей рекомбинантной формы CRF63_02A6, 19 (8,12%) н.п. – субтипа B. Остальные 199 (85,04%) последовательностей образовали достоверный кластер с нуклеотидными последовательностями ВИЧ-1 суб-субтипа A6 (рис. 2).
Рис. 2. Филогенетический анализ нуклеотидных последовательностей гена vif ВИЧ-1 (n = 257, модель замещения нуклеотидов – GTR + I + G4). Референсные последовательности выделены красным цветом, исследуемые – черным.
Fig. 2. Phylogenetic analysis of nucleotide sequences of the HIV-1 vif gene (n = 257, nucleotide substitution model – GTR + I + G4). Reference sequences are highlighted in red, study sequences are highlighted in black.
При этом нуклеотидные последовательности, относящиеся к генетическим вариантам B и G, были получены от пациентов со всеми стадиями заболевания, тогда как последовательности, определенные как CRF63_02A6, – для пациентов с 3-й и 4-й стадиями заболевания. Также было выявлено, что две (1311001061 и 1311001117) из 234 исследуемых аминокислотных последовательностей, полученных от пациентов со 2-й стадией заболевания, имели делецию в 131-м положении аминокислотной последовательности. При этом обе последовательности относились к ВИЧ-1 субтипа B. Одна последовательность (1311001065), также B-субтипа, полученная от пациента с 3-й стадией ВИЧ-инфекции, содержала инсерцию 62insG.
Все полученные в ходе настоящего исследовании нуклеотидные последовательности гена vif ВИЧ-1 (234) были депонированы в международную базу данных генотипов GenBank со следующими номерами: PQ572780–PQ573013.
Консенсусные последовательности белка Vif были сформированы для вариантов ВИЧ-1 суб-субтипа А6, субтипа В и рекомбинантной формы CRF63_02A6 как наиболее часто встречающихся на территории Российской Федерации [26]. Общая консенсусная аминокислотная последовательность Vif суб-субтипа А6 была сформирована на основе 199 исследуемых последовательностей: субтипа В – 19, а CRF63_02A6 – 8 соответственно. Все сформированные консенсусные последовательности состояли из 192 аминокислотных остатков и не содержали ни инсерций, ни делеций. При этом в 8 аминокислотных последовательностях суб-субтипа А6, полученных от пациентов с 3-й и 4-й стадией заболевания, были обнаружены стоп-кодоны в различных позициях: наиболее часто (37,5%) – в 70-й позиции и в 25% в каждой из позиций: 21, 38 и 192.
Дополнительно проведенная оценка силы связи между исследуемыми признаками (наличие стоп-кодонов и стадия ВИЧ-инфекции) с применением критерия ϕ и V Крамера показала несущественную силу взаимосвязи.
Затем было проведено сравнение полученных консенсусных последовательностей. Результаты сравнения консенсусных последовательностей Vif ВИЧ-1 разных генетических вариантов представлены на рис. 3.
Рис. 3. Консенсусные последовательности Vif ВИЧ-1 генетических вариантов, наиболее часто встречающихся на территории России (суб-субтипа А6, субтипа В и рекомбинантной формы CRF63) выравненные относительно референс-штамма HXB2.
Точками обозначены позиции аминокислотных остатков (АК), в которых АК в консенсусах соответствовали референсу. Аминокислоты классифицированы на основе полярности радикалов. Неполярные аминокислоты: G (глицин), A (аланин), V (валин), L (лейцин), I (изолейцин), P (пролин), отмечены синим цветом; полярные незаряженные аминокислоты: S (серин), T (треонин), C (цистеин), M (метионин), N (аспарагин), Q (глутамин) – зеленым; ароматические аминокислоты: F (фенилаланин), Y (тирозин), W (триптофан), H (гистидин) – желтым; отрицательно заряженные аминокислоты: D (аспарагиновая кислота) и E (глутаминовая кислота) – оранжевым; положительно заряженные аминокислоты: K (лизин), R (аргинин) – красным [39, 40].
Fig. 3. Consensus sequences of Vif HIV-1 genetic variants most frequently encountered in Russia. The dots indicate amino acid residues (a.a.r.) positions in which the a.a.r. in the consensus corresponded to the reference.
Non-polar amino acids: G (glycine), A (alanine), V (valine), L (leucine), I (isoleucine), P (proline) – are marked in blue; polar uncharged amino acids: S (serine), T (threonine), C (cysteine), M (methionine), N (asparagine), Q (glutamine) – green; aromatic amino acids: F (phenylalanine), Y (tyrosine), W (tryptophan), H (histidine) – yellow; polar acidic, negatively charged, amino acids: D (aspartic acid) and E (glutamic acid) – orange; polar basic, positively charged amino acids: K (lysine), R (arginine) – in red [39, 40].
При сравнении консенсусных последовательностей вариантов ВИЧ-1, циркулирующих на территории Российской Федерации, были обнаружены различия у Vif ВИЧ-1 разных генетических вариантов, а также относительно референсной последовательности HXB2.
Принимая во внимание абсолютное доминирование ВИЧ-1 суб-субтипа А6 на территории России и в связи с тем, что репрезентативная выборка нуклеотидных последовательностей была получена только для суб-субтипа А6, в дальнейший анализ вошли только последовательности гена vif суб-субтипа А6 (всего 199 н.п., полученных от 199 пациентов с разной стадией заболевания): 35 н.п. – от пациентов со 2-й стадией (стадией начальных проявлений), 68 н.п. – с 3-й (субклинической стадией) и 96 – с 4-й (стадией вторичных проявлений) ВИЧ-инфекции.
При анализе консервативности белка Vif у пациентов с разными стадиями заболевания было выявлено, что консенсусные последовательности в каждой группе содержали преимущественно аминокислоты, аналогичные аминокислотам в общей консенсусной последовательности, однако с разной частотой выявления (рис. 4).
Рис. 4. Консервативность аминокислотных последовательностей белка Vif суб-субтипа А6 в группах пациентов с разными стадиями заболевания.
Аминокислоты в общем консенсусе обозначены однобуквенным кодом: А – аланин; С – цистеин; D – аспарагиновая кислота; Е – глутаминовая кислота; F – фенилаланин; G – глицин; H – гистидин; I – изолейцин; К – лизин; L – лейцин; М – метионин; Н – аспарагин; P – пролин; Q – глутамин; R – аргинин; S – серин; Т – треонин; V – валин; W – триптофан; Y – тирозин.
Fig. 4. Conservation of amino acid sequences of the Vif sub-subtype A6 protein in groups of patients with different stages of the disease.
Amino acids are designated by a general consensus with a one-letter code: A, alanine; C, cysteine; D, aspartic acid; E, glutamic acid; F, phenylalanine; G, glycine; H, histidine; I, isoleucine; K, lysine; L, leucine; M, methionine; N, asparagine; P, proline; Q, glutamine; R, arginine; S, serine; T, threonine; V, valine; W, tryptophan; Y, tyrosine.
Оценка распределения позиций с различной степенью консервативности в белке Vif в разных группах (по стадиям заболевания) пациентов не выявила статистически значимых различий (табл. 2).
Таблица 2. Распределение консервативности позиций в белке Vif в группах пациентов с разными стадиями ВИЧ-инфекции
Table 2. Distribution of conserved positions in the Vif protein in groups of patients with different stages of HIV infection
Консервативность (%) Conserved positions (%) | Число сайтов (n) | Number of sites (n) | p* | ||||
1-я стадия stage 1 | 2-я стадия stage 2 | 3-я стадия stage 2 | p2‒3 | p3‒4 | p2‒4 | |
100 | 92 | 70 | 88 | 0,0299 | 0,0778 | 0,7591 |
90–99 | 49 | 75 | 60 | 0,0062 | 0,1344 | 0,2576 |
76–89 | 34 | 28 | 28 | 0,4883 | 1,0000 | 0,4883 |
51–75 | 15 | 5 | 12 | 0,0363 | 0,1345 | 0,6905 |
≤ 50 | 2 | 14 | 4 | 0,0036 | 0,0271 | 0,6850 |
Примечание. * – указано значение p для разницы в показателе между группами (точный двусторонний тест Фишера с коррекцией множественного теста Бонферрони, p = 0,0033).
Note. * – indicates p-value for difference between groups is indicated (Fisher’s exact two-tailed test with Bonferroni multiple test correction, p = 0,0033).
При оценке вариабельности Vif у пациентов с разными стадиями ВИЧ-инфекции выявлено 35 замен со статистически значимыми различиями (табл. 3). Однако с учетом множественного сравнения, реально статистически значимыми в данном случае различия становятся с учетом поправки Бонферрони, т.е. при уровне значимости p < 0,0005.
Таблица 3. Аминокислотные замены Vif-A6 со статистически значимыми различиями по частоте встречаемости в группах ВИЧ-инфицированных пациентов с разными стадиями заболевания
Table 3. Vif-A6 amino acid substitutions with statistically significant differences in frequency of occurrence in groups of HIV-infected patients with different stages of the disease
Позиция Position | Мутация Mutation | 2-я стадия stage 2 n = 35 | 3-я стадия stage 3 n = 68 | 4-я стадия stage 4 n = 96 | p* | ||
p2–3 | p2–4 | p3–4 | |||||
24 | L24I | 2 | 0 | 3 | 0,047 | – | – |
29 | M29I | 8 | 10 | 9 | – | 0,042 | – |
31 | V31I | 7 | 28 | 21 | 0,032 | – | 0,008 |
31 | V31H | 2 | 0 | 0 | 0,047 | 0,018 | – |
33 | K33G | 2 | 0 | 1 | 0,047 | – | – |
35 | A35V | 2 | 0 | 0 | 0,047 | 0,018 | – |
41 | R41I | 2 | 1 | 0 | – | 0,018 | – |
48 | H48E | 0 | 8 | 4 | 0,035 | – | – |
50 | K50R | 23 | 31 | 43 | – | 0,034 | – |
51 | V51I | 4 | 2 | 11 | – | – | 0,047 |
60 | G60R | 3 | 0 | 3 | 0,014 | – | – |
60 | G60E | 2 | 0 | 4 | 0,047 | – | – |
61 | D61E | 3 | 18 | 12 | 0,033 | – | 0,023 |
61 | D61G | 2 | 0 | 0 | 0,047 | 0,018 | – |
63 | R63M | 2 | 0 | 2 | 0,047 | – | – |
65 | V65I | 6 | 14 | 5 | – | 0,029 | 0,002 |
77 | K77R | 2 | 16 | 10 | 0,024 | – | 0,024 |
122 | K122Q | 2 | 0 | 1 | 0,047 | – | – |
122 | K122R | 0 | 5 | 1 | – | – | 0,034 |
127 | H127Q | 17 | 20 | 22 | – | 0,004 | – |
130 | S130R | 2 | 11 | 3 | – | – | 0,003 |
131 | P131H | 1 | 3 | 0 | – | – | 0,038 |
132 | R132S | 1 | 11 | 7 | 0,046 | – | – |
136 | P136S | 6 | 18 | 13 | – | – | 0,037 |
137 | A137E | 2 | 7 | 1 | – | – | 0,007 |
141 | K141Q | 4 | 3 | 15 | – | – | 0,024 |
156 | P156L | 2 | 1 | 0 | – | 0,018 | – |
157 | T157A | 3 | 1 | 1 | – | 0,027 | – |
158 | R158K | 8 | 5 | 10 | 0,025 | – | – |
159 | E159A | 2 | 0 | 0 | 0,047 | 0,018 | – |
173 | R173K | 2 | 1 | 0 | – | 0,018 | – |
181 | R181K | 1 | 9 | 15 | – | 0,048 | – |
182 | G182D | 0 | 7 | 7 | 0,049 | – | – |
184 | R184K | 4 | 1 | 4 | 0,026 | – | – |
186 | N186H | 2 | 0 | 0 | 0,047 | 0,018 | – |
Примечание. Значения p-value представлены для всех позиций с p < 0,05; позиции с p ≥ 0,05 отмечены знаком «–». Достоверно значимыми считали различия с p с поправкой Бонферрони (p < 0,0005). * – указано значение p для разницы в показателе между группами. Жирным шрифтом отмечены замены в функционально значимых областях (рис. 1).
Note. The p-values are presented for items with p < 0.05; items with p ≥ 0.05 are marked with ‘–’. Differences with p-value with Bonferroni correction (p < 0.0005) were considered significantly significant. * – indicates p-value for difference between groups is indicated Substitutions in functionally significant areas are marked in bold (Figure 1).
Таким образом, с учетом поправки по Бонферрони, не было выявлено ни одного сайта со статистически значимыми различиями в частоте встречаемости у пациентов с разными стадиями заболевания.
Сравнение генетического разнообразия белка Vif ВИЧ-1 суб-субтипа А6 в группе вариантов вирусов, циркулирующих в Московской области, и в референсной группе выявило 39 аминокислотных замен, имеющих разницу в частоте встречаемости (p < 0,05) (табл. 4).
Таблица 4. Сравнение генетического разнообразия Vif-A6 ВИЧ-1 в группе вариантов вируса, циркулирующих в Московской области, и референсной группе
Table 4. Comparing genetic diversity of Vif-A6 HIV-1 between the group of virus variants circulating in the Moscow region and the reference group
Позиция Position | Мутация Mutation | Московская область Moscow region (n = 199) | Референсная группа Reference group (n = 166) | p |
9 | I9V | 10 | 2 | 0,042 |
17 | R17X | 1 | 9 | 0,004 |
19 | R19K | 21 | 6 | 0,012 |
20 | T20X | 1 | 18 | 0,000009 |
27 | H27Y | 19 | 6 | 0,023 |
31 | V31N | 5 | 0 | 0,04 |
31 | V31X | 7 | 16 | 0,017 |
33 | K33R | 17 | 5 | 0,027 |
41 | R41X | 2 | 10 | 0,007 |
45 | E45D | 13 | 3 | 0,028 |
51 | V51G | 0 | 4 | 0,028 |
56 | H56X | 0 | 4 | 0,028 |
61 | D61X | 2 | 7 | 0,049 |
65 | V65X | 2 | 11 | 0,004 |
67 | K67I | 9 | 1 | 0,022 |
73 | H73Q | 17 | 36 | 0,0004 |
74 | T74X | 1 | 13 | 0,0003 |
77 | K77R | 28 | 12 | 0,037 |
78 | D78A | 8 | 1 | 0,036 |
93 | R93K | 21 | 5 | 0,005 |
93 | R93M | 0 | 5 | 0,014 |
93 | R93X | 0 | 11 | 0,0002 |
98 | I98X | 5 | 12 | 0,033 |
113 | D113N | 5 | 0 | 0,04 |
128 | I128X | 0 | 5 | 0,014 |
140 | N140X | 2 | 11 | 0,004 |
141 | K141X | 0 | 5 | 0,014 |
153 | L153I | 1 | 14 | 0,0001 |
155 | T155X | 2 | 9 | 0,014 |
157 | T157A | 5 | 0 | 0,04 |
158 | R158K | 23 | 8 | 0,021 |
158 | R158X | 1 | 7 | 0,016 |
160 | R160X | 0 | 5 | 0,014 |
167 | R167X | 2 | 10 | 0,007 |
176 | K176X | 0 | 4 | 0,028 |
181 | R181X | 2 | 9 | 0,014 |
185 | G185X | 0 | 6 | 0,007 |
188 | T188S | 6 | 0 | 0,024 |
192 | C192R | 8 | 1 | 0,036 |
Примечание. X – аминокислота не определена. Значения p представлены для позиций с p < 0,05. Достоверно значимыми считали различия p с поправкой Бонферрони (p < 0,0013) – выделены жирным шрифтом.
Note. X – amino acid is not defined. p-values are given for the positions with p < 0.05. In bold: significant in the χ2 test with Bonferroni correction p < 0.0013.
С учетом поправки Бонферрони (p < 0,0013) частота встречаемости замен в 5 сайтах достоверно значимо различалась между группами.
Обсуждение
Применение современной антиретровирусной терапии для лечения ВИЧ-инфекции предотвращает клиническое прогрессирование заболевания, однако не приводит к устранению вирусных резервуаров, т.е. не позволяет элиминировать вирус, что определяет необходимость пожизненного приема антиретровирусных препаратов. Постоянный прием антиретровирусных препаратов, в свою очередь, со временем может приводить к появлению побочных эффектов лечения, формированию лекарственной устойчивости вируса и возникновению проблемы межлекарственных взаимодействий. В связи с этим в мире на постоянной основе ведется работа по созданию новых антиретровирусных препаратов, а также немалые усилия прикладывают к созданию средств полного излечения от ВИЧ-инфекции. В рамках этой работы конструируются также антогонисты белка Vif разного принципа действия: ингибиторы мультимеризации белка Vif, ингибиторы прямого связывания Vif с A3G, ингибиторы связывания Vif с Elongin C и ингибиторы связывания Vif с CBF-β [19, 41]. Кроме того, на основе белка Vif разрабатываются основы для создания терапевтических вакцин и генной терапии ВИЧ-инфекции. Так, эксперименты, проведенные in vitro, продемонстрировали, что мультиэпитопная конструкция, содержащая неструктурный белок Vif, имела высокую эффективность проникновения в клетки [42]. Разрабатываются лентивирусные векторы для генной терапии ВИЧ-инфекции, кодирующие A3G, устойчивые к действию белка Vif [43]. В настоящем исследовании впервые изучались особенности белка Vif у вариантов ВИЧ-1, циркулирующих в России.
Результаты распределения генетических вариантов ВИЧ-1 в настоящем исследовании и в ранее проведенном масштабном исследовании генетического разнообразия ВИЧ-1 на территории Российской Федерации хорошо коррелируют между собой: суб-субтип А6 – 85,04%, субтип В – 8,12%, CRF63_02A6 – 3,42% и суб-субтип А6 – 82.9%, субтип В – 7,14%, CRF63_02A6 – 3,59% соответственно. Однако в настоящем исследовании 2,56% последовательностей были генотипированы как субтип G, тогда как, по результатам более масштабного исследования, на долю субтипа G приходилось менее 1% от проанализированных образцов [26]. Такое несовпадение может быть объяснено тем, что часть последовательностей, которые в настоящем исследовании по гену vif были определены как субтип G, на самом деле могут являться циркулирующими рекомбинантными формами или уникальными рекомбинантами. Например, некоторые варианты ВИЧ-1, первоначально генотипированные по гену pol как субтип G, затем методом полногеномного секвенирования были определены как рекомбинантная форма BG [44].
По результатам проведенного исследования было установлено, что две последовательности, полученные от пациентов со 2-й стадией ВИЧ-инфекции, содержали делеции; одна последовательность, полученная от пациента с 3-й стадией ВИЧ-инфекции, содержала инсерцию (62insG). В 8 последовательностях, полученных от пациентов с 3-й (3 последовательности) и 4-й стадией (5 последовательностей) заболевания, были обнаружены стоп-кодоны в разных позициях. Чаще всего (в 37,5% аминокислотных последовательностей, содержащих стоп-кодоны) стоп-кодоны находились в 70-й позиции. В 25% последовательностей встречались стоп-кодоны в каждой из следующих позиций: 21, 38 и 192. Учитывая высокую частоту встречаемости стоп-кодонов в определенных позициях, можно сделать предположение о неслучайном механизме данного явления, которое связано с действием клеточного белка A3G [2, 4].
Сила связи наличия стоп-кодонов и стадии заболевания была несущественной. При этом в ряде исследований отмечается, что на фоне течения ВИЧ-инфекции количество дефектного провируса увеличивается, а его доля может составлять порядка 90% всего резурвуара ВИЧ [45, 46].
Однако существующие ограничения исследования, такие как относительно небольшой объем выборки и отсутствие наличия сиквенсов от одного и того же пациента в динамике, а также индивидуальные особенности течения болезни у каждого конкретного человека не дают нам возможности говорить о каких-либо тенденциях относительно стоп-кодонов в последовательностях Vif. Для белков Rev и Tat неоднократно отмечалась возможность наличия преждевременных стоп-кодонов с сохранением функциональной активности белков, тогда как для белка Vif к настоящему моменту таких данных нет, поэтому оценить функциональную значимость каждой позиции стоп-кодона так же пока не представляется возможным [47, 48].
При сравнении консенсусов циркулирующих в России вариантов ВИЧ-1 было обнаружено, что консенсусная последовательность белка Vif суб-субтипа А6 отличалась от референсной последовательности HXB2 в 30 позициях, субтипа B – в 16 позициях, рекомбинантной формы CRF63_02A6 – в 32 позициях. Наибольшее количество замен, 2 и более, консенсусы белка Vif циркулирующих в России вариантов ВИЧ-1 относительно HXB2 содержали в позициях: 47, 128, 134, 151, 159 и 167. При этом между собой консенсусы белка Vif циркулирующих в России вариантов ВИЧ-1 также отличались: от консенсуса суб-субтипа А6 консенсус субтипа В отличался в 28 из 192 позиций (на 14,6%), а консенсус рекомбинантной формы CRF63_02A6 – в 25 из 192 позиций (на 13%). Кроме того, следует отметить, что циркулирующая рекомбинантная форма CRF63_02A6 представляет собой вторичный рекомбинант, образованный ВИЧ-1 суб-субтипа А6 и CRF02_A6G, а ген vif является областью рекомбинации с включением фрагментов обоих вариантов вируса (https://www.hiv.lanl.gov/components/sequence/HIV/crfdb/crfs.comp).
Важно отметить, что консенсусы вариантов ВИЧ-1, циркулирующих в России, в 7 функционально значимых позициях отличались от референс-штамма HXB2 и содержали следующие замены: K22N, F39V, R63K, T74K, R77K, K91Q, K92E (рис. 1). Замены аргинина (R) на лизин (K) в 63-й и 77-й позиции не приводят к изменению типа аминокислоты в анализируемой позиции, т.е. аминокислота остается положительно заряженной, тогда как замены K22N, F39V, T74K, K91Q, K92E приводят к изменению типа аминокислоты (рис. 1), что может повлиять на изменение функциональных свойства белка. Так, ранее было показано, что наличие положительного заряда в функционально значимом мотиве, в который входит K22, важен для связывания и инактивации A3G [49]. Таким образом, замена лизина (K) ‒ положительно заряженной аминокислоты в 22-м положении, на аспарагин (N) ‒ полярную незаряженную аминокислоту, может привести к снижению положительного заряда, что, в свою очередь, может повлиять на связывание и инактивацию A3G.
Оценка распределения консервативности позиций в белке Vif не выявила достоверной разницы между группами пациентов с разными стадиями заболевания (табл. 3), что согласуется с результатами аналогичных исследований для белка Vpu [29].
По результатам исследования вариабельности Vif у пациентов с разными стадиями ВИЧ-инфекции не было выявлено замен со статистически значимыми различиями в частоте встречаемости (табл. 3), что отличает результаты этого исследования от результатов изучения других неструктурных белков – Tat и Vpu [28, 29]. Анализ замен в области функционально значимых областей белка Vif (табл. 3) у пациентов с разными стадиями заболевания не позволяет также выявить какую-либо тенденцию в частоте их встречаемости. В ранее проведенном исследовании генетического разнообразия белка Vif ВИЧ-1 субтипа В было показано, что у пациентов, инфицированных вариантами вируса, содержащими замену K22H, отмечалась тенденция к более низкому уровню CD4-клеток и более высокой ВН [18]. В настоящем исследовании разницы в частоте встречаемости (p < 0,05) замены K22H у пациентов с разными стадиями заболевания обнаружено не было.
Характерных замен, достоверно чаще встречающихся у вариантов ВИЧ-1 суб-субтипа А6, циркулирующих в Московской области, обнаружено не было, при этом частота встречаемости замен в 5 сайтах (T20X, H73Q, T74X, R93X, L153I) в референсной группе была достоверно выше (табл. 4). Аналогичное исследование генетического разнообразия белка Vif субтипа С в Уганде, Южной Африке и Индии позволило выявить специфичные аминокислотные замены, характерные для исследуемых географических регионов [50]. Полученные нами результаты могут быть связаны с особенностью исследуемого региона. Так, Московский столичный регион (включающий в себя Москву и Московскую область) представляет собой крупнейший транспортный узел в России, что обуславливает пересечение крупных миграционных потоков, в свою очередь приводящих к постоянной циркуляции и смене вариантов ВИЧ-1 суб-субтипа А6 из различных регионов нашей страны, а также стран бывшего Советского Союза. Все это может препятствовать формированию каких-либо выраженных особенностей циркулирующих вариантов ВИЧ-1 суб-субтипа А6 на территории Московской области [51].
Заключение
В проведенном исследовании был впервые проведен комплексный анализ особенностей белка Vif вариантов ВИЧ-1, циркулирующих в России, в Московской области. Состав генетических вариантов ВИЧ-1, циркулирующих в Московской области, определенный по гену vif, преимущественно коррелировал с составом генетических вариантов ВИЧ-1, циркулирующих на территории Российской Федерации в целом. При проведении исследования впервые получены и представлены консенсусные последовательности белка Vif ВИЧ-1 генетических вариантов B и CRF63_02A6, циркулирующих в России. Определены аминокислотные замены в функционально значимых позициях белка Vif, характерные для вариантов ВИЧ-1, наиболее распространенных в России; данные замены потенциально могут оказать влияние на изменение свойств белка и в будущем могут быть учтены при разработке терапевтических препаратов на основе белка Vif. Сравнение консервативности и генетической вариабельности белка Vif-А6 у пациентов с разными стадиями ВИЧ-инфекции не выявило достоверно значимых различий. При этом были определены позиции, содержащие делеции, инсерции и стоп-кодоны в области генома ВИЧ-1, кодирующей белок Vif. В целом генетическое разнообразие белка Vif-A6 ВИЧ-1 вариантов вируса, циркулирующих в Московской области, было меньше, чем в общей популяции вариантов ВИЧ-1 суб-субтипа А6. Проведенное исследование имеет ограничение, связанное с небольшой выборкой последовательностей Vif ВИЧ-1 генетических варинтов – В и CRF63_02A6.
Об авторах
Анастасия Александровна Антонова
Институт вирусологии им. Д.И. Ивановского ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
Email: anastaseika95@mail.ru
ORCID iD: 0000-0002-9180-9846
канд. биол. наук, научный сотрудник лаборатории вирусов лейкозов
Россия, 123098, г. МоскваЛариса Анатольевна Протасова
Институт вирусологии им. Д.И. Ивановского ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
Email: larisa.protasova.03@mail.ru
ORCID iD: 0009-0001-0430-1578
лаборант-исследователь лаборатории вирусов лейкозов
Россия, 123098, г. МоскваКристина Вячеславовна Ким
Институт вирусологии им. Д.И. Ивановского ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
Email: kimsya99@gmail.com
ORCID iD: 0000-0002-4150-2280
младший научных сотрудник лаборатории вирусов лейкозов
Россия, 123098, г. МоскваЯна Михайловна Мунчак
Институт вирусологии им. Д.И. Ивановского ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
Email: yana_munchak@mail.ru
ORCID iD: 0000-0002-4792-8928
младший научный сотрудник лаборатории вирусов лейкозов
Россия, 123098, г. МоскваЕкатерина Никитична Меженская
Институт вирусологии им. Д.И. Ивановского ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
Email: belokopytova.01@mail.ru
ORCID iD: 0000-0002-3110-0843
канд. биол. наук, научный сотрудник лаборатории вирусов лейкозов
Россия, 123098, г. МоскваЕлена Александровна Орлова-Морозова
ГБУЗ Московской области «Центр профилактики и борьбы со СПИД»
Email: orlovamorozova@gmail.com
ORCID iD: 0000-0003-2495-6501
канд. мед. наук, заведующая амбулаторно-поликлиническим отделением
Россия, 140053, Московская область, г. КотельникиАлександр Юрьевич Пронин
ГБУЗ Московской области «Центр профилактики и борьбы со СПИД»
Email: alexanderp909@gmail.com
ORCID iD: 0000-0001-9268-4929
канд. мед. наук, главный врач
Россия, 140053, Московская область, г. КотельникиАлексей Геннадьевич Прилипов
Институт вирусологии им. Д.И. Ивановского ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
Email: a_prilipov@mail.ru
ORCID iD: 0000-0001-8755-1419
д-р биол. наук, ведущий научный сотрудник, заведующий лабораторией молекулярной генетики
Россия, 123098, г. МоскваАнна Игоревна Кузнецова
Институт вирусологии им. Д.И. Ивановского ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
Автор, ответственный за переписку.
Email: a-myznikova@list.ru
ORCID iD: 0000-0001-5299-3081
заведующая лаборатории вирусов лейкозов, канд. биол. наук, ведущий научный сотрудник
Россия, 123098, г. МоскваСписок литературы
- Rose K.M., Marin M., Kozak S.L., Kabat D. The viral infectivity factor (Vif) of HIV-1 unveiled. Trends Mol. Med. 2004; 10(6): 291–7. https://doi.org/10.1016/j.molmed.2004.04.008
- Stupfler B., Verriez C., Gallois-Montbrun S., Marquet R., Paillart J.C. Degradation Independent inhibition of APOBEC3G by the HIV-1 Vif protein. Viruses. 2021; 13(4): 617. https://doi.org/10.3390/v13040617
- Sheehy A.M., Gaddis N.C., Choi J.D., Malim M.H. Isolation of a human gene that inhibits HIV-1 infection and is suppressed by the viral Vif protein. Nature. 2002; 418(6898): 646–50. https://doi.org/10.1038/nature00939
- Mangeat B., Turelli P., Caron G., Friedli M., Perrin L., Trono D. Broad antiretroviral defence by human APOBEC3G through lethal editing of nascent reverse transcripts. Nature. 2003; 424(6944): 99–103. https://doi.org/10.1038/nature01709
- Hultquist J.F., Lengyel J.A., Refsland E.W., LaRue R.S., Lackey L., Brown W.L., et al. Human and rhesus APOBEC3D, APOBEC3F, APOBEC3G, and APOBEC3H demonstrate a conserved capacity to restrict Vif-Deficient HIV-1. J. Virol. 2011; 85(21): 11220–34. https://doi.org/10.1128/JVI.05238-11
- Wang X., Abudu A., Son S., Dang Y., Venta P.J., Zheng Y.H. Analysis of human APOBEC3H haplotypes and anti-human immunodeficiency virus type 1 activity. J. Virol. 2011; 85(7): 3142–52. https://doi.org/10.1128/JVI.02049-10
- Guo F., Cen S., Niu M., Yang Y., Gorelick R.J., Kleiman L. The interaction of APOBEC3G with human immunodeficiency virus type 1 nucleocapsid inhibits tRNA3Lys annealing to viral RNA. J. Virol. 2007;81(20):11322–31. https://doi.org/10.1128/JVI.00162-07
- Xu W.K., Byun H., Dudley J.P. The role of APOBECs in viral replication. Microorganisms. 2020; 8(12): 1899. https://doi.org/10.3390/microorganisms8121899
- Azimi F.C., Lee J.E. Structural perspectives on HIV-1 Vif and APOBEC3 restriction factor interactions. Protein Sci. 2020; 29(2): 391–406. https://doi.org/10.1002/pro.3729
- Friedler A., Zakai N., Karni O., Friedler D., Gilon C., Loyter A. Identification of a nuclear transport inhibitory signal (NTIS) in the basic domain of HIV-1 Vif protein. J. Mol. Biol. 1999; 289(3): 431–7. https://doi.org/10.1006/jmbi.1999.2785
- Takaori-Kondo A., Shindo K. HIV-1 Vif: a guardian of the virus that opens up a new era in the research field of restriction factors. Front. Microbiol. 2013; 4: 34. https://doi.org/10.3389/fmicb.2013.00034
- Simon V., Zennou V., Murray D., Huang Y., Ho D.D., Bieniasz P.D. Natural variation in Vif: differential impact on APOBEC3G/3F and a potential role in HIV-1 diversification. PLoS Pathog. 2005; 1(1): e6. https://doi.org/10.1371/journal.ppat.0010006
- Iwabu Y., Kinomoto M., Tatsumi M., Fujita H., Shimura M., Tanaka Y., et al. Differential anti-APOBEC3G activity of HIV-1 Vif proteins derived from different subtypes. J. Biol. Chem. 2010; 285(46): 35350–8. https://doi.org/10.1074/jbc.M110.173286
- Ronsard L., Raja R., Panwar V., Saini S., Mohankumar K., Sridharan S., et al. Genetic and functional characterization of HIV-1 Vif on APOBEC3G degradation: First report of emergence of B/C recombinants from North India. Sci. Rep. 2015; 5: 15438. https://doi.org/10.1038/srep15438
- Громов К.Б., Лага В.Ю., Мурзакова А.В., Киреев Д.Е. Анализ полиморфизма неструктурных генов ВИЧ-1 Vif и Rev. В кн.: Молекулярная диагностика – 2017: сборник трудов IХ Всероссийской научно-практической конференции с международным участием. М.; 2017: 455–6.
- De Maio F.A., Rocco C.A., Aulicino P.C., Bologna R., Mangano A., Sen L. Effect of HIV-1 Vif variability on progression to pediatric AIDS and its association with APOBEC3G and CUL5 polymorphisms. Infect. Genet. Evol. 2011; 11(6): 1256–62. https://doi.org/10.1016/j.meegid.2011.04.020
- Bizinoto M.C., Yabe S., Leal É., Kishino H., Martins L. de O., de Lima M.L., et al. Codon pairs of the HIV-1 vif gene correlate with CD4+ T cell count. BMC Infect. Dis. 2013; 13: 173. https://doi.org/10.1186/1471-2334-13-173
- Villanova F., Barreiros M., Janini L.M., Diaz R.S., Leal É. Genetic diversity of HIV-1 gene Vif among treatment-naive Brazilians. AIDS Res. Hum. Retroviruses. 2017; 33(9): 952–9. https://doi.org/10.1089/AID.2016.0230
- Bennett R.P., Salter J.D., Smith H.C. A new class of antiretroviral enabling innate immunity by protecting APOBEC3 from HIV Vif-dependent degradation. Trends Mol. Med. 2018; 24(5): 507–20. https://doi.org/10.1016/j.molmed.2018.03.004
- Sharkey M., Sharova N., Mohammed I., Huff S.E., Kummetha I.R., Singh G., et al. HIV-1 escape from small-molecule antagonism of Vif. mBio. 2019; 10(1): e00144-19. https://doi.org/10.1128/mBio.00144-19
- Duan S., Wang S., Song Y., Gao N., Meng L., Gai Y., et al. A novel HIV-1 inhibitor that blocks viral replication and rescues APOBEC3s by interrupting Vif/CBFβ interaction. J. Biol. Chem. 2020; 295(43): 14592–605. https://doi.org/10.1074/jbc.RA120.013404
- Akbari E., Seyedinkhorasani M., Bolhassani A. Conserved multiepitope vaccine constructs: A potent HIV-1 therapeutic vaccine in clinical trials. Braz. J. Infect. Dis. 2023; 27(3): 102774. https://doi.org/10.1016/j.bjid.2023.102774
- Guerra-Palomares S.E., Hernandez-Sanchez P.G., Esparza-Perez M.A., Arguello J.R., Noyola D.E., Garcia-Sepulveda C.A. Molecular characterization of Mexican HIV-1 Vif sequences. AIDS Res. Hum. Retroviruses. 2016; 32(3): 290–5. https://doi.org/10.1089/AID.2015.0290
- Bbosa N., Kaleebu P., Ssemwanga D. HIV subtype diversity worldwide. Curr. Opin. HIV AIDS. 2019; 14(3): 153–60. https://doi.org/10.1097/COH.0000000000000534
- Williams M.E. HIV-1 Vif protein sequence variations in South African people living with HIV and their influence on Vif-APOBEC3G interaction. Eur. J. Clin. Microbiol. Infect. Dis. 2024; 43(2): 325–38. https://doi.org/10.1007/s10096-023-04728-0
- Антонова А.А., Кузнецова А.И., Ожмегова Е.Н., Лебедев А.В., Казеннова Е.В., Ким К.В. и др. Генетическое разнообразие ВИЧ-1 на современном этапе эпидемии в Российской Федерации: увеличение распространенности рекомбинантных форм. ВИЧ-инфекция и иммуносупрессии. 2023; 15(3): 61–72. https://doi.org/10.22328/2077-9828-2023-15-3-61-72 https://elibrary.ru/tpwttn
- Кузнецова А.И., Громов К.Б., Киреев Д.Е., Шлыкова А.В., Лопатухин А.Э., Казеннова Е.В. и др. Анализ особенностей белка Tat вируса иммунодефицита человека 1 типа суб-субтипа А6 (Retroviridae: Orthoretrovirinae: Lentivirus: Human immunodeficiency virus-1). Вопросы вирусологии. 2021; 66(6): 452–63. https://doi.org/10.36233/0507-4088-83 https://elibrary.ru/cmzgyc
- Kuznetsova A., Kim K., Tumanov A., Munchak I., Antonova A., Lebedev A., et al. Features of Tat protein in HIV-1 sub-subtype A6 variants circulating in the Moscow Region, Russia. Viruses. 2023; 15(11): 2212. https://doi.org/10.3390/v15112212 https://elibrary.ru/ucqyal
- Антонова А.А., Лебедев А.В., Казеннова Е.В., Ким К.В., Ожмегова Е.Н., Туманов А.С. и др. Вариабельность белка VPU ВИЧ-1 суб-субтипа А6 у пациентов c различными стадиями ВИЧ-инфекции. ВИЧ-инфекция и иммуносупрессии. 2024; 16(2): 40–50. https://doi.org/10.22328/2077-9828-2024-16-2-40-50 https://elibrary.ru/lpjxqk
- Lebedev A., Kim K., Ozhmegova E., Antonova A., Kazennova E., Tumanov A., et al. Rev protein diversity in HIV-1 group M clades. Viruses. 2024; 16(5): 759. https://doi.org/10.3390/v16050759
- Антонова А.А., Лебедев А.В., Ожмегова Е.Н., Шлыкова А.В., Лаповок И.А., Кузнецова А.И. Вариабельность неструктурных белков у вариантов ВИЧ-1 суб-субтипа А6 (Retroviridae: Orthoretrovirinae: Lentivirus: Human immunodeficiency virus-1, sub-subtype A6), циркулирующих в разных регионах Российской Федерации. Вопросы вирусологии. 2024; 69(5): 470–80. https://doi.org/10.36233/0507-4088-262 https://elibrary.ru/wbbkuq
- Miller S.A., Dykes D.D., Polesky H.F. A simple salting out procedure for extracting DNA from human nucleated cells. Nucleic. Acids. Res. 1988; 16(3): 1215. https://doi.org/10.1093/nar/16.3.1
- Larsson A. AliView: a fast and lightweight alignment viewer and editor for large datasets. Bioinformatics. 2014; 30(22): 3276–8. https://doi.org/10.1093/bioinformatics/btu531
- Struck D., Lawyer G., Ternes A.M., Schmit J.C., Bercoff D.P. COMET: adaptive context-based modeling for ultrafast HIV-1 subtype identification. Nucleic Acids Res. 2014; 42(18): e144. https://doi.org/10.1093/nar/gku739
- Schultz A.K., Bulla I., Abdou-Chekaraou M., Gordien E., Morgenstern B., Zoaulim F., et al. jpHMM: recombination analysis in viruses with circular genomes such as the hepatitis B virus. Nucleic Acids Res. 2012; 40: W193-8. https://doi.org/10.1093/nar/gks414.
- Nguyen L.T., Schmidt H.A., von Haeseler A., Minh B.Q. IQ-TREE: a fast and effective stochastic algorithm for estimating maximum-likelihood phylogenies. Mol. Biol. Evol. 2015; 32(1): 268–74. https://doi.org/10.1093/molbev/msu300
- Darriba D., Taboada G.L., Doallo R., Posada D. jModelTest 2: more models, new heuristics and parallel computing. Nat Methods. 2012; 9(8): 772. https://doi.org/10.1038/nmeth.2109.
- Letunic I., Bork P. Interactive Tree Of Life (iTOL) v5: an online tool for phylogenetic tree display and annotation. Nucleic Acids Res. 2021; 49(W1): W293–6. https://doi.org/10.1093/nar/gkab301
- Березов Т.Т., Коровкин Б.Ф. Биологическая химия. М.: Медицина; 1998.
- Lobanov M.Y., Pereyaslavets L.B., Likhachev I.V., Matkarimov B.T., Galzitskaya O.V. Is there an advantageous arrangement of aromatic residues in proteins? Statistical analysis of aromatic interactions in globular proteins. Comput. Struct. Biotechnol. J. 2021; 19: 5960–8. https://doi.org/10.1016/j.csbj.2021.10.036
- Duan S., Wang S., Song Y., Gao N., Meng L., Gai Y., et al. A novel HIV-1 inhibitor that blocks viral replication and rescues APOBEC3s by interrupting vif/CBFβ interaction. J. Biol. Chem. 2020; 295(43): 14592–605. https://doi.org/10.1074/jbc.RA120.013404
- Kardani K., Hashemi A., Bolhassani A. Comparison of HIV-1 Vif and Vpu accessory proteins for delivery of polyepitope constructs harboring Nef, Gp160 and P24 using various cell penetrating peptides. PLoS One. 2019; 14(10): e0223844. https://doi.org/10.1371/journal.pone.0223844
- Delviks-Frankenberry K.A., Ackerman D., Timberlake N.D., Hamscher M., Nikolaitchik O.A., Hu W.S., et al. Development of Lentiviral Vectors for HIV-1 Gene Therapy with Vif-Resistant APOBEC3G. Mol. Ther. Nucleic Acids. 2019; 18: 1023–38. https://doi.org/10.1016/j.omtn.2019.10.024
- Murzakova A., Kireev D., Baryshev P., Lopatukhin A., Serova E., Shemshura A., et al. Molecular epidemiology of HIV-1 subtype G in the Russian Federation. Viruses. 2019; 11(4): 348. https://doi.org/10.3390/v11040348
- Бобкова М.Р. Дефектные провирусы ВИЧ: возможное участие в патогенезе ВИЧ-инфекции. Вопросы вирусологии. 2024; 69(5): 399–414. https://doi.org/10.36233/0507-4088-261 https://elibrary.ru/pselci
- Веселова Е.И., Каминский Г.Д., Самойлова А.Г., Васильева И.А. Резервуар ВИЧ у больных ВИЧ-инфекцией. Туберкулёз и болезни лёгких. 2019; 97(5): 50–7. http://doi.org/10.21292/2075-1230-2019-97-5-50-57 https://elibrary.ru/hfadpt
- Jayaraman B., Fernandes J.D., Yang S., Smith C., Frankel A.D. Highly mutable linker regions regulate HIV-1 rev function and stability. Sci. Rep. 2019; 9(1): 5139. https://doi.org/10.1038/s41598-019-41582-7
- Li L., Dahiya S., Kortagere S., Aiamkitsumrit B., Cunningham D., Pirrone V., et al. Impact of Tat genetic variation on HIV-1 disease. Adv. Virol. 2012; 2012: 123605. https://doi.org/10.1155/2012/123605
- Chen G., He Z., Wang T., Xu R., Yu X.F. A patch of positively charged amino acids surrounding the human immunodeficiency virus type 1 Vif SLVx4Yx9Y motif influences its interaction with APOBEC3G. J. Virol. 2009; 83(17): 8674–82. https://doi.org/10.1128/JVI.00653-09
- Williams ME. HIV-1 Vif protein sequence variations in South African people living with HIV and their influence on Vif-APOBEC3G interaction. Eur. J. Clin. Microbiol. Infect. Dis. 2024; 43(2): 325–38. https://doi.org/10.1007/s10096-023-04728-0
- Савченко-Бельский В.Ю., Мальцева М.В., Маслова А.П. Проблемы и перспективы развития транспортной системы Московской агломерации. Транспортное дело России. 2022; (1): 124–7. https://doi.org/10.52375/20728689_2022_1_124 https://elibrary.ru/cctqsp
Дополнительные файлы