Первое обнаружение вирусов гриппа А субтипов H1N1 и H3N8 в Антарктическом регионе: о. Кинг-Джордж, 2023 год
- Авторы: Охлопкова О.В.1,2, Гончаров А.Е.3,4, Асланов Б.И.4, Фадеев А.В.5, Давидюк Ю.Н.6, Мошкин А.Д.2, Столбунова К.А.2, Степанюк М.А.2, Соболев И.А.2, Тюменцева М.А.1, Тюменцев А.И.1, Шестопалов А.М.2, Акимкин В.Г.1
-
Учреждения:
- ФБУН «Центральный научно-исследовательский институт эпидемиологии» Роспотребнадзора
- ФГБНУ «Федеральный исследовательский центр фундаментальной и трансляционной медицины»
- ФГБНУ «Институт экспериментальной медицины»
- ФГБОУ ВО «Северо-Западный государственный медицинский университет им. И.И. Мечникова» Минздрава России
- ФГБУ Научно-исследовательский институт гриппа им. А.А. Смородинцева» Минздрава России
- ФГАОУ ВО «Казанский (Приволжский) федеральный университет»
- Выпуск: Том 69, № 4 (2024)
- Страницы: 377-389
- Раздел: ОРИГИНАЛЬНЫЕ ИССЛЕДОВАНИЯ
- URL: https://virusjour.crie.ru/jour/article/view/16674
- DOI: https://doi.org/10.36233/0507-4088-257
- EDN: https://elibrary.ru/qujzfv
- ID: 16674
Цитировать
Аннотация
Актуальность. Вирус гриппа типа «А» (далее ‒ вирус гриппа А) относится к семейству Orthomyxoviridae и характеризуется сегментированным одноцепочечным РНК-геномом. Такая организация генома вируса обусловливает возможность реассортации, которая может приводить к появлению новых вариантов вируса. Основным естественным резервуаром большинства субтипов вируса гриппа A являются дикие водоплавающие птицы. Сезонные миграции собирают водоплавающих птиц со всех основных миграционных путей в районы гнездования около Северного и Южного полярных кругов. Это делает возможным межконтинентальное распространение субтипов вируса гриппа А. Наблюдение за развитием птичьего гриппа, особенно на территориях, ранее изолированных от человеческой деятельности, каким является Антарктический регион, имеет большое значение.
Цель исследования ‒ проведение молекулярно-генетического мониторинга и изучение филогенетических связей вариантов вируса гриппа А, циркулирующего на территории Антарктики в 2023 г.
Материалы и методы. Исследовали 84 образца биологического материала, полученного от птиц и морских млекопитающих в апреле–мае 2023 г. на прибрежных территориях Антарктики. Для 3 образцов проводили секвенирование на платформе Miseq, Illumina и филогенетический анализ полученных нуклеотидных последовательностей геномов вируса гриппа А.
Результаты. Подтверждена циркуляция птичьего гриппа в Антарктическом регионе. Выявлена гетерогенность пула циркулировавших вариантов вируса гриппа А (H3N8, H1N1). Секвенированы полноразмерные геномы вируса птичьего гриппа и размещены в открытой базе данных GISAID, что дополняет глобальную картину эволюционной изменчивости вирусов гриппа в мире (EPI_ISL_19032103, 19174530, 19174467).
Заключение. Изучение генетического разнообразия циркулирующих в полярных регионах Земли вирусов гриппа А и выявление присущих им условий возникновения новых генетических вариантов имеет важное теоретическое значение с научной точки зрения и является актуальной задачей для разработки мер по предотвращению биологических угроз.
Ключевые слова
Полный текст
Введение
Вирус гриппа типа «А» (далее ‒ вирус гриппа А) относится к семейству Orthomyxoviridae и характеризуется сегментированным одноцепочечным РНК-геномом. Такая организация генома вируса обусловливает возможность реассортации, которая может приводить к появлению новых вариантов вируса [1]. Основным естественным резервуаром ортомиксовирусов, в том числе большинства субтипов вирусов гриппа, являются колониальные морские птицы [2‒5]. Способность птиц этой группы совершать длительные миграции, наряду с их массовостью в пределах колоний, способствует трансконтинентальному распространению субтипов вируса гриппа А и расширяет возможности передачи последних разным видам животных, что в свою очередь, может способствовать появлению новых вариантов и видов вирусов вследствие рекомбинации и реассортации их геномов [1, 6‒8].
Приполярные области планеты в силу сочетания особых физико-географических и экологических условий являются точками притяжения миллионов перелетных морских птиц и млекопитающих, представляя собой активную зону межвидовых контактов [9‒13]. Циркуляция вируса гриппа А в природных биоценозах Антарктики и Арктики известна с середины 70-х гг. прошлого века. Вирус изолировали здесь от птиц и китообразных. В Антарктическом регионе вирусы птичьего гриппа были впервые выявлены в 1976 г. – 14 штаммов субтипа вируса гриппа А H1N3 были выделены из тканей легкого и печени китов семейства полосатиковых (Balaenopteridae) [14]. В арктической части Канады в 1984‒1997 гг. у 1,2% белух серологическими методами были обнаружены антитела к вирусам гриппа А [15]. Позднее, в начале 2000-х гг. в Антарктике была выявлена циркуляция вируса причьего гриппа среди птиц [16].
Одной из полярных территорий, располагающих существенным биологическим разнообразием, являются Южные Шетландские острова, расположенные к северу от Антарктического полуострова. Самым крупным из островов этого архипелага является остров Кинг-Джордж (Ватерлоо).
На острове расположен ряд полярных исследовательских станций, принадлежащих Аргентине («Карлини»), Бразилии («Команданте Феррас»), Китаю («Чанчэн – Великая Стена»), Польше («Генрик Арктовский»), России («Беллинсгаузен»), Уругваю («Генерал Артигас»), Перу («Мачу-Пикчу»), Чили («Президент Эдуардо Фрей») и Южной Корее («Король Седжон»). Кроме того, остров располагает туристическим потенциалом: на нем регулярно проводят короткие экскурсии для десятков туристов, прибывающих морским или авиационным транспортом. На территории острова обитает 12 видов птиц, в том числе поморники Лоннберга (Catharacta antarctica lonnbergi), южнополярные поморники (Catharacta maccormicki), белая ржанка (Chionis alba), капский буревестник (Daption capense), доминиканская чайка (Larus dominicanus), южный гигантский буревестник (Macronectes giganteus), качурка Вильсон (Oceanites oceanicus), чернобрюхая качурка (Fregetta tropica), пингвин Адели (Pygoscelis adeliae), антарктический пингвин (P. antarctica), пингвин папуа (P. papua) и антарктическая крачка (Sterna vittata) [17, 18]. Из видов млекопитающих наиболее многочисленными являются тюлени Уэдделла (Leptonychotes weddellii) и южные морские слоны (Mirounga leonina). Район также посещаем антарктическими морскими котиками (Arctocephalus gazella) и морскими леопардами (Hydrurga leptonyx) [19].
Таким образом, эта территория обладает сочетанием уникальных факторов, включающим обилие мест гнездования дальнеперелетных птиц, большие популяции потенциально восприимчивых диких животных – эндемиков, ротирующиеся группы туристов и персонала полярных станций.
В 2011 г. впервые была выявлена циркуляция вируса гриппа А на Антарктическом континенте. Вирус имел субтип H4N7 и был получен из биоматериала от гигантского буревестника. Также в этом году было идентифицировано два образца субтипа H6N8 от антарктического поморника и антарктического пингвина [16]. В 2013 г. лаборатория респираторных вирусов Института Освальдо Круза (Бразилия) осуществила сбор 95 образцов птичьих фекалий, которые были отобраны в колониях пингвинов на Южных Шетландских островах, недалеко от Антарктики, и протестированы методом полимеразной цепной реакции с обратной транскрипцией (ОТ-ПЦР) для обнаружения вируса птичьего гриппа. В результате были получены 4 полноразмерных генома вируса гриппа А субтипа H11N2. Биоматериалом послужили фекалии пингвинов Адели [16]. В 2015 г. Сотрудничающим центром Всемирной организации здравоохранения (ВОЗ) по исследованиям в области гриппа в Мельбурне (Австралия) были получены образцы от антарктических пингвинов, в которых удалось детектировать субтип H5N5. В 2017 г. также было получено несколько образцов субтипа H11N2 [20]. В то же время в базе данных GISAID отсутствует информация о субтипах вируса гриппа А, циркулировавших в Антарктическом регионе в период с 2018 по 2022 г. Первые сообщения в СМИ о том, что высокопатогенный птичий грипп субтипа H5N1 достиг антарктического региона, поступили в октябре 2023 г.1 Его выявили на субантарктических островах. Вирус идентифицировали на территории Южной Джорджии и на Южных Сандвичевых островах, примерно в 1600 км от материковой части Антарктиды. Также он достиг Фолклендских островов. Первыми от птичьего гриппа стали массово погибать чайки, поморники и крачки, затем болезнь распространилась на альбатросов, пингвинов и антарктических глупышей. Грипп А субтипа H5N1 был выявлен также у млекопитающих – им болели морские слоны и морские котики. Всего в базе данных GISAID за 2023 г. представлено 18 изолятов субтипа H5N1 из суммарно 21 секвенированного образца гриппа птиц из Антарктики. Субтипы гриппа А, которые были идентифицированы в 2023 г. помимо H5N1, – это H3N8 (2 образца) и H1N1 (1 образец) [21].
Таким образом, в связи с высокой изменчивостью вирусов гриппа, ВОЗ постоянно подчеркивает необходимость ведения глобального эпиднадзора для выявления генетических изменений в геноме вирусов гриппа А, особенно в отношении широко циркулирующих субтипов, каким является H3N8. Ввиду перечисленных фактов, мониторинг возбудителей птичьего гриппа на территориях Антарктики является необходимым и актуальным.
Цель работы заключалась в выявлении генетических вариантов вируса гриппа А, циркулирующих на прибрежных территориях Антарктики, и определении характеристик их генома.
Материалы и методы
Биологический материал для исследования (фекалии, выделения птиц и морских млекопитающих, всего 84 образца) был собран в ходе маршрутных работ, проведенных в пределах полуострова Файлдс острова Кинг-Джордж в апреле‒мае 2023 г. Видовой состав обследованных представителей антарктической фауны: чернобрюхая качурка (9 положительных образцов при детекции РНК гриппа А), антарктический буревестник (1 положительный образец), толстоклювый прион (3 положительных образца), полярная крачка (2 положительных образца), антарктический баклан (1 положительный образец), снежный буревестник (7 положительных образцов), пингвин папуа (3 положительных образца), доминиканская чайка (2 положительных образца), морской слон (6 положительных образцов), белая ржанка (1 положительный образец), южный гигантский буревестник (1 положительный образец), южнополярный поморник (7 положительных образцов), пингвин Адели (3 положительных образца).
Экстракцию общей РНК проводили с использованием набора для выделения РНК из клеток животных/бактерий, мазка/соскоба эпителиальных клеток, вирусов на колонках от компании ООО «Биолабмикс» (Россия). Индикацию РНК гриппа птиц осуществляли с помощью ОТ-ПЦР с детекцией результатов методом гель-электрофореза с использованием набора «БиоМастер ОТ-ПЦР–Премиум (2×)» (ООО «Биолабмикс», Россия).
Секвенирование полноразмерного генома вируса гриппа птиц выполняли с помощью наборов реагентов компании ООО «БиоЛинк» (Россия): набор для ферментативной фрагментации нуклеиновых кислот; набор реагентов для подготовки NGS-библиотек для платформы Illumina. Очистку комплементарной ДНК производили на магнитных частицах для очистки ДНК VAHTS DNA Clean Beads, (Vazyme, Китай). Геномные библиотеки секвенировали на платформе MiSeq, Illumina с использованием MiSeq Reagent Kit v3 600-cycle. Для выравнивания полученных прочтений на референсную последовательность использовали алгоритм BWA. Для получения консенсусных последовательностей использовали инструменты Samtools и Ivar.
Для составления выборки последовательностей был проведен предварительный филогенетический анализ. Для построения первичных филогенетических деревьев с помощью алгоритма IQTree2 в режиме автоматического подбора параметров были использованы 657 изолятов субтипа H1N1 и 1908 изолятов субтипа H3N8 из базы данных GISAID EpiFLU. Далее выборки были сокращены до 50 последовательностей с помощью инструмента PARNAS. Сравнительный филогенетический анализ нуклеотидных последовательностей проводили методом максимального правдоподобия (maximum likelihood) с использованием алгоритма RAxML [22] и модели нуклеотидных замен GTRGAMMA. Статистическую поддержку ветвей дерева определяли методом быстрого бутстрепа (rapid bootstrap) c 1000 репликаций. Филогенетическое дерево укореняли на среднюю точку (midpoint rooting). Реконструкцию предковых последовательностей проводили с использованием алгоритма RAxML [23]. Визуализацию и аннотацию филогенетических деревьев осуществляли с помощью инструментов Figtree и Inkscape. Анализ замен в аминокислотных последовательностях проводили с использованием программного обеспечения MEGA и SnapGene.
Результаты
Были проанализированы 84 пробы биологического материала методом ОТ-ПЦР для обнаружения РНК вируса гриппа А. Из них для полногеномного секвенирования были отобраны 46 перспективных образцов. По результатам сравнительного анализа полученных нуклеотидных последовательностей и филогенетического анализа исследованные образцы были идентифицированы как субтипы вируса гриппа А H1N1 (1 образец) и H3N8 (2 образца).
Как известно, геном вируса гриппа А кодирует 12 белков. Из них наибольший интерес для проведения сравнительного и филогенетического анализа представляют гены поверхностных антигенов гемагглютинина и нейраминидазы, а также неструктурного белка NS1, ответственного за ингибирование интерферона и характеризующегося значительным разнообразием среди штаммов гриппа птиц [24].
H1N1 – это субтип вируса гриппа А, который является одним из наиболее распространенных в мире и который был причиной пандемий 1918, 1977 и 2009 гг. [25].
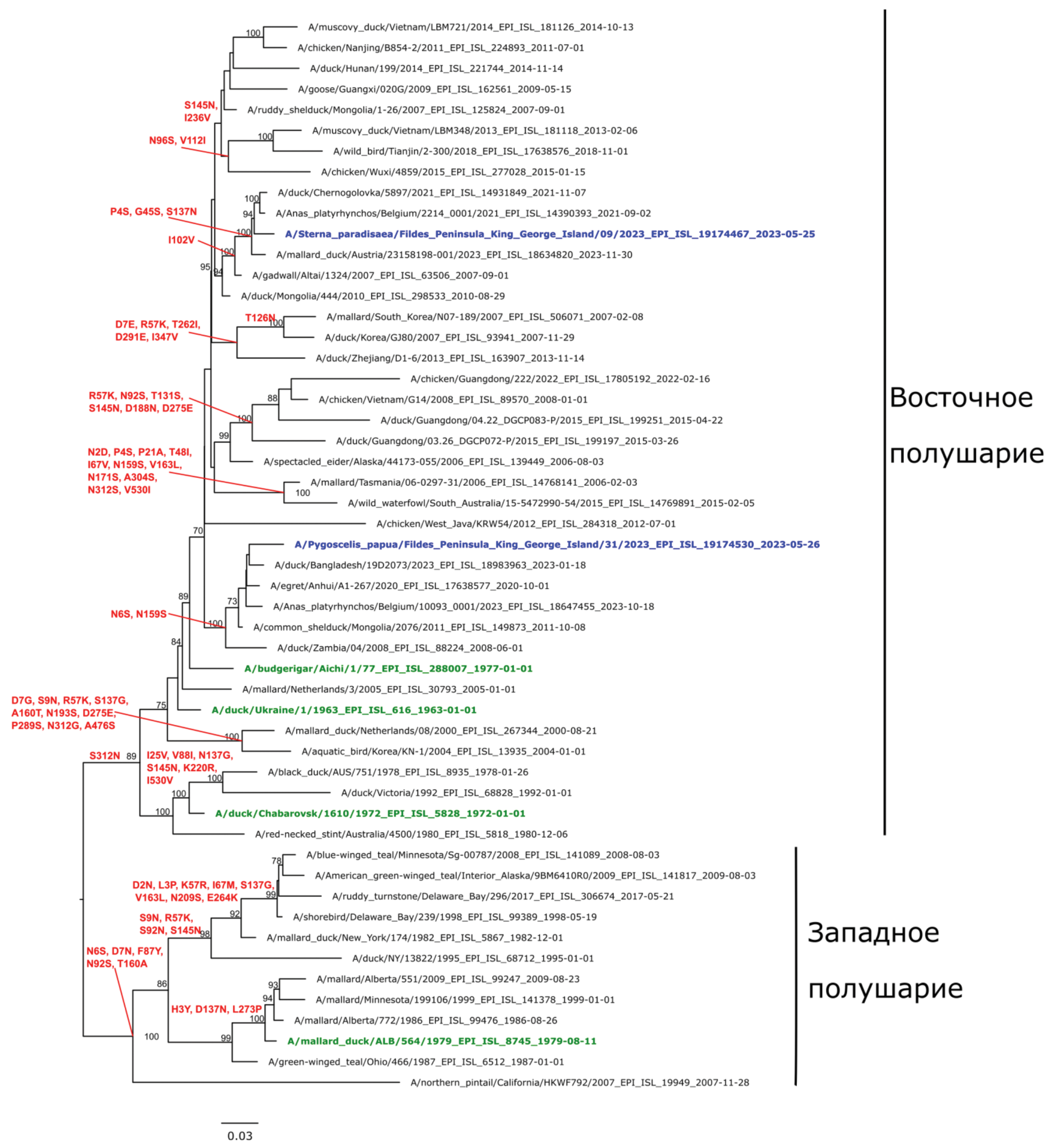
Филогенетическое окружение генома H1N1 по гену, кодирующему гемагглютинин, выявленного в результате секвенирования (рис. 1), состоит из двух географических групп: азиатской и европейской, причем в европейской можно выделить две подгруппы – это европейская часть России (Московская область), страны Европы (Бельгия, Нидерланды) и азиатская часть России (Новосибирская область), страны Азии (Корея, Китай, Бангладеш, Монголия). В результате филогенетического анализа гена гемагглютинина исследованной последовательности H1N1 была выявлена близкородственная связь с генами гемагглютинина вирусов, обнаруженных у уток на территориях Бельгии и азиатской части России, уровень подобия составил 97‒98%. При этом наиболее близкородственными оказались штаммы, циркулировавшие в основном в период 2018‒2023 гг. у диких водоплавающих птиц (рис. 1). В целом нуклеотидная последовательность гена гемагглютинина исследуемого образца является характерной для птичьих изолятов Восточного полушария.
Рис. 1. Филогенетическое дерево по гену гемагглютинина для изолятов вируса гриппа A субтипа H1N1. Здесь и на рис. 2‒5: филогенетическое дерево построено с помощью алгоритма RAxML методом максимального правдоподобия (ML) с использованием модели нуклеотидных замен GTRGAMMA. Статистическую поддержку ветвей дерева определяли методом быстрого бутстрепа (rapid bootstrap) c 1000 репликаций. Значения поддержки ниже 70 не показаны. Филогенетическое дерево укоренено на среднюю точку (midpoint rooting). Реконструкцию предковых последовательностей проводили с использованием алгоритма RAxML. Синим цветом на деревьях указаны последовательности образцов, секвенированные в ходе данного исследования. Зеленым цветом выделены старейшие штаммы для генетических групп по гемагглютинину. Красным цветом над узлами дерева обозначены замены в соответствующих реконструированных аминокислотных последовательностях.
Fig. 1. Phylogenetic tree for the hemagglutinin gene for influenza A virus subtype H1N1 isolates. Here and in Fig. 2‒5: The phylogenetic tree was constructed by the RAxML algorithm using the maximum likelihood (ML) method and the GTRGAMMA nucleotide substitution model. Statistical support for tree branches was determined by the rapid bootstrap method with 1000 replications. Support values below 70 are not shown. The phylogenetic tree is midpoint rooted. Ancestral sequences were reconstructed using the RAxML algorithm. Blue color on the trees indicates the sequences from this study. The oldest strains for genetic groups by hemagglutinin are shown in green. Substitutions in the corresponding reconstructed amino acid sequences are shown in red above the tree nodes.
На филогенетическом дереве гена нейраминидазы (рис. 2) выявленный штамм H1N1 имеет положение на дереве, близкое к изолятам, циркулировавшим на территориях таких стран, как Бельгия, Нидерланды, европейская часть России (Московская область), в период 2016‒2023 гг., а также на территориях Бельгии, Южной Кореи и Новосибирской области с 2019 по 2023 г. Причем наиболее близкими (рис. 2) оказались A/Anas_platyrhynchos/Belgium/00358_0006/2023 и A/mallard/Novosibirsk_region/3445k/2020 с различием в 3‒4% относительно исследуемого изолята H1N1.
Рис. 2. Филогенетическое дерево по гену нейраминидазы для изолятов вируса гриппа A субтипа H1N1.
Fig. 2. Phylogenetic tree for the neuraminidase gene for influenza A virus subtype H1N1 isolates.
Вирус гриппа A субтипа H3N8 относится к субтипам, наиболее часто встречающимся у птиц. Межвидовая передача субтипа H3N8 была зарегистрирована у разных видов млекопитающих, причем у собак и лошадей она носила эндемический характер. На текущий момент зарегистрировано 3 случая инфицирования человека вирусом птичьего гриппа A субтипа H3N8; все случаи были зарегистрированы в Китае в период 2022‒2023 гг., заражение, предположительно, произошло в результате прямого или косвенного контакта с инфицированной домашней птицей [26].
Филогенетически выявленные изоляты субтипа H3N8 (EPI_ISL_19174530 и EPI_ISL_ 19174467) по гену, кодирующему гемагглютинин, имеют обособленное положение относительно друг друга. Возможно, это связано с их независимой эволюцией в районе распространения. В целом филогенетическое окружение для изолята H3N8 (EPI_ISL_19174530) представлено штаммами, циркулировавшими в период 2018‒2023 гг. в Азии (рис. 3). Наиболее близкими на дереве по гену гемагглютинина являлись изоляты, полученные из Китая и Бангладеш в период 2019‒2023 гг. (рис. 3).
Рис. 3. Филогенетическое дерево по гену гемагглютинина для изолятов вируса гриппа A субтипа H3N8.
Fig. 3. Phylogenetic tree for the hemagglutinin gene for influenza A virus subtype H3N8 isolates.
Филогенетическое окружение изолята H3N8 (EPI_ISL_ 19174467) также состоит из азиатских (Китай, Казахстан, Монголия, Бангладеш) и европейских штаммов (Польша, Бельгия, Германия, Австрия) по гену гемагглютинина. Наиболее близкими изолятами являлись штаммы, которые циркулировали на территории азиатской части России в период 2018‒2021 гг. у диких водоплавающих птиц (рис. 3).
Гомология секвенированного изолята A/Pygoscelis papua/Fildes Peninsula, King George Island/31/2023 (H3N8) по гену нейраминидазы составляла от 96‒97% относительно изолятов от диких водоплавающих птиц азиатской части России A/common teal/Novosibirsk region/3556k/2020 (H3N8), Монголии A/duck/Mongolia/WKU-42/2022 (H3N8) и европейских штаммов, таких как: A/duck/Chernogolovka/5897/2021(H3N8), A/Anas_platyrhynchos/Belgium/2987_0001/2021(H3N8), A/mallard/Germany-BB/2023AI07658/2023(H3N8). Максимальное различие наблюдалось между исследуемым изолятом и штаммом A/duck/Bangladesh/19D2073/2023 (H3N8) (рис. 4).
Рис. 4. Филогенетическое дерево по гену нейраминидазы для изолятов вируса гриппа A субтипа H3N8.
Fig. 4. Phylogenetic tree for the neuraminidase gene for influenza A virus subtype H3N8 isolates.
В результате исследования обнаружено, что по гену нейраминидазы изолят A/Sterna paradisaea/Fildes Peninsula, King George Island/09/2023 (H3N8) наиболее филогенетически близок к азиатским изолятам A/mallard/Novosibirsk region/3314k/2020 (H3N8) и A/duck/Mongolia/29/2011 (H3N8). Значительное различие (5%) было зарегистрировано между нуклеотидными последовательностями исследуемого изолята и штамма A/wild duck/Germany-NW/2023AI07895/2023 (H3N8) (рис. 4). В целом филогения исследуемых изолятов из Антарктики имеет примерно схожую тенденцию относительно представленных генов – это вклад как европейских, так и азиатских штаммов в реассортацию, причем все географические точки объединены территориальной близостью друг к другу, как, например, Нидерланды, Бельгия и Германия. Также результаты анализа показали, что исследуемые изоляты имеют достаточно большую генетическую дистанцию относительно штаммов, циркулирующих в Северной Америке.
Восьмой сегмент РНК вируса гриппа А состоит из 890 нуклеотидов и кодирует 2 неструктурных белка – NS1 и NS2. В 1980 г. C. Scholtissek и V. Von Hoyningen-Huene [27] определили, что вирусы гриппа А можно разделить на 2 группы (группа 1 и более разнородная группа 2) относительно гомологии последовательности оснований их неструктурных генов (NS). Внутри данной группы гомология составляет от 94 до 100% или 75 и 100% соответственно, тогда как между двумя группами она находится в диапазоне всего 40%. В настоящее время принято определять эти группы как аллель А и аллель B, причем ассоциировать аллель B исключительно с вирусом, циркулирующим только среди птиц, а аллель А ‒ с вирусами, имеющими предрасположенность к циркуляции среди млекопитающих. Также ген NS большинства высокопатогенных вирусов птичьего гриппа H5N1, выделенных у человека с 1997 г., связан с аллелью A [28].
Образцы субтипа H3N8, выявленные в настоящем исследовании, относятся к евразийской генетической линии, по гену NS к аллели B. Причем при сравнении нуклеотидных последовательностей исследуемых изолятов со штаммом A/Goose/Guangdong/1/1996 уровень гомологии составил 92‒93%, а со штаммом A/Mallard/Sweden/2724/2006 – 67% соответственно. Результаты филогенетического анализа по гену NS изолята A/Pygoscelis papua/ Fildes Peninsula, King George Island/31/2023 (H3N8) и изолята A/Sterna paradisaea/Fildes Peninsula, King George Island/09/2023 (H3N8) показали их значительное родство (рис. 5). Филогенетическое окружение представлено в основном образцами, выявленными на территориях Бельгии, Нидерландов и европейской части России. Максимальный процент гомологии наблюдался между представленными изолятами и штаммом A/Anas_platyrhynchos/Belgium/2214_0001/2021 (H3N8). Наибольшее различие по гену NS было относительно штамма A/aquatic bird/Korea/KN-1/2004 (H3N8).
Рис. 5. Филогенетическое дерево по гену NS1 для изолятов вируса гриппа A субтипов H3N8 и H1N1.
Fig. 5. Phylogenetic tree for the NS1 gene for influenza A virus isolates of subtypes H3N8 and H1N1.
Генетический анализ гена NS исследуемого образца субтипа H1N1 показывает его соответствие евразийской линии, аллели A. При сравнении с образцом A/Mallard/Sweden/2724/2006 процент гомологии по нуклеотидным последовательностям составил 87%. Относительно A/Goose/Guangdong/1/1996 уровень гомологии составил 69%. Филогенетическое окружение представлено образцами из Кореи, Бангладеш, Китая, Монголии и азиатской части РФ. Наиболее близким (98% подобие) по гену NS к исследуемому образцу был A/Bean goose/Korea/KNU-16/2022.
Наибольшую генетическую дистанцию относительно выявленного изолята имели образцы из Аргентины, Австралии и США. Результаты филогенетического анализа по гену NS позволяют предположить, что штаммы вируса гриппа генетически довольно разнообразны и содержат свои уникальные нуклеотидные последовательности, что вероятнее всего связано с функцией этого гена в геноме как антагониста интерферона (рис. 5).
Обсуждение
Таким образом, результаты настоящего исследования продемонстрировали, что выявленные изоляты возникли, вероятнее всего, в результате реассортации между европейскими (Бельгия, Нидерланды, Россия) и азиатскими (Монголия, Китай, Бангладеш) штаммами. Значительный вклад в формирование реассортантов был обусловлен циркулирующими в период 2018‒2023 гг., на территории азиатской части России у диких водоплавающих птиц изолятами гриппа А, это может косвенно свидетельствовать о множественных заносах различных субтипов патогена с территории азиатской части России на территорию Антарктики, причем геновариантов, циркулирующих на текущий момент времени в Европе и Азии в популяциях диких птиц.
Как известно, для успешной межвидовой трансмиссии необходимо изменение свойств гемагглютинина вирусов гриппа птиц. Показано, что эти изменения связаны с аминокислотными заменами в рецептор-связывающем сайте гемагглютинина [29].
У гемагглютинина H1 вируса гриппа, как свидетельствуют данные литературы, были обнаружены следующие аминокислотные замены, связанные с межвидовой передачей вируса: S138A, E190D и G225D. Для гемагглютинина H3 межвидовая передача была связана с мутациями Q226L и G228S [30].
В этой связи нами был проведен анализ аминокислотных последовательностей на наличие таковых мутаций у идентифицированного изолята H1N1. В результате проведенного анализа были выявлены аминокислотные замены S138Y и E190T, в то же время отсутствовали изменения в положении 225, соответствующему глицину.
Для изолята A/Pygoscelis papua/Fildes Peninsula, King George Island/31/2023 (H3N8) были выявлены аминокислотные замены: пролин в положении 226, Q226P и тирозин в 228 (G228T). Соответственно для изолята A/Sterna paradisaea/Fildes Peninsula, King George Island/09/2023 (H3N8): пролин в положении 226, Q226P и гистидин в 228, G228H.
Таким образом, можно сделать вывод о накоплении в геномах антарктических изолятов несинонимичных мутаций, однако влияние соответствующих им аминокислотных замен на биологические свойства вирусов в настоящее время остается неизученным.
В целом эволюция вирусов гриппа имеет сложный и динамичный характер, ввиду особенностей организации генома и активной циркуляции среди большого числа диких водоплавающих птиц. Геномное многообразие вирусов зоонозного гриппа, которые уже вызывали случаи инфицирования человека, требует укрепления надзора за вирусами гриппа как среди людей, так и в дикой природе.
Экосистемы прибрежной Антарктики являются важнейшей географической локацией с точки зрения организации систематических мониторинговых исследований. Следует отметить что отдельные виды антарктических птиц способны к межконтинентальным миграциям (например, полярные крачки ежегодно мигрируют из Антарктики в российскую Арктику). Данное обстоятельство актуализирует вопрос о роли дальнеперелетных мигрантов в качестве векторов переноса зоонозных инфекций, включая грипп А.
Заключение
Полученные данные свидетельствуют о циркуляции на территории Антарктики вирусов гриппа А субтипов H1N1 и H3N8, с уникальными геномными характеристиками, что, в частности, проявляется спектром мутаций, ассоциированных с их потенциальной межвидовой передачей. Природная среда Антарктики, несмотря на экстремальные климатические условия, способна формировать возможные зоны образования и эволюции вирусов, в частности гриппа А. По нашему мнению, изучение генетического разнообразия циркулирующих в полярных регионах Земли вирусов гриппа А и выявление присущих им условий возникновения новых генетических вариантов и видов вирусов имеет важное теоретическое значение с научной точки зрения и является, безусловно, актуальной задачей для разработки мер по предотвращению биологических угроз.
Благодарность. Коллектив авторов выражает признательность руководству Российской антарктической экспедиции за помощь в проведении полевых исследований. Авторы признательны всем исследователям, участвовавшим на разных этапах реализации этой работы, особенно сотрудникам лаборатории молекулярной вирусологии ФГБУ НИИ гриппа им. А.А. Смородинцева за методическую поддержку, а также В.Л. Семину (Институт океанологии РАН).
Источник финансирования. Получение биологического материала от полярных крачек (Sterna paradisaea) проводилось в рамках исследований по изучению микробиоты арктических видов птиц, поддержанных грантом РНФ № 23-25-00128 «Антибиотикорезистентные бактерии, ассоциированные с орнитогенными экосистемами Арктики: генетические особенности и эпидемический потенциал».
Конфликт интересов. Авторы декларируют отсутствие явных и потенциальных конфликтов интересов, связанных с публикацией настоящей статьи.
Об авторах
Олеся Викторовна Охлопкова
ФБУН «Центральный научно-исследовательский институт эпидемиологии» Роспотребнадзора; ФГБНУ «Федеральный исследовательский центр фундаментальной и трансляционной медицины»
Автор, ответственный за переписку.
Email: ohlopkova.lesia@yandex.ru
ORCID iD: 0000-0002-8214-7828
канд. биол. наук, старший научный сотрудник лаборатории экспериментальной фармакологии отдела молекулярной диагностики и эпидемиологии, Научно-исследовательский институт вирусологии
Россия, 111123, г. Москва; 630060, г. НовосибирскАртемий Евгеньевич Гончаров
ФГБНУ «Институт экспериментальной медицины»; ФГБОУ ВО «Северо-Западный государственный медицинский университет им. И.И. Мечникова» Минздрава России
Email: phage1@yandex.ru
ORCID iD: 0000-0002-5206-6656
д-р мед. наук, заведующий лабораторией функциональной геномики и протеомики микроорганизмов; профессор кафедры эпидемиологии, паразитологии и дезинфектологии
Россия, 197022, г. Санкт-Петербург; 191015, г. Санкт-ПетербургБатырбек Исмелович Асланов
ФГБОУ ВО «Северо-Западный государственный медицинский университет им. И.И. Мечникова» Минздрава России
Email: Batyrbek.Aslanov@szgmu.ru
ORCID iD: 0000-0002-6890-8096
д-р мед. наук, профессор, заведующий кафедрой эпидемиологии, паразитологии и дезинфектологии
Россия, 191015, г. Санкт-ПетербургАртём Викторович Фадеев
ФГБУ Научно-исследовательский институт гриппа им. А.А. Смородинцева» Минздрава России
Email: artem.fadeev@influenza.spb.ru
ORCID iD: 0000-0003-3558-3261
старший научный сотрудник лаборатории молекулярной вирусологии
Россия, 197376, г. Санкт-ПетербургЮрий Николаевич Давидюк
ФГАОУ ВО «Казанский (Приволжский) федеральный университет»
Email: JNDavidjuk@kpfu.ru
ORCID iD: 0000-0002-4409-2942
канд. биол. наук, старший научный сотрудник Института фундаментальной медицины и биологии
Россия, 420008, г. КазаньАлексей Дмитриевич Мошкин
ФГБНУ «Федеральный исследовательский центр фундаментальной и трансляционной медицины»
Email: alex.moshkin727@gmail.com
ORCID iD: 0000-0002-1182-8247
младший научный сотрудник НИИ вирусологии
Россия, 630060, г. НовосибирскКристина Александровна Столбунова
ФГБНУ «Федеральный исследовательский центр фундаментальной и трансляционной медицины»
Email: kristina.sunwo@yandex.ru
ORCID iD: 0000-0003-3376-945X
младший научный сотрудник НИИ вирусологии
Россия, 630060, г. НовосибирскМарина Алексеевна Степанюк
ФГБНУ «Федеральный исследовательский центр фундаментальной и трансляционной медицины»
Email: stepanunya1996@gmail.com
ORCID iD: 0009-0002-2658-7746
младший научный сотрудник НИИ вирусологии
Россия, 630060, г. НовосибирскИван Андреевич Соболев
ФГБНУ «Федеральный исследовательский центр фундаментальной и трансляционной медицины»
Email: sobolev_i@centercem.ru
ORCID iD: 0000-0002-4561-6517
канд. биол. наук, заведующий лабораторией геномики и эволюции вирусов НИИ вирусологии
Россия, 630060, г. НовосибирскМарина Алексеевна Тюменцева
ФБУН «Центральный научно-исследовательский институт эпидемиологии» Роспотребнадзора
Email: tyumantseva@cmd.ru
ORCID iD: 0000-0002-3145-3702
канд. биол. наук, заведующий лабораторией геномного редактирования отдела молекулярной диагностики и эпидемиологии
Россия, 111123, г. МоскваАлександр Игоревич Тюменцев
ФБУН «Центральный научно-исследовательский институт эпидемиологии» Роспотребнадзора
Email: tyumantsev@cmd.ru
ORCID iD: 0000-0003-0537-2586
канд. биол. наук, заведующий лабораторией экспериментальной фармакологии отдела молекулярной диагностики и эпидемиологии
Россия, 111123, г. МоскваАлександр Михайлович Шестопалов
ФГБНУ «Федеральный исследовательский центр фундаментальной и трансляционной медицины»
Email: amshestopalov@frcftm.ru
ORCID iD: 0000-0002-9734-0620
д-р биол. наук, профессор, заслуженный деятель науки РФ, директор НИИ Вирусологии
Россия, 630060, г. НовосибирскВасилий Геннадьевич Акимкин
ФБУН «Центральный научно-исследовательский институт эпидемиологии» Роспотребнадзора
Email: akinkin@pcr.ms
ORCID iD: 0000-0003-4228-9044
д-р мед. наук, академик РАН, профессор, заслуженный врач Российской Федерации, лауреат Премии Правительства Российской Федерации в области науки и техники, директор
Россия, 111123, г. МоскваСписок литературы
- Bouvier N.M., Palese P. The biology of influenza viruses. Vaccine. 2008; 26(Suppl. 4): D49–53. https://doi.org/10.1016/j.vaccine.2008.07.039
- Fries A.C., Nolting J.M., Bowman A.S., Lin X., Halpin R.A., Wester E., et al. Spread and persistence of influenza A viruses in waterfowl hosts in the North American Mississippi migratory flyway. J. Virol. 2015; 89(10): 5371–81. https://doi.org/10.1128/JVI.03249-14
- Lvov D.K., Zhdanov V.M. Circulation of influenza virus genes in the biosphere. In: Zhdanov V.M., ed. Soviet Medical Reviews. Section E: Virology Reviews. Volume 1. Harwood Academic Publ. GmbH; 1984: 129–52.
- Lvov D.K. Circulation of Influenza Virus in Natural Biocenosis. In: Kurstak E., Maramorosh K., eds. Viruses and Enviroment. Academic Press; 1978: 351–80.
- Lvov D.K. Influenza A virus – a sum of populations with a common protected gene pool. In: Zhdanov V.M., ed. Soviet Medical Reviews. Section E: Virology Reviews. Volume 2. Harwood Academic Publ. GmbH; 1984: 15–37.
- Lvov D.K., Kaverin N.V. Avian influenza in Northern Eurasia. In: Klenk H.D., Matrosovich M.N., eds. Avian Influenza. Volume 27. Basel: Karger; 2008: 41–58.
- Львов Д.К., Ильичев В.Д., ред. Миграция птиц и перенос возбудителей инфекции. М.: Наука; 1979.
- Walker P.J., Siddell S.G., Lefkowitz E.J., Mushegian A.R., Adriaenssens E.M., Alfenas-Zerbini P., et al. Recent changes to virus taxonomy ratified by the International Committee on Taxonomy of Viruses. Arch. Virol. 2022; 167(11): 2429–40. https://doi.org/10.1007/s00705-022-05516-5
- Gass J.D. Jr., Dusek R.J., Hall J.S., Hallgrimsson G.T., Halldórsson H.P., Vignisson S.R., et al. Global dissemination of influenza A virus is driven by wild bird migration through arctic and subarctic zones. Mol. Ecol. 2023; 32(1): 198–213. https://doi.org/10.1111/mec.16738
- Wille M., Holmes E.C. The ecology and evolution of influenza viruses. Cold. Spring Harb. Perspect. Med. 2020; 10(7): a038489. https://doi.org/10.1101/cshperspect.a038489
- Cowling B.J., Ip D.K., Fang V.J., Suntarattiwong P., Olsen S.J., Levy J., et al. Aerosol transmission is an important mode of influenza A virus spread. Nat. Commun. 2013; 4: 1935. https://doi.org/10.1038/ncomms2922
- Barratclough A., Ferguson S.H., Lydersen C., Thomas P.O., Kovacs K.M. A review of circumpolar arctic marine mammal health – a call to action in a time of rapid environmental change. Pathogens. 2023; 12(7): 937. https://doi.org/10.3390/pathogens12070937
- Львов Д.К. Экология вирусов. В кн.: Львов Д.К., ред. Руководство по вирусологии. Вирусы и вирусные инфекции человека и животных. М.: МИА; 2013: 66–86.
- Lvov D.K., Zdanov V.M., Sazonov A.A., Braude N.A., Vladimirtceva E.A., Agafonova L.V., et al. Comparison of influenza viruses isolated from man and from whales. Bull World Health Organ. 1978; 56(6): 923–30.
- Nielsen O., Clavijo A., Boughen J.A. Serologic evidence of influenza A infection in marine mammals of Arctic Canada. J. Wildl. Dis. 2001; 37(4): 820–25. https://doi.org/10.7589/0090-3558-37.4.820
- Celis J.E., Espejo W., Barra R., Gonzalez-Acuña D., Gonzalez F., Jara S. Assessment of trace metals in droppings of Adélie penguins (Pygoscelis Adeliae) from different locations of the Antarctic peninsula area. Adv. Polar Sci. 2015; 26(1): 1–7. https://doi.org/10.13679/j.advps.2015.1.00001
- Hahn S., Peter H.U., Quillfeldt P., Reinhardt K. The birds of the Potter Peninsula, King George Island, South Shetland Islands, Antarctica, 1965-1998. Marine Ornithology. 1998; 26: 1–6.
- Wille M., Aban M., Wang J., Moore N., Shan S., Marshall J., et al. Antarctic penguins as reservoirs of diversity for avian avulaviruses. J. Virol. 2019; 93(11): e00271-19. https://doi.org/10.1128/JVI.00271-19
- Siniff D.B., Garrott R.A., Rotella J.J., Fraser W.R., Ainley D.G. Opinion: Projecting the effects of environmental change on Antarctic seals. Antarct. Sci. 2008; 20(5): 425–35. doi: 10.1017/S0954102008001351
- Shao W., Li X., Goraya M.U., Wang S., Chen J.L. Evolution of influenza A virus by mutation and re-assortment. Int. J. Mol. Sci. 2017; 18(8): 1650. https://doi.org/10.3390/ijms18081650
- Thorsson E., Zohari S., Roos A., Banihashem F., Bröjer C., Neimanis A. Highly pathogenic avian influenza A(H5N1) virus in a Harbor porpoise, Sweden. Emerg. Infect. Dis. 2023; 29(4): 852–5. https://doi.org/10.3201/eid2904.221426
- Markin A., Wagle S., Grover S., Vincent Baker A.L., Eulenstein O., Anderson T.K. PARNAS: objectively selecting the most representative taxa on a phylogeny. Syst. Biol. 2023; 72(5): 1052–63. https://doi.org/10.1093/sysbio/syad028
- Stamatakis A. RAxML version 8: a tool for phylogenetic analysis and post-analysis of large phylogenies. Bioinformatics. 2014; 30(9): 1312–3. https://doi.org/10.1093/bioinformatics/btu033
- Short K.R., Richard M., Verhagen J.H., van Riel D., Schrauwen E.J., van den Brand J.M., et al. One health, multiple challenges: The inter-species transmission of influenza A virus. One Health. 2015; 1: 1–13. https://doi.org/10.1016/j.onehlt.2015.03.001
- Al Hajjar S., McIntosh K. The first influenza pandemic of the 21st century. Ann. Saudi Med. 2010; 30(1): 1–10. https://doi.org/10.4103/0256-4947.59365
- Yu J., Yao Q., Liu J., Zhou Y., Huo M., Ge Y. Concern regarding H3-subtype avian influenza virus. Front. Microbiol. 2023; 14: 1327470. https://doi.org/10.3389/fmicb.2023.1327470
- Scholtissek C., von Hoyningen-Huene V. Genetic relatedness of the gene which codes for the nonstructural (NS) protein of different influenza A strains. Virology. 1980; 102(1): 13–20. https://doi.org/10.1016/0042-6822(80)90065-3
- Jahangir A., Ruenphet S., Sultana N., Shoham D., Takehara K. Genetic analysis of avian influenza viruses: cocirculation of avian influenza viruses with allele A and B nonstructural gene in Northern Pintail (Anas acuta) ducks wintering in Japan. Influenza Res. Treat. 2012; 2012: 847505. https://doi.org/10.1155/2012/847505
- Herfst S., Zhang J., Richard M., McBride R., Lexmond P., Bestebroer T.M., et al. Hemagglutinin traits determine transmission of avian A/H10N7 influenza virus between mammals. Cell Host Microbe. 2020; 28(4): 602–13.e7. https://doi.org/10.1016/j.chom.2020.08.011
- Shi Y., Wu Y., Zhang W., Qi J., Gao G.F. Enabling the ‘host jump’: structural determinants of receptor-binding specificity in influenza A viruses. Nat. Rev. Microbiol. 2014; 12(12): 822–31. https://doi.org/10.1038/nrmicro3362
Дополнительные файлы