Необычные ВА222-подобные штаммы Rotavirus A (Sedoreoviridae: Rotavirus: Rotavirus A): молекулярно-генетический анализ на основе всех сегментов генома
- Авторы: Великжанина Е.И.1, Сашина Т.А.1, Морозова О.В.1, Кашников А.Ю.1, Епифанова Н.В.1, Новикова Н.А.1
-
Учреждения:
- ФБУН «Нижегородский научно-исследовательский институт эпидемиологии и микробиологии им. академика И.Н. Блохиной» Федеральной службы по надзору в сфере защиты прав потребителей и благополучия человека (Роспотребнадзор)
- Выпуск: Том 69, № 4 (2024)
- Страницы: 363-376
- Раздел: ОРИГИНАЛЬНЫЕ ИССЛЕДОВАНИЯ
- URL: https://virusjour.crie.ru/jour/article/view/16664
- DOI: https://doi.org/10.36233/0507-4088-254
- EDN: https://elibrary.ru/ogoquq
- ID: 16664
Цитировать
Аннотация
Введение. Ротавирусная инфекция является основной причиной тяжелых дегидратирующих диарей, требующих госпитализации, у детей младшего возраста во всем мире. Благодаря сегментированному геному ротавирусы способны к реассортации генов, что делает возможным появление и распространение генетически новых штаммов.
Цель исследования ‒ поиск необычных ротавирусов, циркулировавших в Нижнем Новгороде в 2021‒2023 гг., и их молекулярно-генетическая характеристика на основе всех сегментов генома.
Материалы и методы. Ротавирус-положительные образцы стула детей исследовали методами ПЦР-генотипирования и электрофореза в полиакриламидном геле. Для отобранных штаммов были секвенированы фрагменты комплементарной ДНК каждого из 11 генов (VP1‒VP4, VP6, VP7, NSP1‒NSP5) длиной от 570 до 850 пар нуклеотидов. Филогенетический анализ проводили в программе MEGA X.
Результаты. В исследуемый период 2021‒2023 гг. было идентифицировано 11 G[P]-комбинаций с преобладанием G3P[8] (59,5%). Выявлено 6 нетипичных штаммов ротавируса А (РВА): 2 штамма генотипа G2P[4] (G2-P[4]-I2-R2-C2-M2-A3-N2-T3-E2-H3, G2-P[4]-I2-R2-C2-M2-A3-N2-T3-E3-H2) и 4 штамма G3P[9] (все штаммы имели генотип G3-P[9]-I2-R2-C2-M2-A3-N2-T3-E3-H3). Филогенетический анализ на основе всех генов показал эволюционное родство между ротавирусами, подобными ротавирусам кошек и собак (ВА222-подобными), и необычными штаммами генотипа G2P[4], для которых смешанная комбинация генотипов была выявлена и охарактеризована впервые.
Заключение. Полученные результаты расширяют представления о разнообразии реассортантных РВА, а также дополняют данные о генотиповой структуре популяции ротавирусов на территории Нижнего Новгорода. Результаты исследования в совокупности с полученными ранее данными расширяют представление о генетическом разнообразии ротавирусов и роли реассортантов в его поддержании, что важно для создания новых ротавирусных вакцин и понимания эволюционных процессов в популяции РВА.
Полный текст
Введение
В настоящее время большинство кишечных инфекций установленной этиологии вызывают вирусные патогены. Спектр кишечных вирусов весьма разнообразен, но ведущее место по заболеваемости среди детей занимает ротавирус А (РВА, Rotavirus А, род Rotavirus, семейство Sedoreoviridae, порядок Reovirales, класс Resentoviricetes, тип Duplornaviricota). Ежегодно на долю ротавирусной инфекции (РВИ) во всем мире приходится около 228 000 смертельных случаев, в том числе примерно 128 500 среди детей в возрасте до 5 лет [1, 2]. Наибольшее бремя РВИ приходится на детей, живущих в странах с низким и средним уровнем дохода, особенно из-за неблагоприятных условий жизни, ограниченного снабжения чистой питьевой водой и плохих санитарных условий [3]. В России, несмотря на ежегодно увеличивающееся число вакцинированных против РВИ детей, охват вакцинацией целевой когорты остается крайне низким, для того чтобы оказать влияние на эпидемический процесс (2020 г. – 3,68%; 2021 г. – 6,23%; 2022 г. – 7,15%; 2023 г. – 12,7%) [4, 5].
Отличительной чертой ротавирусов (РВ) является сегментированный геном, состоящий из 11 сегментов двухнитевой РНК в сумме длиной около 18 555 нуклеотидов, кодирующих 6 структурных (VP1‒VP4, VP6‒VP7) и 6 неструктурных (NSP1‒NSP6) белков [6‒8]. РВ характеризуются широким антигенным и генетическим разнообразием. Наиболее полно охарактеризованы гены VP4 и VP7, на основе которых с использованием бинарной классификации определяют G- и P-генотипы [9]. На сегодняшний день для РВА известно 42 G-генотипа и 58 P-генотипов [10]. Наиболее распространенными в мире, в том числе в России, считаются 6 комбинаций G[P]-типов РВА: G1P[8], G2P[4], G3P[8], G4P[8], G9P[8] и G12P[8], на долю которых приходится примерно 90% случаев заражения РВИ [11, 12]. Число менее распространенных G[P]-вариантов значительно больше, но они составляют всего 4,9% случаев инфекции. Например, к ним относятся такие генотипы, как G9P[4], G9P[9], G8P[8], G2P[8], G4P[4], G3P[9] и др. [13‒16].
Бинарная система классификации фокусируется только на сегментах генов VP4 и VP7 и не дает информации о разнообразии других генов. Поэтому для более глубокого изучения эпидемиологии и эволюции РВ была предложена система полногеномной классификация, позволяющая отнести каждый сегмент генома вируса к определенному генотипу. Для описания полного генотипа используют обозначение Gx-P[x]-Ix-Rx-Cx-Mx-Ax-Nx-Tx-Ex-Hx для генов, кодирующих белки VP7-VP4-VP6-VP1-VP2-VP3-NSP1-NSP2-NSP3-NSP4-NSP5/NSP6 соответственно [17]. Определение полного генотипа облегчает выявление редких и необычных РВА и обеспечивает более глубокое понимание происхождения и разнообразия штаммов, что, в свою очередь, важно для успешного прогнозирования эпидемической ситуации и оценки влияния вакцинопрофилактики на популяцию возбудителя.
Основываясь на полногеномном типировании, среди РВА выделяют 3 геногруппы. Во всем мире часто встречающиеся комбинации G1, G3, G4, G9, G12 и P[8] обычно ассоциируются с Wa-подобной (1-й) геногруппой (G1/G3/G4/G9-P[8]-I1-R1-C1-M1-A1-N1-T1-E1-H1), имеющей отдаленное родство с РВ свиней [18, 19]. Такие РВ характеризуются «длинным» профилем миграции сегментов генома в полиакриламидном геле (ПААГ). Штаммы генотипа G2P[4], в свою очередь, относятся к DS-1-подобной (2-й) геногруппе (G2-P[4]-I2-R2-C2-M2-A2-N2-T2-E2-H2), эволюционно связанной с РВ крупного рогатого скота [17, 19]. Они имеют «короткий» профиль миграции геномных сегментов. Существует 3-я, минорная, AU-1-подобная геногруппа (G3P[9]-I3-R3-C3-M3-A3-N3-T3-E3-H3), которая включает штаммы, подобные РВ кошек и собак, характеризующиеся «широким» профилем миграции сегментов [20, 21]. Штаммы с чистым AU-1-подобным сочетанием редко встречаются у людей, в то время как более распространены варианты со смешанным набором AU-1-подобных и DS-1-подобных генов. Их принято объединять в отдельную группу, прототипным для которой является штамм РВА ВА222 (G3-P[9]-I2-R2-C2-M2-A3-N1-T3-E2-H3), выделенный от домашнего кота в Италии в 2005 г. [22, 23].
Наличие сегментированного генома обусловливает способность РВ к обмену сегментами (реассортации) при одновременном инфицировании клетки двумя или более штаммами, что позволяет генам с разной специфичностью группироваться независимо друг от друга, тем самым поддерживая разнообразие в популяции. В мире все чаще отмечают распространение эпидемически значимых реассортантных штаммов РВА, которые несут генетический материал нескольких геногрупп [9, 24, 25].
В связи с этим, целью настоящей работы явились поиск необычных РВ на территории Нижнего Новгорода и их молекулярно-генетическая характеристика на основе всех сегментов генома.
Материалы и методы
В работе использовали образцы стула детей, госпитализированных в детский инфекционный стационар Нижнего Новгорода с симптомами острой кишечной инфекции (ОКИ) в период 2021‒2023 гг.
Экстракцию нуклеиновых кислот и постановку полимеразной цепной реакции (ПЦР) с обратной транскрипцией (ОТ-ПЦР) выполняли с помощью наборов реагентов РИБО-преп и РЕВЕРТА-L (Центральный научно-исследовательский институт эпидемиологии Роспотребнадзора (ЦНИИЭ), Россия). Обнаружение ротавирусной РНК проводили с помощью ПЦР-тест-систем с детекцией результата в реальном времени «АмплиСенс Rotavirus/Norovirus/Astrovirus FL» и «АмплиСенс ОКИ виро-скрин-FL» (ЦНИИЭ). Дополнительно РНК вирусов исследовали методом электрофореза в полиакриламидном геле (РНК-ПААГ) [26].
G[Р]-генотип РВ определяли методом мультиплексной ПЦР на основе праймеров, специфичных в отношении генотипов G1‒G4, G6, G8, G9, G12, P[4], P[6], P[8], P[9] [26‒32]. Детекцию результатов осуществляли с помощью электрофореза в агарозном геле, содержащем бромид этидия.
Для каждого гена был получен один фрагмент комплементарной ДНК (кДНК) длиной от 570 до 850 пар нуклеотидов с помощью праймеров, опубликованных ранее [32]. Фрагменты кДНК секвенировали по двум цепям с помощью прямого и обратного праймеров на приборе «Нанофор 05» (Институт аналитического приборостроения (ИАП) РАН, Россия) с применением набора для секвенирования BigDye Terminator v3.1 (Thermo Fisher Scientific, США). Нуклеотидные последовательности доступны в базе данных GenBank под номерами PP475712‒PP475777.
Поиск родственных последовательностей выполняли с использованием онлайн-сервиса BLAST. Для проведения филогенетического анализа из GenBank были взяты нуклеотидные последовательности 11 генов РВ, циркулировавших в разных странах, в том числе в России (Новосибирск, Омск, Москва), а также последовательности РВ из Нижнего Новгорода, полученные ранее. Регистрационные номера GenBank этих штаммов приведены в названиях изолятов на филогенетических деревьях. Выравнивание и анализ нуклеотидных последовательностей проводили в программе MEGA X версия 10.0.5. Филогенетические деревья строили методом максимального правдоподобия (Maximum Likelihood) [33, 34]. Bootstrap-анализ осуществляли на основе 1000 случайных выборок. Проценты сходства нуклеотидных последовательностей были рассчитаны методом попарных дистанций (Pairwise Distances). Принадлежность изучаемых штаммов к филогенетическим линиям и сублиниям определяли на основе кластеризации изолятов на филогенетических деревьях с индексом поддержки узлов более 75 и высокого уровня сходства нуклеотидных последовательностей (98,5‒100,0% для разных генов). Филогенетические линии и сублинии обозначали согласно принятой в литературе классификации [32, 35‒40].
Исследование проводилось при добровольном информированном согласии пациентов.
Протокол исследования одобрен Локальным этическим комитетом ФБУН «Нижегородский НИИ эпидемиологии и микробиологии им. академика И.Н. Блохиной» Федеральной службы по надзору в сфере защиты прав потребителей и благополучия человека (Роспотребнадзор) (Протокол № 6 от 24.03.2021).
Результаты
Характеристика спектра G[P]-генотипов РВА в Нижнем Новгороде
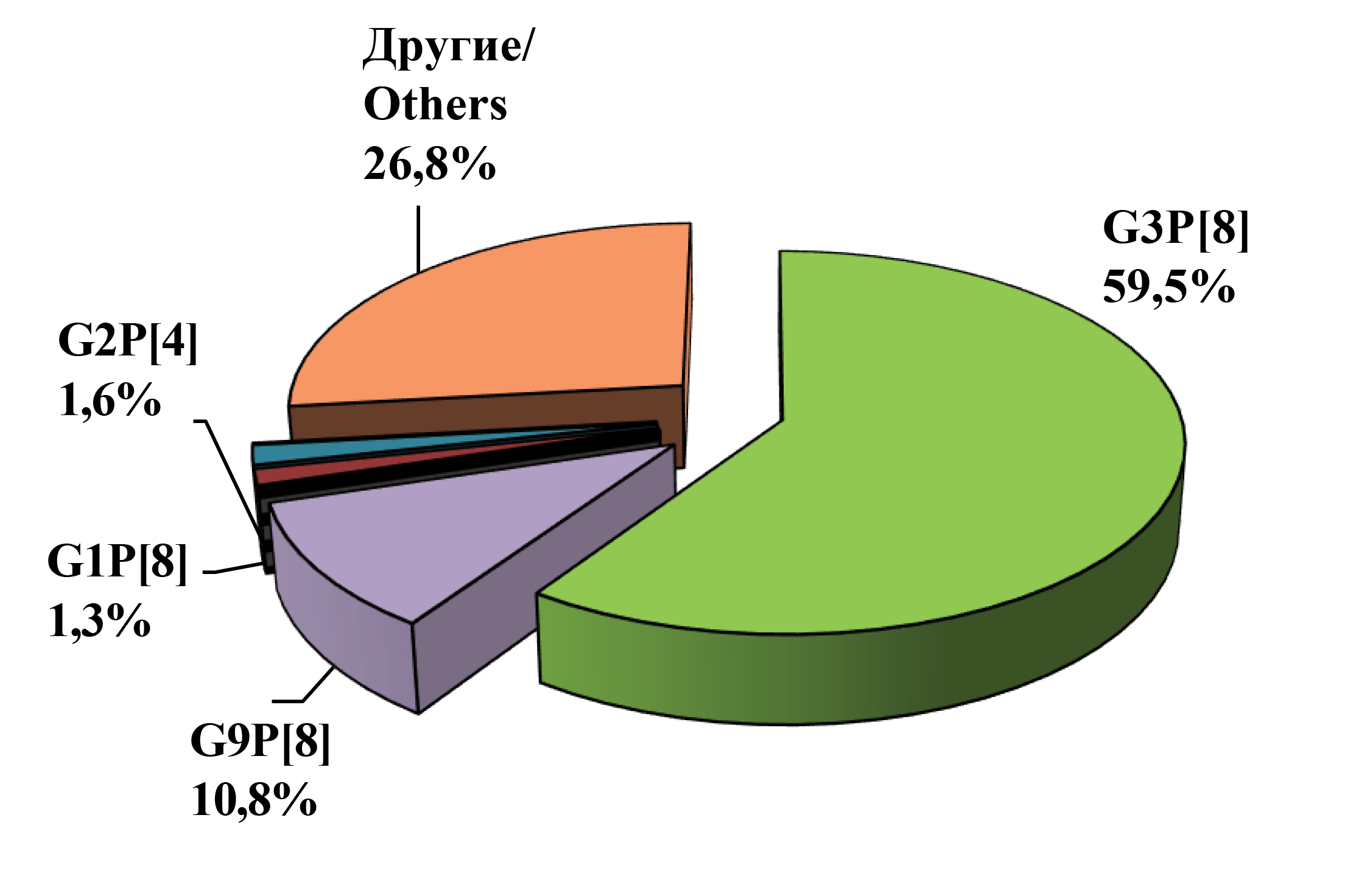
На наличие ротавирусной РНК было исследовано 3715 образцов стула детей, госпитализированных в инфекционный стационар с симптомами острого гастроэнтерита в период 2021‒2023 гг. В 1085 случаях (29,2%) была обнаружена РНК РВ, которую в дальнейшем использовали для G/[P]-генотипирования методом ОТ-ПЦР и/или секвенирования. В 792 образцах (72,9%) был определен G[P]-генотип РВ. В 192 образцах (17,7%) был частично установлен G- либо P-генотип. Результаты генотипирования, проведенного по двум генам (VP4 и VP7), показали, что РВА, циркулирующие на территории Нижнего Новгорода, характеризовались высоким разнообразием. Спектр идентифицированных методом ПЦР G-генотипов РВ включал 4 широко распространенных в мире (G1, G2, G3 и G9), и 2 редких для России (G6 и G8). Набор Р-генотипов включал 4 типа, среди которых были часто встречающиеся P[4], P[8] и редкие P[6] и P[9]. Всего было обнаружено 11 G/P-комбинаций: широко распространенные G3P[8], G9P[8], G2P[4], G1P[8], а также редкие G8P[8], G3P[9], G9P[4], G3P[6], G2P[8], G6P[9] и G3P[4] (рис. 1).
Рис. 1. Распределение штаммов РВА основных G[P]-генотипов в Нижнем Новгороде в сезон 2021‒2023 гг. (в %).
Fig. 1. Distribution of RVA strains of the main G[P]-genotypes in Nizhny Novgorod in the season 2021‒2023 (in %).
Доминирующую позицию в изучаемый период занимали штаммы генотипа G3P[8] (59,5%), за ним следовали штаммы генотипа G9P[8] (10,8%). Для остальных генотипов был показан низкий долевой вклад: G9P[4] – 2,1%, G1P[8] – 1,3%, G2P[4] – 1,6%, G3P[4] – 1,3%, G8P[8] – 1,1%, G3P[6] – 0,8%, G3P[9] – 1,0%, G2P[8] – 0,8%, G6P[9] – 0,1%.
Выявление реассортантных ротавирусов и установление их полных генотипов
В ходе изучения РВА-содержащих образцов методом РНК-ПААГ были выявлены 6 штаммов (2 штамма генотипа G2P[4] и 4 – G3P[9]), которые имели так называемые «широкие» (AU-1-подобные) профили миграции геномных сегментов в ПААГ (медленно мигрирующий 5-й сегмент, быстро мигрирующие 6-й и 11-й сегменты) (рис. 2).
Рис. 2. Профили миграции сегментов днРНК типичных представителей DS-1-/Wa-подобных ротавирусов и исследуемых штаммов генотипов G2P[4] и G3P[9] в ПААГ. * − необычное положение сегмента относительно типичных представителей DS- и Wa-подобных геногрупп.
Fig. 2. Migration profiles of dsRNA segments of typical representatives of DS-1-/Wa-like rotaviruses and studied strains of genotypes G2P[4] and G3P[9] in PAGE. * − an unusual position of the segment relative to typical representatives of DS- and Wa-like gene groups.
Штаммы были секвенированы по 11 генам (VP1‒VP4, VP6, VP7, NSP1‒NSP5/6), что позволило определить их полные генотипы: G2-P[4]-I2-R2-C2-M2-A3-N2-T3-E2-H3, G2-P[4]-I2-R2-C2-M2-A3-N2-T3-E3-H2 и G3-P[9]-I2-R2-C2-M2-A3-N2-T3-E3-H3. Исследуемые штаммы имели смешанный набор генов двух разных геногрупп – DS-1-подобной (генотипы под номером 2) и AU-1-подобной (генотипы под номером 3), что свидетельствует об их реассортантном происхождении.
Филогенетический анализ ротавирусов на основе всех сегментов генома
Для изучения филогенетических взаимоотношений 6 выявленных РВ генотипов G2P[4] и G3P[9] были использованы нуклеотидные последовательности 11 генов. Филогенетический анализ позволил изучить внутригенотиповое разнообразие исследуемых штаммов на уровне линий и сублиний. Адаптированные деревья показаны на рис. 3, 4.
Рис. 3. Филогенетические деревья, построенные на основе нуклеотидных последовательностей структурных генов (VP1–VP4, VP6, VP7) штаммов ротавируса А. ■ − штаммы, полученные в данной работе; □ − нижегородские штаммы, взятые из GenBank.
Fig. 3. Phylogenetic trees based on nucleotide sequences structural genes (VP1–VP4, VP6, VP7) of rotavirus strains A. ■ − strains obtained in this work; □ − Nizhny Novgorod strains retrieved from GenBank.
Рис. 4. Филогенетические деревья, построенные на основе нуклеотидных последовательностей неструктурных генов (NSP1–NSP5/6) штаммов ротавируса А. ■ − штаммы, полученные в данной работе; □ − нижегородские штаммы, взятые из GenBank.
Fig. 4. Phylogenetic trees based on nucleotide sequences: nonstructural genes (NSP1–NSP5/6) of rotavirus strains A. ■ − strains obtained in this work; □ − Nizhny Novgorod strains retrieved from GenBank.
По каждому гену исследуемые штаммы входили в состав 1 или 2 кластеров. Наибольшее разнообразие показали гены VP4, VP7, NSP4 и NSP5/6 (таблица).
Таблица. Геномные констелляции ротавирусов Нижнего Новгорода на уровне субгенотипов
Table. Subgenotype-level genomic constellations of rotaviruses from Nizhny Novgorod
№ | Штаммы/ Strains | Сегменты генома / Genome segments | ||||||||||
VP7 | VP4 | VP6 | VP1 | VP2 | VP3 | NSP1 | NSP2 | NSP3 | NSP4 | NSP5/6 | ||
Штаммы, подобные РВ кошек / Feline-like strains | ||||||||||||
1 | ВА222 | G3-3-e | P[9] | I2-XIV | R2-XIII | C2-IX | M2-XI | A3 | N1 | T3 | E2-XVII | H3 |
2 | NN1061/16 | G6-I | P[9] | I2-XIV | R2-XIII | C2-IX | M2-XI | A3 | N2-XVI | T3 | E2-XVII | H3 |
3 | NN148/17 | G3-3-e | P[9] | I2-XIV | R2-XIII | C2-IX | M2-XI | A3 | N2-XVI | T3 | E3 | H3 |
4 | NN2748/18 | G3-3-e | P[9] | I2-XIV | R2-XIII | C2-IX | M2-XI | A3 | N2-XVI | T3 | E3 | H3 |
Типичные Ds-1-подобные штаммы / Typical DS-1-like strains | ||||||||||||
5 | NN96/18 | G2-IVa-3 | P[4]-IV-b | I2-V-1 | R2-V-1 | C2-IVa-1 | M2-VII | A2-IVa-1 | N2-V-1 | T2-V-1 | E2-VI | H2-IVa-1 |
6 | NN425/18 | G2-IVa-3 | P[4]-IV-b | I2-V-1 | R2-V-1 | C2-IVa-1 | M2-VII | A2-IVa-1 | N2-V-1 | T2-V-1 | E2-VI | H2-IVa-1 |
7 | NN437/18 | G2-IVa-1 | P[4]-IV-b | I2-V-1 | R2-V-1 | C2-IVa-1 | M2-VII | A2-IVa-1 | N2-V-1 | T2-V-1 | E2-VI | H2-IVa-1 |
8 | NN560/18 | G2-IVa-2 | P[4]-IV-a | I2-V-2 | R2-V-2 | C2-IVa-2 | M2-V | A2-IVa-2 | N2-V-2 | T2-V-2 | E2-VII | H2-IVa-2 |
Штаммы, полученные в данной работе / Strains obtained in this work | ||||||||||||
9 | 2853/21 | G3-3-e | P[9] | I2-XIV | R2-XIII | C2-IX | M2-XI | A3 | N2-XVI | T3 | E3 | H3 |
10 | 2885/21 | G3-3-e | P[9] | I2-XIV | R2-XIII | C2-IX | M2-XI | A3 | N2-XVI | T3 | E3 | H3 |
11 | 347/22 | G3-3-e | P[9] | I2-XIV | R2-XIII | C2-IX | M2-XI | A3 | N2-XVI | T3 | E3 | H3 |
12 | 2619/22 | G3-3-e | P[9] | I2-XIV | R2-XIII | C2-IX | M2-XI | A3 | N2-XVI | T3 | E3 | H3 |
13 | 1473/21 | G2-IVa-3 | P[4]-IV | I2-XIV | R2-XIII | C2-IX | M2-XI | A3 | N2-XVI | T3 | E2-XVII | H3 |
14 | 2924/21 | G2-IVa-3 | P[4]-IV | I2-XIV | R2-XIII | C2-IX | M2-XI | A3 | N2-XVI | T3 | E3 | H2-IVa |
Примечание. Исследуемые штаммы выделены жирным шрифтом. Красным цветом отмечены DS-1-подобные аллели, голубым – аллели РВ кошек, зеленым – Wa-подобные аллели.
Note. The studied strains are highlighted in bold. DS-1-like alleles are marked in red, alleles of RV cats are blue, and Wa-like alleles are green.
Штаммы, подобные кошачьему ротавирусу ВА222
Выявленные в данной работе 4 штамма генотипа G3P[9] относились к группе РВ, подобных штамму ВА222, выделенному от кошки. Эти штаммы (2853/21, 2885/21, 347/22 и 2619/21) были сходны по 9 генам (VP1‒VP4,VP6, VP7, NSP1, NSP3, NSP5/6) с прототипным штаммом РВА ВА222, но отличались от него генами NSP2 и NSP4, которые обладали другими генотипами. Наибольший уровень сходства нуклеотидных последовательностей был установлен для генов VP2, VP3, VP7, NSP1, NSP3, NSP5/6 и составил 98,7‒100,0%. Для генов VP1, VP4 и VP6 показан более низкий уровень гомологии ‒ 96,3‒98,1%.
Также исследуемые штаммы были родственны нижегородским ВА222-подобным РВ генотипов G3P[9] (NN148/17, NN2748/18) и G6P[9] (NN1061-16), выявленным ранее, в 2016‒2018 гг. С изолятами генотипа G3P[9] (NN148/17 и NN2748/18) исследуемые штаммы имели высокий уровень гомологии нуклеотидных последовательностей по всем 11 генам (99,1‒100,0%). Со штаммом генотипа G6P[9] (NN1061/16) было показано наиболее близкое родство по 8 генам (VP1‒VP4, NSP1‒NSP3, NSP5/6) с уровнем идентичности нуклеотидных последовательностей 98,7‒100,0%. Для гена VP6 установлен более низкий уровень сходства (96,0%). Гены VP7 и NSP4 имели другой генотип.
Нетипичные штаммы генотипа G2P[4]
Выявленные в настоящей работе два штамма РВА генотипа G2P[4] (1473/21 и 2924/21) имели смешанный набор генов. Они несли ВА222-подобную основу, в то время как гены VP4, VP7 и в одном случае ген NSP5/6 были получены в результате реассортации от типичных представителей DS-1-подобной геногруппы.
Наиболее близкородственными по дивергентным генам (VP4, VP7 и в одном случае NSP5/6) явились штаммы генотипа G2P[4] из Нижнего Новгорода, выявленные ранее в 2018 г. (NN560/18, NN96/18 и NN425/18). Уровень сходства нуклеотидных последовательностей при этом был довольно высок и составил 98,7‒99,9%.
По остальным генам (VP1‒VP3, VP6, NSP1‒NSP5/6) исследуемые штаммы имели близкое родство с ВА222-подобной группой РВ. Непосредственно с прототипным ВА222 образцы 1473/21 и 2924/21 были наиболее родственны по 3 генам: VP2, NSP3, NSP4 и VP2, VP3, NSP3 соответственно. Сходство нуклеотидных последовательностей в случае этих генов достигало 98,6‒99,8%, в то время как для остальных генов было ниже ‒ 95,2‒97,6%.
Также была установлена родственная связь исследуемых штаммов генотипа G2P[4] с нижегородскими РВ генотипов G3P[9] и G6P[9] (NN1061/16, NN148/17, NN2748/18), выявленными ранее, по всем ВА222-подобным генам. Так, исследуемый штамм 1473/21 разделял наибольшее количество генов (8 из 11) со штаммом NN148/17 (G3P[9]) (VP1‒VP3, VP6, NSP1‒NSP3, NSP5/6), демонстрируя при этом высокую идентичность нуклеотидных последовательностей ‒ 99,1‒99,9%. Со штаммом NN1061/16 (G6P[9]) он разделял 7 генов (VP1‒VP3, NSP2‒NSP5/6) с уровнем сходства нуклеотидных последовательностей 98,6‒99,8%. Пять генов были родственны таковым у штамма NN2748/18 (G3P[9]) (VP1, VP2, VP6, NSP2, NSP3). Гомология их нуклеотидных последовательностей составила 98,7‒99,7%.
Аналогично, штамм 2924/21 имел тесное филогенетическое родство со штаммом NN148/17 (G3P[9]) по 8 генам (VP1‒VP3, VP6, NSP1‒NSP4), демонстрируя высокую идентичность нуклеотидных последовательностей ‒ 98,8‒99,8%. Со штаммом NN2748/18 (G3P[9]) он разделял 6 генов (VP1‒VP3, VP6, NSP2, NSP3) с уровнем гомологии 98,5‒99,5%. В случае штамма NN1061/16 (G6P[9]) показано близкое родство по 5 генам (VP1‒VP3, NSP2, NSP3) с высоким уровнем сходства нуклеотидных последовательностей ‒ 99,1‒99,8%.
Обсуждение
В настоящей работе дана характеристика разнообразия генотипов РВА, циркулировавших в Нижнем Новгороде в 2021‒2023 гг. Спектр представлен 11 типами с доминированием G3P[8] (59,5%), за которым следовал G9P[8] (10,8%). К июню 2021 г. отмечалось снижение доли генотипов G9P[8] в нижегородской популяции РВ, с последующей сменой доминирующего генотипа на G3P[8] в июле 2021 г. [41]. Полученные сведения служат дополнением к информации о генетических перестройках в популяции РВ в процессе их многолетней циркуляции в Нижнем Новгороде, наблюдения за которыми ведутся с 1984 г. [13, 26, 32, 40‒44].
Представленные результаты также дополняют имеющиеся данные о генотиповой структуре популяций РВ на территории России. По данным референс-центра по мониторингу ОКИ, в зимне-весенний период 2021 г. на территории Российской Федерации (Московская, Томская, Иркутская, Свердловская, Новосибирская области, Республика Дагестан, Хабаровский край, Камчатский край) сохранялось доминирование генотипа G9P[8], но уже отмечалось постепенное возрастание доли генотипа G3P[8]. В 2022 г. произошло значительное уменьшение частоты встречаемости генотипа G9P[8] и показано превалирование в циркуляции на территории Российской Федерации РВ генотипа G3P[8], которое сохранилось и в 2023 г. [4, 5]. Представленная информация согласуется с данными, полученными в настоящей работе.
РВ генотипа G3 гена VP7 обладают широким спектром хозяев и встречаются у большинства восприимчивых видов животных (включая человека, кроликов, обезьян, свиней, птиц, кошек, собак, лошадей, мышей, коров и ягнят) [7, 45, 46]. У РВ человека он в большинстве случаев ассоциирован с генотипом P[8] гена VP4, но реже встречается в сочетании с генотипом P[9] [47‒49]. Комбинация G3P[9] распространена в основном у РВ кошек и собак [50], но благодаря реассортации, штаммы, подобные РВ животных, могут встречаться у человека [51]. Впервые РВА генотипа G3P[9] были обнаружены у человека в 1982 г. в Японии и Израиле [17, 22, 52]. Позднее их появление было зарегистрировано в Таиланде и Испании [53‒59]. На территории Нижнего Новгорода штаммы G3P[9] эпизодически наблюдались в популяции с 1984 г. В ранних исследованиях они изучались на основе отдельно взятых генов (VP4, VP6, VP7, NSP4) [60, 61], первое изучение данных штаммов на основе всех сегментов генома было проведено в 2023 г. [40].
РВ G3P[9] спорадически обнаруживаются у людей, образуя отдельную геногруппу, генетически обособленную от Wa-подобных и DS-1-подобных вирусов [49, 59, 62‒64]. Для человеческого G3P[9] показана возможность существования как минимум 8 разных комбинаций полных генотипов: G3-P[9]-I3-R3-C3-M3-A3-N3-T3-E3-H3 [9, 17], G3-P[9]-I3-R3-C3-M3-A3-N3-Т3-E3-H6 [16, 22, 51], G3-P[9]-I2-R2-C2-M2-A3-N2-T1-E2-H3 [46, 52], G3-P[9]-I2-R2-C2-M2-A3-N1-T6-E2-H3, G3-P[9]-I2-R2-C2-M2-A3-N2-T6-E2-H3 [65], G3-P[9]-I2-R2-C2-M2-A3-N1-T3-E2-H3 [66, 67], G3-P[9]-I3-R3-C2-M3-A3-N1-T6-E3-H3 [68, 69] и G3-P[9]-I2-R2-C2-M2-A3-N2-T3-E3-H3 [68, 70]. Интересно, что ген NSP1 всех штаммов G3P[9] животного и человеческого происхождения неизменно обладает генотипом A3, то время как генотип остальных генов может варьировать (VP6 – I2/I3; VP1 – R2/R3; VP2 – C2/C3; VP3 – M2/M3; NSP2 – N1/N2/N3; NSP3 – T1/T3/T6; NSP4 – E2/E3; NSP5/6 – H3/H6).
Выявленные и исследованные в настоящей работе РВА G3P[9] обладали комбинацией генотипов G3-P[9]-I2-R2-C2-M2-A3-N2-T3-E3-H3. Филогенетический анализ показал, что они разделяли 9 генов (VP1‒VP4, VP6, VP7, NSP1, NSP3, NSP5/6) с кошачьим РВ ВА222 (Италия, 2005 г.). Из них гены VP2, VP3, VP7, NSP1, NSP3 и NSP5 имели высокий процент идентичности нуклеотидных последовательностей (98,7‒100,0%), а VP1, VP4 и VP6 – более низкий (96,3‒98,1%). Нижегородские штаммы отличались от типового ВА222 генами NSP2 и NSP4, которые обладали другими генотипами.
Полученные результаты показывают близкое родство исследованных РВ с более ранними штаммами генотипа G3P[9] из Нижнего Новгорода. В период 2016‒2018 гг. были выявлены и исследованы на основе всех сегментов генома 3 аналогичных штамма РВА генотипа G3P[9] [40]. Их нуклеотидные последовательности использовались для проведения филогенетического анализа в настоящей работе. Сходство нижегородских штаммов G3P[9] разных лет составило для разных генов 99,7‒100,0%.
Межгрупповые реассортанты на основе ВА222-подобной комбинации ранее встречались на территории Южной Кореи. S. Jeong и соавт. (2014) в своей работе исследовали РВА генотипа G3P[9], выделенный в 2012 г. от невакцинированной 9-летней девочки с симптомами тяжелого гастроэнтерита. Исследуемый штамм имел генотип G3-P[9]-I2-R2-C2-M2-A3-N2-T3-E3-H3. Филогенетический анализ показал, что он разделял 9 генов (VP1‒VP4, VP6, VP7, NSP1, NSP3, NSP5/6) с прототипным ВА222, с разным уровнем сходства нуклеотидных последовательностей. По гену NSP2 была установлена кластеризация исследуемых штаммов с типичными DS-1-подобными РВ человека, а для NSP4 – с австралийским штаммом РВА кошек и собак. Этот штамм имел сложное эволюционное происхождение, потенциально включающее события реассортации между РВ кошек и DS-1-подобных РВА человека [70].
Аналогичные данные были получены группой исследователей из Китая. M. Cao и соавт. в 2023 г. исследовали 2 штамма генотипа G3P[9], выделенные в 2020 и 2023 гг. от 12-месячной девочки и 16-месячного мальчика соответственно. Оба штамма имели комбинацию генотипов: G3-P[9]-I2-R2-C2-M2-A3-N2-T3-E3-H3. Филогенетический анализ установил, что оба исследуемых РВА разделяли 8 генов (VP1‒VP4, VP7, NSP1, NSP3 и NSP4) с РВА, подобными РВ кошек и собак, с разным уровнем сходства нуклеотидных последовательностей (99,3‒99,6%). Оставшиеся 3 гена (VP6, NSP2 и NSP5/6) были связаны с типичными DS-1-подобными РВ человека (99,3‒100,0%) [68].
В проведенном исследовании показана циркуляция необычных штаммов, которые имели генотипы G2-P[4]-I2-R2-C2-M2-A3-N2-T3-E2-H3 и G2-P[4]-I2-R2-C2-M2-A3-N2-T3-E3-H2. Гены VP1‒VP3, VP6, NSP1, NSP3‒NSP5/6 одного из них и VP1‒VP3, VP6, NSP1, NSP3, NSP4 второго были связаны с прототипным штаммом ВА222 РВА (Италия, 2005 г.). Остальные гены (а именно VP4, VP7 и в случае одного штамма NSP5/6) произошли от человеческих DS-1-подобных штаммов генотипа G2P[4]. Штаммы с такой комбинацией генотипов были выявлены и охарактеризованы с помощью секвенирования всех сегментов генома впервые. В России и в мире сообщений о выявлении аналогичных штаммов G2P[4] ранее не отмечено. По всем генам, как по ВА222, так и по DS-1-подобным, данные штаммы имели ближайших родственников из Нижнего Новгорода, что позволяет предположить их локальное происхождение.
Реассортантные штаммы, охарактеризованные в данной работе, не могли быть выявлены с помощью G/[P]-генотипирования методом ПЦР, которое рутинно используется для изучения РВ. Это подчеркивает важность применения полногеномной системы классификации и современных методов секвенирования для мониторинга циркуляции этого возбудителя.
Заключение
Результаты исследования в совокупности с полученными ранее данными расширяют представление о значительном генетическом разнообразии РВ и роли реассортантов в его поддержании. Эта информация важна для создания новых ротавирусных вакцин, понимания эволюционных процессов в популяции РВА и указывает на актуальность изучения генетической эволюции редких и новых штаммов вирусов. Для контроля появления новых вариантов необходим постоянный молекулярный мониторинг циркулирующих РВА с использованием полногеномной классификации.
Финансирование. Исследование проведено в рамках выполнения Государственного задания Федеральной службы по надзору в сфере защиты прав потребителей и благополучия человека (Роспотребнадзор).
Конфликт интересов. Авторы декларируют отсутствие явных и потенциальных конфликтов интересов, связанных с публикацией настоящей статьи.
Этическое утверждение. Исследование проводилось при добровольном информированном согласии пациентов. Протокол исследования одобрен Локальным этическим комитетом ФБУН «Нижегородский НИИ эпидемиологии и микробиологии им. академика И.Н. Блохиной» Федеральной службы по надзору в сфере защиты прав потребителей и благополучия человека (Роспотребнадзор) (Протокол № 6 от 24.03.2021).
Об авторах
Елена Игоревна Великжанина
ФБУН «Нижегородский научно-исследовательский институт эпидемиологии и микробиологии им. академика И.Н. Блохиной» Федеральной службы по надзору в сфере защиты прав потребителей и благополучия человека (Роспотребнадзор)
Автор, ответственный за переписку.
Email: www.e_velikzhanina@mail.ru
ORCID iD: 0000-0003-4069-1427
младший научный сотрудник лаборатории молекулярной эпидемиологии вирусных инфекций
Россия, 603950, г. Нижний НовгородТатьяна Александровна Сашина
ФБУН «Нижегородский научно-исследовательский институт эпидемиологии и микробиологии им. академика И.Н. Блохиной» Федеральной службы по надзору в сфере защиты прав потребителей и благополучия человека (Роспотребнадзор)
Email: tatyana.sashina@gmail.com
ORCID iD: 0000-0003-3203-7863
канд. биол. наук, старший научный сотрудник лаборатории молекулярной эпидемиологии вирусных инфекций
Россия, 603950, г. Нижний НовгородОльга Владимировна Морозова
ФБУН «Нижегородский научно-исследовательский институт эпидемиологии и микробиологии им. академика И.Н. Блохиной» Федеральной службы по надзору в сфере защиты прав потребителей и благополучия человека (Роспотребнадзор)
Email: Olga.morozova.bsc@gmail.com
ORCID iD: 0000-0002-8058-8187
канд. биол. наук, старший научный сотрудник лаборатории молекулярной эпидемиологии вирусных инфекций
Россия, 603950, г. Нижний НовгородАлександр Юрьевич Кашников
ФБУН «Нижегородский научно-исследовательский институт эпидемиологии и микробиологии им. академика И.Н. Блохиной» Федеральной службы по надзору в сфере защиты прав потребителей и благополучия человека (Роспотребнадзор)
Email: a.kashn@yandex.ru
ORCID iD: 0000-0003-1033-7347
научный сотрудник лаборатории молекулярной эпидемиологии вирусных инфекций
Россия, 603950, г. Нижний НовгородНаталия Владимировна Епифанова
ФБУН «Нижегородский научно-исследовательский институт эпидемиологии и микробиологии им. академика И.Н. Блохиной» Федеральной службы по надзору в сфере защиты прав потребителей и благополучия человека (Роспотребнадзор)
Email: epifanovanv@mail.ru
ORCID iD: 0000-0001-7679-8029
канд. биол. наук, ведущий научный сотрудник лаборатории молекулярной эпидемиологии вирусных инфекций
Россия, 603950, г. Нижний НовгородНадежда Алексеевна Новикова
ФБУН «Нижегородский научно-исследовательский институт эпидемиологии и микробиологии им. академика И.Н. Блохиной» Федеральной службы по надзору в сфере защиты прав потребителей и благополучия человека (Роспотребнадзор)
Email: novikova_na@mail.ru
ORCID iD: 0000-0002-3710-6648
д-р биол. наук, профессор, ведущий научный сотрудник, заведующая лабораторией молекулярной эпидемиологии вирусных инфекций
Россия, 603950, г. Нижний НовгородСписок литературы
- GBD 2016 Diarrheal Disease Collaborators. Estimates of the global, regional, and national morbidity, mortality, and etiologies of diarrhea in 195 countries: a systematic analysis for the Global Burden of Disease Study 2016. Lancet Infect. Dis. 2018; 18(11): 1211–28. https://doi.org/10.1016/S1473-3099(18)30362-1
- Troeger С., Khalil I.A., Rao P.C., Cao S., Blacker B.F., Ahmed T., et al. Rotavirus vaccination and the global burden of rotavirus diarrhea among children younger than 5 years. JAMA Pediatr. 2018; 172(10): 958–65. https://doi.org/10.1001/jamapediatrics.2018.1960
- Sindhu K.N., Babji S., Ganesan S. Impact of rotavirus vaccines in low and middle-income countries. Curr. Opin. Infect. Dis. 2017; 30(5): 473–81. https://doi.org/10.1097/QCO.0000000000000397
- Государственный доклад «О состоянии санитарно-эпидемиологического благополучия населения в Российской Федерации в 2022 году». М.; 2023.
- Государственный доклад «О состоянии санитарно-эпидемиологического благополучия населения в Российской Федерации в 2023 году». М.; 2024.
- Rixon F., Taylor P., Desselberger U. Rotavirus RVA segments sized by electron microscopy. J. Gen. Virol. 1984; 56(1): 233–9. https://doi.org/10.1099/0022-1317-65-1-233
- Estes M.K., Kapikian A.Z. Rotaviruses. In: Fields B.N., Knipe D.M., Howley P.M., eds. Fields Virology. Philadelphia: Wolters Kluwer Health/Lippincott Williams & Wilkins; 2007: 1917–73.
- Estes M.K., Greenberg H.B. Rotaviruses. In: Knipe D.M., Howley P.M. eds. Fields Virology. Philadelphia: Wolters Kluwer Health/Lippincott Williams & Wilkins; 2013: 1347–1401.
- Matthijnssens J., Ciarlet M., Rahman M., Attoui H., Banyai K., Estes M.K., et al. Recommendations for the classification of group A rotaviruses using all 11 genomic RNA segments. Arch. Virol. 2008; 153(8): 1621–9. https://doi.org/10.1007/s00705-008-0155-1
- RCWG. Rotavirus classification working group; 2024. Available at: https://rega.kuleuven.be/cev/viralmetagenomics/virus-classification/rcwg
- Matthijnssens J., Heylen E., Zeller M., Rahman M., Lemey P., Van Ranst M. Phylodynamic analyses of rotavirus genotypes G9 and G12 underscore their potential for swift global spread. Mol. Biol. Evol. 2010; 27(10): 2431–6. https://doi.org/10.1093/molbev/msq137
- Maes P., Matthijnssens J., Rahman M., Van Ranst M. Rota C: a web-based tool for the complete genome classification of group A rotaviruses. BMC Microbiol. 2009; 9: 238. https://doi.org/10.1186/1471-2180-9-238
- Sashina T.A., Morozova O.V., Epifanova N.V., Novikova N.A. Predominance of new G9P[8] rotaviruses closely related to Turkish strains in Nizhny Novgorod (Russia). Arch. Virol. 2017; 162(8): 2387–92. https://doi.org/10.1007/s00705-017-3364-7
- Kiseleva V., Faizuloev E., Meskina E., Marova A., Oksanich A., Samartseva T., et al. Molecular-genetic characterization of human rotavirus A strains circulating in Moscow, Russia (2009–2014). Virol. Sin. 2018; 33(4): 304–13. https://doi.org/10.1007/s12250-018-0043-0
- Doro R., Laszlo B., Martella V., Leshem E., Gentsch J., Parashar U., et al. Review of global rotavirus strain prevalence data from six years post vaccine licensure surveillance: is there evidence of strain selection from vaccine pressure? Infect. Genet. Evol. 2014; 28: 446–61. https://doi.org/10.1016/j.meegid.2014.08.017
- Santos N., Hoshino Y. Global distribution of rotavirus serotypes/genotypes and its implication for the development and implementation of an effective rotavirus vaccine. Rev. Med. Virol. 2005; 15(1): 29–56. https://doi.org/10.1002/rmv.448
- Matthijnssens J., Ciarlet M., Heiman E., Arijs I., Delbeke T., Mcdonald S.M., et al. Full genome-based classification of rotaviruses reveals a common origin between human Wa-like and porcine rotavirus strains and human DS-1-like and bovine rotavirus strains. J. Virol. 2008; 82(7): 3204–19. https://doi.org/10.1128/JVI.02257-07
- Heiman E.M., Mcdonald S.M., Barro M., Taraporewala Z.F., Bar-Magen T. Ndpatton J.T. Group A human rotavirus genomics: evidence that gene constellations are influenced by viral protein interactions. J. Virol. 2008; 82(22): 11106–16. https://doi.org/10.1128/JVI.01402-08
- Uprety T., Wang D., Li F. Recent advances in rotavirus reverse genetics and its utilization in basic research and vaccine development. Arch. Virol. 2021; 166(9): 2369–86. https://doi.org/10.1007/s00705-021-05142-7
- Nakagomi O., Ohshima A., Aboudy Y., Shif I., Mochizuki M., Nakagomi T., et al. Molecular identification by RNA-RNA hybridization of a human rotavirus that is closely related to rotaviruses of feline and canine origin. J. Clin. Microbiol. 1990; 28(6): 1198–203. https://doi.org/10.1128/jcm.28.6.1198-1203.1990
- Matthijnssens J., Van Ranst M. Genotype constellation and evolution of group A rotaviruses infecting humans. Curr. Opin. Virol. 2012; 2(4): 426–33. https://doi.org/10.1016/j.coviro.2012.04.007
- Nakagomi T., Nakagomi O. RNA-RNA hybridization identifies a human rotavirus that is genetically related to feline rotavirus. J. Virol. 1989; 63(3): 1431–4. https://doi.org/10.1128/JVI.63.3.1431-1434.1989
- Tsugawa T., Rainwater-Lovett K., Tsutsumi H. Human G3P[9] rotavirus strains possessing an identical genotype constellation to AU-1 isolated at high prevalence in Brazil, 1997–1999. J. Gen. Virol. 2015; 96(Pt. 3): 590–600. https://doi.org/10.1099/vir.0.071373-0
- Cook N., Bridger J., Kendall K., Gomara M.I., El-Attar L., Gray J. The zoonotic potential of rotavirus. J. Infect. 2004; 48(4): 289–302. https://doi.org/10.1016/j.jinf.2004.01.018
- Jain S., Vashistt J., Changotra H. Rotaviruses: is their surveillance needed? Vaccine. 2014; 32(27): 3367–78. https://doi.org/10.1016/j.vaccine.2014.04.037
- Novikova N.A., Sashina T.A., Epifanova N.V., Kashnikov A.U., Morozova O.V. Long-term monitoring of G1P[8] rotaviruses circulating without vaccine pressure in Nizhny Novgorod, Russia, 1984–2019. Arch. Virol. 2020; 165(4): 865–75. https://doi.org/10.1007/s00705-020-04553-2
- Gouvea V., Glass R.I., Woods P., Taniguchi K., Clark H.F., Forrester B., et al. Polymerase chain reaction amplification and typing of rotavirus nucleic acid from stool specimens. J. Clin. Microbiol. 1990; 28(2): 276–82. https://doi.org/10.1128/jcm.28.2.276-282.1990
- Gentsch J.R., Glass R.I., Woods P., Gouvea V., Gorziglia M., Flores J., et al. Identification of group A rotavirus gene 4 types by polymerase chain reaction. J. Clin. Microbiol. 1992; 30(6): 1365–73. https://doi.org/10.1128/jcm.30.6.1365-1373.1992
- Maunula L., von Bonsdorff C.H. Short sequences define genetic lineages: phylogenetic analysis of group A rotaviruses based on partial sequences of genome segments 4 and 9. J. Gen. Virol. 1998; 79(Pt. 2): 321–32. https://doi.org/10.1099/0022-1317-79-2-321
- Iturriza-Gómara M., Isherwood B., Desselberger U., Gray J. Reassortment in vivo: driving force for diversity of human rotavirus strains isolated in the United Kingdom between 1995 and 1999. J. Virol. 2001; 75(8): 3696–705. https://doi.org/10.1128/JVI.75.8.3696-3705.2001
- Iturriza-Gómara M., Kang G., Gray J. Rotavirus genotyping: keeping up with an evolving population of human rotaviruses. J. Clin. Virol. 2004; 31(4): 259–65. https://doi.org/10.1016/j.jcv.2004.04.009
- Sashina T.A., Morozova O.V., Epifanova N.V., Novikova N.A. Genotype constellations of the rotavirus A strains circulating in Nizhny Novgorod, Russia, 2017-2018. Infect. Genet. Evol. 2020; 85: 104578. https://doi.org/10.1016/j.meegid.2020.104578
- Kimura M. A simple method for estimating evolutionary rates of base substitutions through comparative studies of nucleotide sequences. J. Mol. Evol. 1980; 16(2): 111–20. https://doi.org/10.1007/BF01731581
- Kumar S., Stecher G., Li M., Knyaz C., Tamura K. MEGA X: Molecular Evolutionary Genetics Analysis across Computing Platforms. Mol. Biol. Evol. 2018; 35(6): 1547–9. https://doi.org/10.1093/molbev/msy096
- Rahman M., Matthijnssens J., Yang X., Delbeke T., Arijs I., Taniguchi K., et al. Evolutionary history and global spread of the emerging g12 human rotaviruses. J. Virol. 2007; 81(5): 2382–90. https://doi.org/10.1128/JVI.01622-06
- Mukherjee A., Dutta D., Ghosh S., Bagchi P., Chattopadhyay S., Nagashima S., et al. Full genomic analysis of a human group A rotavirus G9P[6] strain from Eastern India provides evidence for porcine-to-human interspecies transmission. Arch. Virol. 2009; 154(5): 733–46. https://doi.org/10.1007/s00705-009-0363-3
- Ndze V.N., Esona M.D., Achidi E.A., Gonsu K.H., Doro R., Marton S., et al. Full genome characterization of human Rotavirus A strains isolated in Cameroon, 2010-2011: diverse combinations of the G and P genes and lack of reassortment of the backbone genes. Infect. Genet. Evol. 2014; 28: 537–60. https://doi.org/10.1016/j.meegid.2014.10.009
- Wang Y.H., Pang B.B., Ghosh S., Zhou X., Shintani T., Urushibara N., et al. Molecular epidemiology and genetic evolution of the whole genome of G3P[8] human rotavirus in Wuhan, China, from 2000 through 2013. PLoS One. 2014; 9(3): e88850. https://doi.org/10.1371/journal.pone.0088850
- Agbemabiese C.A., Nakagomi T., Doan Y.H., Nakagomi O. Whole genomic constellation of the first human G8 rotavirus strain detected in Japan. Infect. Genet. Evol. 2015; 35: 184–93. https://doi.org/10.1016/j.meegid.2015.07.033
- Sashina T.A., Velikzhanina E.I., Morozova O.V., Epifanova N.V., Novikova N.A. Detection and full-genotype characterization of rare and reassortant Rotavirus A strains in Nizhny Novgorod, European part of Russia. Arch. Virol. 2023; 168(8): 215. https://doi.org/10.1007/s00705-023-05838-y
- Morozova O.V., Sashina T.A., Epifanova N.V., Velikzhanina E.I., Novikova N.A. Phylodynamic characteristics of reassortant DS-1-like G3P[8]-strains of rotavirus type A isolated in Nizhny Novgorod (Russia). Braz. J. Microbiol. 2023; 54(4): 2867–77. https://doi.org/10.1007/s42770-023-01155-3
- Новикова Н.А., Сашина Т.А., Солнцев Л.А., Епифанова Н.В., Кашников А.Ю., Погодина Л.В. и др. Проявления эпидемического процесса ротавирусной инфекции в Нижнем Новгороде в предвакцинальный период. Журнал микробиологии, эпидемиологии и иммунобиологии. 2017; 94(5): 46–52. https://doi.org/10.36233/0372-9311-2017-5-46-52 https://elibrary.ru/ysqaeq
- Морозова О.В., Сашина Т.А., Новикова Н.А. Обнаружение и молекулярная характеристика реассортантных DS-1-подобных G1P[8] штаммов ротавируса группы А. Вопросы вирусологии. 2017; 62(2): 91–6. https://doi.org/10.18821/0507-4088-2017-62-2-91-96 https://elibrary.ru/yjkhhh
- Сашина Т.А., Морозова О.В., Епифанова Н.В., Кашников А.Ю., Леонов А.В., Новикова Н.А. Молекулярный мониторинг ротавирусов (Reoviridae: Sedoreovirinae: Rotavirus: Rotavirus A), циркулирующих в Нижнем Новгороде (2012–2020 гг.): обнаружение штаммов с новыми генетическими характеристиками. Вопросы вирусологии. 2021; 66(2): 140–51. https://doi.org/10.36233/0507-4088-46 https://elibrary.ru/azvpec
- Martella V., Ciarlet M., Camarda A., Pratelli A., Tempesta M., Greco G. et al. Molecular characterization of the VP4, VP6, VP7, and NSP4 genes of lapine rotaviruses identified in Italy: emergence of a novel VP4 genotype. Virology. 2003; 314(1): 358–70. https://doi.org/10.1016/s0042-6822(03)00418-5
- Grant L., Esona M., Gentsch J., Watt J., Reid R., Weatherholtz R., et al. Detection of G3P[3] and G3P[9] rotavirus strains in American Indian children with evidence of gene reassortment between human and animal rotaviruses. J. Med. Virol. 2011; 83(7): 1288–99. https://doi.org/10.1002/jmv.22076
- Nakagomi O., Kaga E. Distinctness of NSP1 gene of human rotavirus AU-1 from NSP1 gene of other human genogroups. Res. Virol. 1995; 146(6): 423–8. https://doi.org/10.1016/0923-2516(96)80902-2
- Theamboonlers A., Veravigrom M., Yambangyang O., Trairatvorakul P., Chongsrisawat V., Poovorawan Y. The incidence of rotavirus a isolates of G genotype in Thailand in 2002–2004. Acta Virol. 2005; 49(2): 111–5.
- Martella V.A., Potgieter C., Lorusso E., De Grazia S., Giammanco G.M., Matthijnssens J., et al. A feline rotavirus G3P[9] carries traces of multiple reassortment events and resembles rare human G3P[9] rotaviruses. J. Gen. Virol. 2011; 92(Pt. 5): 1214–21. https://doi.org/10.1099/vir.0.027425-0
- Matthijnssens J., Ciarlet M., Mcdonald S.M., Attoui H., Banyai K., Brister J.R. et al. Uniformity of rotavirus strain nomenclature proposed by the rotavirus classification working group (RCWG). Arch. Virol. 2011; 156(8): 1397–413. https://doi.org/10.1007/s00705-011-1006-z
- Theamboonlers A., Maiklang O., Thongmee T., Chieochansin T., Vuthitanachot V., Poovorawan Y. Complete genome analysis of a rare human G3P[9] rotavirus posing as an AU-1 like strain. Springerplus. 2013; 2: 569. https://doi.org/10.1186/2193-1801-2-569
- Wang Y.H., Pang B.B., Zhou X., Ghosh S., Tang W.F. Peng J.S., et al. Complex evolutionary patterns of two rare human G3P[9] rotavirus strains possessing a feline/canine-like H6 genotype on an AU-1-like genotype constellation. Infect. Genet. Evol. 2013; 16: 103–12. https://doi.org/10.1016/j.meegid.2013.01.016
- Sanchez-Fauquier A., Montero V., Moreno S., Sole M., Colomina J., Iturriza-Gomara M., et al. Human rotavirus G9 and G3 as major cause of diarrhea in hospitalized children, Spain. Emerg. Infect. Dis. 2006; 12(10): 1536–41. https://doi.org/10.3201/eid1210.060384
- Khamrin P., Maneekarn N., Peerakome S., Tonusin S., Phan T.G., Okitsu S., et al. Molecular characterization of rare G3P[9] rotavirus strains isolated from children hospitalized with acute gastroenteritis. J. Med. Virol. 2007; 79(6): 843–51. https://doi.org/10.1002/jmv.20840
- Inoue Y., Kitahori Y. Rare group a rotavirus G3P[9] isolated in Nara Prefecture, Japan. Jpn J. Infect. Dis. 2006; 59(2): 139–40.
- Iizuka M., Chiba M., Masamune O., Kaga E., Nakagomi T., Nakagomi O. A highly conserved genomic RNA constellation of Japanese isolates of human rotaviruses carrying G serotype 3 and P serotype 9. Res. Virol. 1994; 145(1): 21–4. https://doi.org/10.1016/s0923-2516(07)80003-3
- Griffin D.D., Nakagomi T., Hoshino Y., Nakagomi O., Kirkwood C.D., Parashar U.D., et al. Characterization of nontypeable rotavirus strains from the United States: identification of a new rotavirus reassortant (P2A[6], G12) and rare P3[9] strains related to bovine rotaviruses. Virology. 2002; 294(2): 256–69. https://doi.org/10.1006/viro.2001.1333
- Gollop R., Nakagomi O., Silberstein I., Shulman L.M., Greenberg H.B., Mendelson E., et al. Three forms of AU-1 like human rotaviruses differentiated by their overall genomic constellation and by the sequence of their VP8*. Arch. Virol. 1998; 143(2): 263–77. https://doi.org/10.1007/s007050050285
- Kaga E., Iizuka M., Nakagomi T., Nakagomi O. The distribution of G (VP7) and P (VP4) serotypes among human rotaviruses recovered from Japanese children with diarrhea. Microbiol. Immunol. 1994; 38(4): 317–20. https://doi.org/10.1111/j.1348-0421.1994.tb01784.x
- Сашина Т.А., Морозова О.В., Новикова Н.А. G/[P]-типы ротавируса А в г. Нижнем Новгороде: 2012-2014 гг. Инфекция и иммунитет. 2014; 4(1): 91. https://elibrary.ru/vzqyan
- Новикова Н.А., Пономарева Н.В., Новиков Д.В., Прилипов А.Г., Епифанова Н.В., Голицына Л.Н. Анализ нуклеотидных последовательностей гена NSP4 ротавирусов группы А, изолированных в Нижнем Новгороде. Вопросы вирусологии. 2008; 53(6): 35–9. https://elibrary.ru/kaxfub
- Nakagomi O., Nakagomi T., Oyamada H., Suto T. Relative frequency of human rotavirus subgroups 1 and 2 in Japanese children with acute gastroenteritis. J. Med. Virol. 1985; 17(1): 29–34. https://doi.org/10.1002/jmv.1890170105
- Nakagomi O., Nakagomi T. Molecular evidence for naturally occurring single VP7 gene substitution reassortant between human rotaviruses belonging to two different genogroups. Arch. Virol. 1991; 119(1-2): 67–81. https://doi.org/10.1007/BF01314324
- Nakagomi O., Nakagomi T. Molecular epidemiology of human rotaviruses: genogrouping by RNA–RNA hybridization. Arch. Virol. 1996; 12: 93–8. https://doi.org/10.1007/978-3-7091-6553-9_11
- De Grazia S., Giammanco G.M., Potgieter C.A., Matthijnssens J., Banyai K., Platia M.A., et al. Unusual assortment of segments in 2 rare human rotavirus genomes. Emerg. Infect. Dis. 2010; 16(5): 859–62. https://doi.org/10.3201/eid1605.091826
- Banyai K., Laszlo B., Duque J., Steele A.D., Nelson E. Anthony S., et al. Systematic review of regional and temporal trends in global rotavirus strain diversity in the pre rotavirus vaccine era: insights for understanding the impact of rotavirus vaccination programs. Vaccine. 2012; 30(1): 122–30. https://doi.org/10.1016/j.vaccine.2011.09.111
- Tsugawa T., Hoshino Y. Whole genome sequence and phylogenetic analyses reveal human rotavirus G3P[3] strains Ro1845 and HCR3A are examples of direct virion transmission of canine/feline rotaviruses to humans. Virology. 2008; 380(2): 344–53. https://doi.org/10.1016/j.virol.2008.07.041
- Cao M., Yuan F., Zhang W., Wang X., Ma J., Ma X., et al. Genomic analysis of two rare human G3P[9] rotavirus strains in Ningxia, China. Infect. Genet. Evol. 2023; 116: 105518. https://doi.org/10.1016/j.meegid.2023.105518
- Mijatovic-Rustempasic S., Roy S., Sturgeon M., Rungsrisuriyachai K., Esona M.D., Degroat D., et al. Full-genome sequence of a rare human G3P[9] rotavirus strain. Genome Announc. 2014; 2(2): e00143–14. https://doi.org/10.1128/genomeA.00143-14
- Jeong S., Lim I., Kim W. Whole-genome analysis of a rare human Korean G3P[9] rotavirus strain suggests a complex evolutionary origin potentially in evolving reassortment events between feline and bovine rotaviruses. PLoS One. 2014; 9(5): e97127. https://doi.org/10.1371/journal.pone.0097127
Дополнительные файлы