Птичий грипп: «по ком звонит колокол»?
- Авторы: Жирнов О.П.1,2, Львов Д.К.1
-
Учреждения:
- Институт вирусологии имени Д.И. Ивановского ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи», Минздрава России
- Русско-немецкая академия медико-социальных и биотехнологических наук, Инновационный центр Сколково
- Выпуск: Том 69, № 2 (2024)
- Страницы: 101-118
- Раздел: РЕДАКЦИОННАЯ КОНЦЕПЦИЯ
- URL: https://virusjour.crie.ru/jour/article/view/16636
- DOI: https://doi.org/10.36233/10.36233/0507-4088-213
- EDN: https://elibrary.ru/gtekdx
- ID: 16636
Цитировать
Аннотация
Семейство Orthomyxoviridae состоит из 9 родов, включая Alphainfluenza viruses, куда входят вирусы гриппа птиц. У двух субтипов ‒ Н5 и Н7 ‒ помимо обычных низковирулентных штаммов описана особая разновидность высоковирулентного вируса птиц, способного вызывать заболеваемость у птиц с летальностью выше 60%. Эти и ряд других разновидностей вируса гриппа, тесно связанных с птицами, принято обозначать термином «птичий вирус гриппа». Различие высоко- (HPAI) и низковирулентных (LPAI) вирусов гриппа обусловлено структурой аргининсодержащего сайта протеолитической активации в белке НА. Вирус высоковирулентного птичьего гриппа Н5 выявлен около 100 лет назад и на протяжении этого времени вызывает эпизоотии среди диких и домашних птиц, и лишь несколько локальных эпизодов болезни зарегистрированы среди людей с начала XXI в. В последние годы зафиксирован резкий подъем заболеваемости птиц высоковирулентным вирусом Н5N1 (клайд h2.3.4.4b) на всех континентах планеты, сопровождающийся переходом вируса на разные виды млекопитающих. Регистрируемая глобальная смертность среди домашних и сельскохозяйственных птиц от данного субтипа приближается к уровню 1 млрд особей. Опасным эпидемическим фактором становятся участившиеся вспышки птичьего гриппа с высокой летальностью среди млекопитающих, в частности морских львов и тюленей в Северной и Южной Америке, норок и пушных зверей в Испании и Финляндии, домашних и уличных кошек в Польше. Изолированные от млекопитающих штаммы птичьего гриппа H5N1 клайда h2.3.4.4b имеют признаки частичной адаптации к организму человека в генах РВ2, NP, HA, NA, играющих главную роль в регуляции аэрозольной трансмиссии и круга хозяев вируса. Создавшаяся ситуация представляет реальную угрозу предадаптации вируса в организме млекопитающих как промежуточных хозяев с последующим переходом предадаптированного вируса в популяцию людей и формированием пандемии с катастрофическими последствиями.
Ключевые слова
Полный текст
Птичий грипп: «По ком звонит колокол»?*
Рост связанных с гриппом птиц неблагоприятных и угрожающих событий в мире определяет необходимость вновь вернуться к обсуждению данной проблемы.
Общая характеристика семейства Orthomyxoviridae
Семейство Orthomyxoviridae согласно современной классификации подразделяется на 9 родов: Alpha-, Beta-, Delta-, Gamma-, Thogoto-, Quaranja-, Mycis-, Sardino- и Isainfluenza viruses (табл. 1) [1]. В Orthomyxoviridae входят вирусы, имеющие наружную липопротеидную оболочку (оболочечные вирусы), одноцепочечную РНК негативной полярности в форме 7 или 8 сегментов (у разных родов), кодирующих до 16 уникальных белков [2]. Ортомиксовирусы имеют широкий ареал на планете во всех трех средах (водной, воздушной и на земле) и обладают тропизмом к широкому кругу организмов – членистоногих, рыб, птиц и млекопитающих, включая человека (табл. 1) [3]. В популяциях птиц – основного природного резервуара вирусов гриппа А, инфицирование протекает фекально-оральным путем.
Таблица 1. Классификация семейства Orthomyxoviridae
Table 1. Classification of the family Orthomyxoviridae
Номер рода вирусов Virus genus number | Наименование рода Genus name | Спектр хозяев вируса Host spectrum of the virus |
1. 2. 3. 4. | Genus: Alphainfluenzavirus (Flu A) Genus: Betainfluenzavirus (Flu B) Genus: Deltainfluenzavirus (Flu C) Genus: Gammainfluenzavirus (Flu D) | Птицы/Birds (Aves), Человек/Human (Homo sapiens), Млекопитающие/Mammalians (Mammalia) |
5. 6. | Genus: Thogotovirus Genus: Quaranjavirus | Членистоногие/Arthropods (Arthropoda): Клещи иксодовые/Ticks (Ixodidae), Клещи аргасовые/Argasid mites (Argasidae) |
7. 8. 9. | Genus Mykissvirus Genus: Sardinovirus Genus: Isavirus | Рыбы/Fishes (Ichthya) |
Примечание. Классификация семейства Orthomyxoviridae в соответствии с рекомендациями ICTV-2022 [1].
Note. Classification of the family Orthomyxoviridae in accordance with the recommendations of ICTV-2022 [1].
Особенностью ортомиксовирусов является их высокая изменчивость, которая обусловлена функциональными свойствами вирусной РНК-полимеразы и сегментированной структурой вирусного генома. Вирусная РНК-полимераза обеспечивает синтез и репликацию вирусного генома, которые она осуществляет с большим количеством случайных ошибок, составляющим около одной мутации на 104 нуклеотидов генома за одно прочитывание. Для сравнения отметим, что ДНК-зависимая РНК-полимераза II человека допускает ошибочность в одну мутацию лишь при полимеризации 108 нуклеотидов в молекуле синтезированной матричной РНК (мРНК) [4]. Высокая ошибочность вирусной полимеразы порождает высокую изменчивость вируса и образование большого количества вирусных вариантов, так называемых вирусных квазивидов, в вирусном потомстве. Генетическое разнообразие вирусной популяции позволяет вирусу реализовать транстаксонный переход к новому хозяину. Данный механизм постепенного накопления мутаций адаптационного характера получил название «вирусного дрейфа». Важно отметить, что в основе дрейфа лежит двоякий механизм, регулирующий, во-первых, приспособление вируса к факторам организма-хозяина и, во-вторых, ускользание от его иммунного ответа. В результате взаимодействия на молекулярно-генетическом уровне популяций вируса и хозяина происходит формирование популяционного генофонда вируса [5]. Период его формирования составляет миллионы лет, на протяжении которых вирус тесно взаимодействует с элементами биосферы в меняющихся условиях среды обитания.
Второй механизм, обеспечивающий высокую природную вариабельность ортомиксовирусов, заключается в обмене (реассортации) сегментов геномной РНК между вирусами, адаптированными к разным видам животных [6, 7]. Скрещивание таких вирусных вариантов-адаптантов лежит в основе явления «генетического шифта», ведущего к появлению кардинально новых вирусных реассортантов и уникальных антигенных и функциональных разновидностей вируса гриппа, имеющих потенциал транстаксонного перехода вируса с одного вида макроорганизма на другой и смены круга хозяев и формирования новых пандемических вариантов.
Два уровня вирулентности вирусов гриппа А (род Alphainfluenzavirus)
Высокая изменчивость вируса гриппа А обусловливает формирование богатого популяционного генофонда и распространение в природе широкого спектра вирусных вариантов, обладающих выраженным разнообразием вирулентных и антигенных свойств [8–10]. Род Alphainfluenzavirus (вирусы гриппа А) состоит из 18 антигенных субтипов гемагглютинина (НА), имеющих нумерацию H1–H18, и 11 субтипов белка нейраминидазы (NA), имеющих нумерацию N1–N11. Согласно такой нумерации в названии варианта (штамма) вирусов гриппа указывают антигенный субтип HA и NA [11–13].
Среди 18 известных субтипов рода Alphavirus особое место занимают вирусы субтипов Н5 и Н7. Популяции данных вирусных субтипов состоят из двух биологических видов вирусов, обладающих низкой (LPAI) и высокой (HPAI) вирулентностью для макроорганизмов-хозяев [13, 14]. Вирусы с низкой вирулентностью вызывают локальный инфекционный процесс, как правило, респираторного или кишечного трактов хозяина, часто без клинически выраженных симптомов, с уровнем популяционной смертности не более 1% [15]. Высоковирулентные вирусы субтипов Н5 и Н7, получившие первоначальное историческое название в конце XIX столетия «куриная чума» или «куриный тиф», вызывают у большинства чувствительных животных, как правило домашних и сельскохозяйственных (с/х) видов, массивную вирусемию, обусловливая диссеминацию вируса в организме с развитием генерализованной (пантропной) формы инфекции с высокой летальностью, близкой к 100% [16]. В популяциях птиц, являющихся основным природным резервуаром вируса гриппа А, инфицирование происходит фекально-оральным путем. Биологическая система «вирус гриппа А – птицы», вероятно, функционирует в биосфере более 100 млн лет – с мелового периода мезозойской эры [5]. Кроме того, птицы являются ареной рекомбинантных процессов в результате межпопуляционных контактов, возникающих в периоды миграции, особенно в миграционных хабах, где скапливаются в огромных количествах популяции птиц из разных районов ареала [17–20]. Среди птиц главными резервентами вирусов гриппа А в природе служат виды отряда гусеобразных (Anseriformes), семейства утиных (Anatidae), отряда ржанокообразных (Charadriiformes), семейства куликов (Scolopatidae), чайковых (Laridae) и др. В природе вирусы гриппа А изолированы от более 100 видов птиц (12 отрядов, 25 семейств, более 50 родов) [5, 21–23]. Первый высоковирулентный вирус гриппа субтипа H5 был изолирован в Шотландии в 1959 г. от цыплят – штамм A/chicken/Scotland/1959 (H5N1) [24], затем в ЮАР в период эпизоотии среди мигрирующих арктических крачек Sterna macrura – штамм A/tern/South Africa/61 (H5N3) [25]. В 70-х годах сформировалась концепция о природной очаговости вируса гриппа А (рис. 1) [5, 8, 18, 21, 22, 26–29]. Следует иметь в виду, что птицы являются главным носителем и резервуаром в природе высоковирулентной разновидности представителей субтипа H5N1 рода Alphainfluenza-virus [28].
Рис. 1. Основоположники концепции природной очаговости вируса гриппа А (Orthomyxoviridae: Influenza A virus). На фото слева направо: Грэм Лавер (Австралия), Дмитрий Львов (СССР), Роберт Вебстер (США) [8, 22, 26, 27].
Fig. 1. Founders of the concept of natural focality of the Influenza A virus (Orthomyxoviridae: Influenza A virus). Pictured from left to right: Graham Laver (Australia), Dmitry Lvov (USSR), Robert Webster (USA) [8, 22, 26, 27].
Молекулярную основу кардинального различия вирулентных свойств вируса гриппа А определяет структура участка (т.н. протеолитического сайта) вирусного гемагглютинина НА0 (м.м. 75 кДа), в котором происходит протеолитическое разрезание на две субъединицы: НА1 (55 кДа) и НА2 (20 кДа), сохраняющие после разрезания между собой дисульфидную связь (рис. 2). Такое расщепление белка вирусного гемагглютинина активирует его функцию слияния липидных мембран (функция образования симпластов), делает вирион инфекционным и способным заражать новые клетки-мишени [30–33]. Феномен усиления вирулентности вируса гриппа посредством протеолиза НА0 на НА1+НА2 получил название протеолитической активации вируса [33, 34].
Рис. 2. Структура сайта протеолитической активации белка гемагглютинина (НА) вирусов гриппа А.
Показан участок белка НА в зоне соединения субъединиц НА1 (55 кДа) и НА2 (20 кДа), формирующихся после расщепления молекулы-предшественника НА0 (75 кДа). Слабопатогенные вирусы субтипов Н1–Н18 содержат единичный остаток аргинина (Arg) (панель А), тогда как у высокопатогенных субтипов Н5 и Н7 в участке протеолиза присутствуют несколько остатков аргинина и лизина (Lys), так называемый полиаргининовый сайт протеолиза (панель Б). Полиаргининовый сайт имеет высокую чувствительность к широкому спектру клеточных протеаз и легко расщепляется в подавляющем большинстве типов клеток-мишеней [16, 33, 35–37].
Fig. 2. Structure of the proteolytic activation site of the influenza A virus hemagglutinin (HA) protein.
The HA protein site in the junction zone of HA1 (55 kDa) and HA2 (20 kDa) subunits formed after cleavage of the precursor molecule, HA0 (75 kDa), is shown. Low pathogenic viruses of subtypes H1–H18 contain a single arginine (Arg) residue (panel A), whereas highly pathogenic subtypes H5 and H7 have several arginine and lysine (Lys) residues present in the proteolysis site, the so-called polyarginine site of proteolysis (panel B). The polyarginine site is highly sensitive to a wide range of cellular proteases and is readily cleaved in the vast majority of target cell types [16, 33, 35–37].
У вирусов с низкой вирулентностью протеолитический сайт НА состоит из одного остатка аргинина (Arg) ‒ так называемый «моноаргининовый сайт», тогда как у высоковирулентных вирусов гриппа он состоит из нескольких остатков аргинина и, возможно, лизина (Lys) ‒ так называемый «полиаргининовый сайт». Разрезание моноаргининового НА0 осуществляется трипсиноподобными протеазами и, как правило, происходит пострансляционно на клеточной плазматической мембране при выходе вириона из инфицированной клетки. Эффективность такого расщепления в инфицированыx клетках составляла около 30–40% молекул синтезированного белка НА0 [35, 36]. У высоковирулентных вирусов субтипов Н5 и Н7 расщепление НА0→НА1+НА2 происходит уже внутри инфицированных клеток котрансляционно в процессе синтеза молекулы НА0, и вирус выходит из клетки в инфекционной форме с расщепленным белком НА1+НА2. Разрезание НА0 таких вирусов осуществляется внутриклеточными убиквитарными протеазами фуринового ряда на мембранах аппарата Гольджи c высокой эффективностью, близкой к 100% молекул НА0 [37]. В результате интенсивного протеолиза НА0→НА1+НА2 синтезированные высоковирулентные вирусы легко проходят через межклеточные контакты и базальные мембраны в первичном инфекционном очаге респираторного и/или кишечного трактов, проникают в кровяное русло и вызывают генерализованную пантропную инфекцию макроорганизма [16, 33]. Низковирулентные вирусы с моноаргининовым сайтом в белке НА0 в силу расщепления только части молекул в инфицированной клетке и не столь эффективной активации инфекционности и фузионных свойств вирионов обладают низкой способностью преодоления защитных клеточных барьеров в организме-хозяине. Как правило, такие вирусные штаммы, к которым относятся низковирулентные вирусы гриппа птиц и сезонного гриппа людей, индуцируют локальный патологический процесс в первичном органе и не вызывают полиорганную диссеминацию вируса и генерализованную форму инфекционного процесса в пораженном организме [33, 37–39].
Факторы, регулирующие круг хозяев вируса
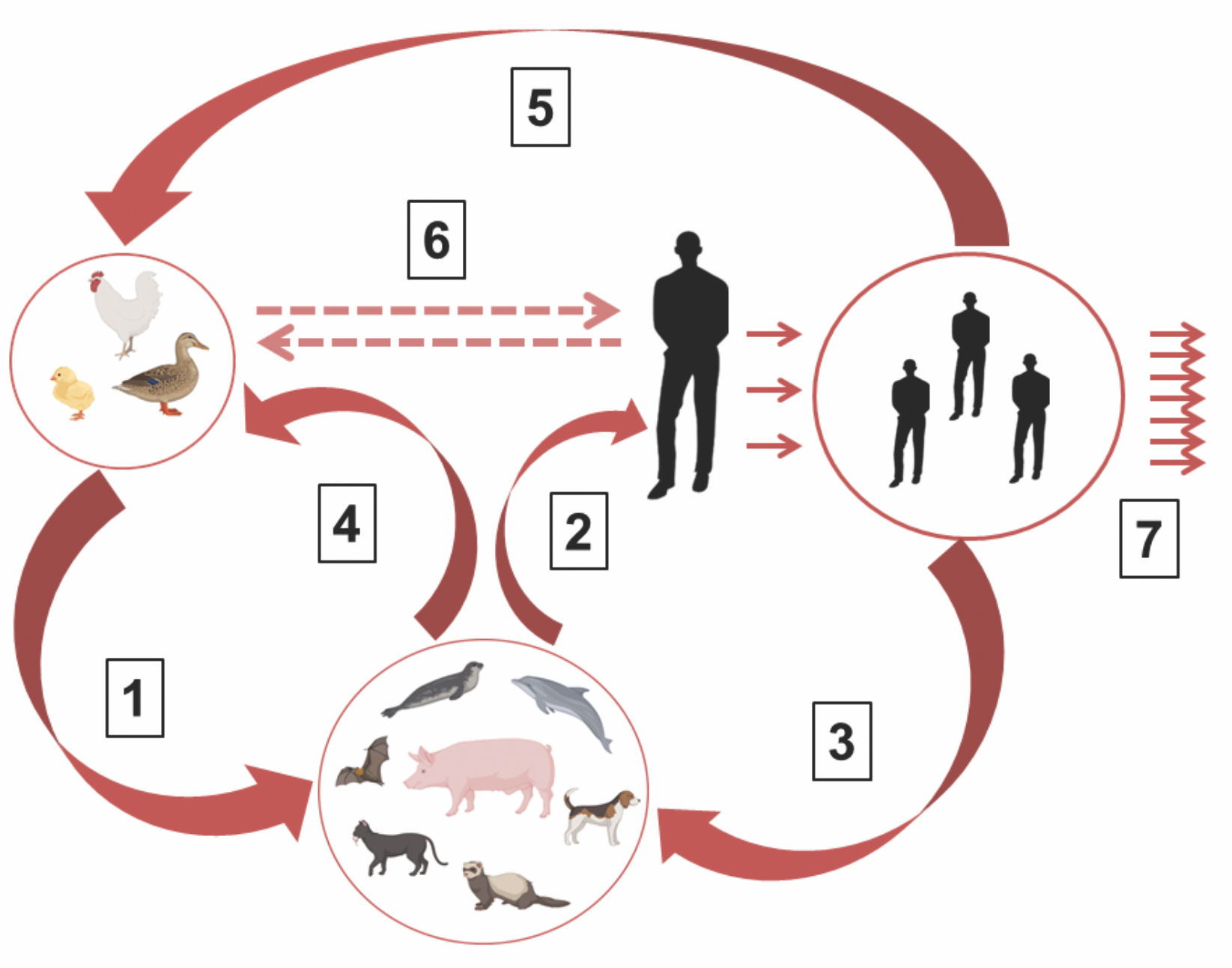
Важной особенностью вируса гриппа А является его свойство поражать широкий круг животных, включая птиц (основной природный резервуар вируса гриппа А) и человека (рис. 3). Вирус гриппа, будучи облигатным паразитом по отношению к клетке хозяина, взаимодействует с клеточными факторами и имеет для этой цели структурные паттерны в вирусных белках, так называемые генетические штампы (genetic signatures). Для успешного размножения эти структурные домены (штампы) должны быть конкордатными клеткам определенного вида хозяина. При смене хозяина вирусу необходимо эти паттерны изменить и адаптировать к новому виду хозяина.
Рис. 3. Этапы адаптации вируса птичьего гриппа H5N1 и возникновения пандемического варианта вируса.
Птичий вирус гриппа H5N1 способен непосредственно переходить на человека (прямой путь передачи) или опосредованно ‒ сначала на млекопитающих животных, таких как свиньи, пушные звери, хищные звери, тюлени и др., с последующим переходом на людей (непрямой путь передачи). В организме человека и млекопитающих животных птичий вирус претерпевает частичную адаптацию к организму человека посредством адаптационных мутаций либо реассортацией сегментов вирусной геномной РНК между вирусами гриппа птиц и человека. Частично адаптированный к человеку вирус птичьего гриппа H5N1 может возвращаться в популяцию птиц и животных и формировать природный резервуар перманентной потенциальной угрозы возникновения эпидемического вируса высокопатогенного вируса человека субтипов H5 и H7. Обозначения: 1 ‒ первичная адаптация вируса гриппа к человеку (мутации в генах NP, PB2, HA, NA и др.); 2 ‒ частичная адаптация вируса к человеку; 3 ‒ поддержание межвидовой передачи вируса в биосфере; 4 ‒ передача вируса от млекопитающих птицам; 5 ‒ поддержание межвидовой передачи в биосфере; 6 ‒ прямой путь передачи инфекции от птиц к человеку и обратно; 7 ‒ развитие эпидемического процесса.
Fig. 3. Adaptation stages of the H5N1 avian influenza virus and the emergence of a pandemic variant of the human influenza plague virus.
The H5N1 avian influenza virus can be transmitted directly to humans (direct transmission route) or indirectly, first to mammals such as pigs, fur-bearing animals, animals of prey, seals, etc., followed by an indirect transmission route to humans). In the human body (with direct transmission) and mammals, the avian virus undergoes partial adaptation to the human body through adaptive mutations or reassortation of viral genomic RNA segments between avian and human influenza viruses. The H5N1 avian influenza virus, partially adapted to humans, can return to the population of birds and mammals and form a natural reservoir of a permanent potential threat of the emergence of highly pathogenic epidemic virus (HPAV) subtypes H5 and H7 and its subsequent return to the human population. Designations: 1 ‒ Initial adaptation of influenza virus to humans (mutations in the NP, PB2, HA, NA genes and others); 2 ‒ Partial adaptation of virus to human; 3 ‒ Maintenance of interspecies virus transmissions in biosphere; 4 ‒ Virus transmission from mammals to birds; 5 ‒ Maintenance of interspecies transmissions in biosphere; 6 ‒ Direct transmission way from birds to humans and back; 7 ‒ Development of epidemic process.
В процессе репликации вируса гриппа А можно выделить 7 основных точек (стадий) взаимодействия с аппаратом клетки-хозяина:
В перечисленных стадиях участвуют различные вирусные белки, которые регулируют взаимодействие с клеткой, дающей в результате новое вирусное потомство, и обусловливают развитие инфекционного процесса в пораженном организме [38].
Таблица 2. Сравнение аминокислотных паттернов белка NP у вирусов гриппа А, изолированных от млекопитающих и птиц
Table 2. Comparison of amino acid patterns of NP protein in influenza A viruses isolated from mammals and birds
Субтип и происхождение вируса (вид штампа)1 Subtype and origin of the virus (type of signature)1 | Номера позиций аминокислот в генетическом штампе белка NP2 Numbers of amino acid positions in the genetic signature of the NP protein2 | Совпадения позиций в штампе NP вируса человека, % Matching positions in the human virus NP signature, % | Совпадения позиций в штампе NP вируса птиц, % Matching positions in the avian virus NP signature, % | ||||||||||||||
16 | 33 | 61 | 100 | 109 | 214 | 283 | 293 | 305 | 313 | 357 | 372 | 422 | 442 | 455 | |||
H1N1 человека (классический) H1N1 human (classic) | D | I | L | V | V | K | P | K | K | Y | K | D | K | A | E | 100 | 0 |
H5N1 норки H1N1 minks | G/D3 | I | I | R | V | R | L | R | R | F | Q | E | R | T | D | 13 | 87 |
H5N1 птичий H5N1 avian | G | V | I | R | I | R | L | R | R | F | Q | E | R | T | D | 0 | 100 |
Примечание. 1 ‒ первичные последовательности белка NP взяты из баз данных GenBank и GISAID по 10‒15 штаммов для каждой группы; 2 ‒ нумерация позиций аминокислот начинается с N-концевого метионина в молекуле белка NP (70 кДа; общая протяженность 498 аминокислот); 3 ‒ отмеченная позиция имеет гетерогенность в вирусных популяциях, в которых аминокислота D обнаруживается в 10‒20% вирусных изолятов.
Note. 1 ‒ primary NP protein sequences were taken from GenBank and GISAID databases with 10–15 strains for each group; 2 ‒ the numbering of amino acid positions begins with the N-terminal methionine in the NP protein molecule (70 kDa; the total sequence of 498 amino acids); 3 ‒ the marked position has heterogeneity in viral populations, in which the amino acid D is identified in 10‒20% of viral isolates.
В регуляцию тропизма и контагиозности вируса к хозяину наиболее ощутимый вклад вносят белки NP, PB2, HA, NA (табл. 2). Основной белок вирусного нуклеокапсида NP играет главную роль в преодолении врожденного иммунитета хозяина, обусловленного фактором Мх [40]. Белок NP вирусов гриппа птиц распознается ядерным фактором Мх человека, который связывает вирусный нуклеокапсид в полимерные комплексы и останавливает размножение вируса [41]. Напротив, NP вирусов человека не распознается человеческим фактором Мх; вирус ускользает от иммунного ответа человека и успешно поддерживает свою репликацию в его организме. Помимо распознавания фактора Мх, роль белка NP в регуляции круга хозяев обусловлена его взаимодействием с клеточными ядерными факторами ANP32A и ANP32B, участвующими во внутриядерном процессе ацетилирования гистонов и цитоплазматического транспорта кэпированных вирусных мРНК, а также в блокировании антивирусного митохондриального белка MAVS [42–45]; альфа-импортином, регулирующим транспорт вирусного РНП через ядерную пору в инфицированной клетке [46, 47]; клеточным фактором UAP56, участвующим в сплайсинге ядерных РНК [48]. Генетические штампы белка NP вирусов человека включают ключевые аминокислоты в позициях: 16/D; 33/I; 61/L; 100/I/V; 109/V; 214/K; 283/P; 293/K; 305/K; 313/Y; 357/K; 357/K; 372/D; 422/K; 442/A; 455/E; тогда как у птичьих вирусов гриппа этот аминокислотный штамп имеет явные отличия (табл. 3) [49–54]. Следовательно, в случае перехода птичьих вирусов гриппа от птиц к человеку аминокислотный штамп должен измениться и иметь алгоритм человека для преодоления барьера Мх и успешной репликации вирусного генома в клеточном ядре.
Таблица 3. Мутации, контролирующие круг хозяев вируса гриппа H5N1
Table 3. Mutations that control the host range of the H5N1 influenza virus
Вирусный ген/белок Viral gene/protein | Функции вирусного белка, определяющие связь с хозяином Viral protein functions that determine association with the host | Мутации, ведущие к адаптации у млекопитающих Mutations leading to adaptation in mammals |
NP | Преодолевает фактор МхА врожденного иммунитета человека Регулирует внутриядерный транспорт вирусного РНП через поры клеточного ядра Взаимодействует с ядерной РНК хеликазой UAP56, участвующей в сплайсинге и транспорте внутриядерных про-мРНК, а также блокирует антивирусный митохондриальный белок MAVS, взаимодействует с клеточными каспазами, факторами TRIM4, TRIF3, Rab11a, eEF1D и др. Overcomes the MxA factor of human innate immunity Regulates the intranuclear transport of viral RNP through the pores of the cell nucleus Interacts with nuclear RNA helicase UAP56, which is involved in the splicing and transport of intranuclear pro-mRNAs, and also blocks the antiviral mitochondrial protein MAVS, interacts with cellular caspases, factors TRIM4, TRIF3, Rab11a, eEF1D, etc. | 16/D; 33/I; 61/L; 100/I/V; 109/V; 214/K; 283/P; 293/K; 305/K; 313/Y; 357/K; 357/K; 372/D; 422/K; 442/A; 455/E |
PB2 | Распознает и гидролизует «кэпированные» клеточные мРНК при транскрипции вирусного генома в ядре клеток Взаимодействие с ядерными факторами ANP32A и ANP32B человека Recognizes and hydrolyzes “capped” cellular mRNAs during transcription of the viral genome in the cell nucleus Interacts with human nuclear factors ANP32A and ANP32B | 271/A; 526/K 590/S; 627/K; 701/N |
HA | Определяет адсорбцию на SA рецепторах 2‒6-го типа клеток человека Формирует функциональный комплекс с вирусным белком NA специфического г идролиза остатков SA 2‒6-го типа связи на поверхностных рецепторах клеток хозяина Регулирует контагиозность вируса Determines adsorption on SA receptors of human cell types 2‒6 Forms a functional complex with the viral protein NA for the specific hydrolysis of SA residues of type 2‒6 bonds on the surface receptors of host cells Regulates the contagiousness of the virus | 137/A; 155/T; 160/A; 190/D; 193/R; 225/D; 226/L; 228/S |
NA | Распознает и гидролизует остатки SA с типом связи α2-6 на клеточных рецепторах и в муцине респираторного секрета Участвует в регуляции контагиозности вируса Recognizes and hydrolyzes SA residues with the α2-6 bond type on cellular receptors and in respiratory mucin Participates in the regulation of virus contagiousness | Отсутствие делеции в ножке и 2-го рецепторного центра в NA/ No deletion in the stalk and the lack of a 2nd receptor center in the NA; Мутации/Mutations: 46/D; 74/S; 151/D; 163/L; 204/M; 369I; 396M; 420/G |
Примечание. Описание мутаций и соответствующие ссылки на публикации приведены в тексте статьи.
Note. Description of mutations and corresponding links to publications are given in the text of the article.
В кооперации с белком NP функционирует полимеразный белок РВ2 (табл. 3). У данного белка РВ2 для адаптации к организму человека и эффективного синтеза вирусных РНК важны позиции аминокислот T271A, E627K, D701N, которые отличаются у птичьих вирусов гриппа [42]. Замены аминокислот в этих позициях у вирусов птиц повышают сродство к клеточным факторам человека, что приводит к усилению репликации таких вирусных вариантов при инфекции человека [54, 55]. Точками взаимодействия с факторами хозяина у белков NP и РВ2 при выполнении своих вирусспецифических функций синтеза РНК служат стадии внутриядерной репликации генома вируса (см. выше описанные стадии 3–5).
Вторую важнейшую группу факторов адаптации к хозяину составляет тандем наружных рецепторных белков HA и NA вируса гриппа. Данные белки обусловливают устойчивость и распространение вируса во внешней среде и определяют специфичность распознавания рецепторов на поверхности эпителия на начальной стадии инфекции при инфицировании хозяина, а также клеточных гликопротеидов в зоне почкования на заключительном этапе сборки вновь синтезированных вирионов на поверхности инфицированных клеток. Данные белки функционируют в кооперации [56, 57], и существует реципрокный баланс их функций, играющий важную роль при адаптации вируса к хозяину [58, 59].
У вирусов гриппа птиц рецепторная и ферментативная гликозидазная функции белков HA и NA реализуются предпочтительно через взаимодействие с гликопротеидными рецепторами с типом связи α2-3 терминальных остатков сиаловой кислоты (СК) с остатком галактозы, которыми обогащены респираторный и кишечный тракт большинства птиц. У вирусов человека белковый тандем HA–NA имеет преимущественный аффинитет к гликопротеидам с терминальным остатком СК, связанным с галактозой связью α2-6 [60]. В такой рецепторной специализации для организма человека ключевыми позициями в генетических штампах для вирусов субтипа Н5 могут служить аминокислоты: 137/Ala, 155/Thr, 163/Ala, 190/Asp, 193/Arg, 225/Asp, 226/Leu, 228/Ser в белке НА [61, 62]. В белке NA птичьих вирусов важными аминокислотными позициями для адаптации к организму млекопитающих могут быть 46/Asp; 74/Ser; 151/Asp; 163/Leu; 204/Met; 369/Ile; 396/Met; 420/Gly [63, 64]. Дополнительно в молекуле NA вирусов гриппа птиц H5N1 идентифицирован характерный второй сайт рецепции сиаловых остатков α2-3 (так называемый второй центр гемадсорбции), тогда как у вирусов гриппа человека такой сайт отсутствовал, что специфически усиливало аффинитет NA к рецепторам типа α2-6 [65]. У вирусов H5N1, изолированных от норок в 2022 г., указанный центр в полноценном виде не обнаруживался, что сближало эти изоляты с вирусами человека и повышало их аффинитет к сиалорецепторам типа α2-6 [66]. Дополнительным сайтом усиления эпидемического потенциала для человека у птичьих вирусов H5N1 может служить делеция в гене NA и укорочение ножки NA, что часто обнаруживалось как у птичьих вирусов H5N1, изолированных от человека [67, 68], так и у пандемических вирусов гриппа человека [69, 70].
Отмеченные структурные и функциональные изменения в белках NP, PB2, HA, NA являются наиболее значимыми для адаптации и перехода вирусов Н5 и Н7 от птиц к человеку. Вместе с тем адаптивные изменения при смене хозяина могут отмечаться в других вирусных белках, таких как PB1, PA, PA-X, NS1, NEP, M1 и др. [71–76], которые можно рассматривать как дополнительные экстрагенные кофакторы, обеспечивающие смену вирусного хозяина наряду с описанными выше главными регуляторами круга хозяев вируса.
В заключении раздела следует отметить, что описанные адаптивные мутации генов вируса гриппа птиц необходимы для формирования двух главных фенотипических признаков эпидемического вируса людей: (1) свойство поддерживать высокий уровень размножения возникшего нового вирусного штамма в организме человека и (2) сохранение высокой воздушно-капельной контагиозности вируса в человеческой популяции после его перехода от птиц человеку. Формирование данных признаков вируса и его эпидемического потенциала для популяции людей контролируется главным образом 4 основными вирусными белками: NP, PB2, HA и NA.
Возможные пути перехода высоковирулентных вирусов Н5 и Н7 от птиц человеку
Возможны 2 пути перехода высоковирулентных вирусов гриппа от птиц к человеку (рис. 3). Первый путь – это прямая передача от птиц непосредственно человеку. Второй возможный путь – передача вируса через промежуточного хозяина, которым может быть организм млекопитающего.
Первый путь представляется затруднительным, поскольку еще неадаптированный птичий вирус должен приспособиться к новому организму – человеку, приобретая необходимые адаптационные мутации непосредственно в ходе самого процесса смены хозяина. Имеющиеся наблюдения нескольких эпизодов эпидемических вспышек высокопатогенного птичьего гриппа H5N1 и H7N9 согласуются с данным положением. Такие вспышки, произошедшие в Китае (1997), Гонконге (2005), Китае (2017) и ряде других районов мира, вызванные прямым вирусным заражением человека от птиц, носили локальный характер и не имели эпидемического развития [39]. Неадаптированный птичий вирус не обладал достаточной контагиозностью для людей и не вызывал процесса аэрозольной передачи инфекции от человека к человеку.
Второй возможный путь передачи вирусов субтипов Н5 и Н7 может проходить через промежуточных хозяев – популяцию млекопитающих, как правило, связанных с птицами цепочкой «хищник–жертва». В этом промежуточном организме, более близком генетически человеку, птичий вирус приобретет адаптацию к критическим механизмам организма-хозяина. Такая начальная предадаптация в организме млекопитающих может подготовить птичий вирус и значительно облегчить его переход в популяцию людей и дать начало формированию высоковирулентного гриппозного вируса субтипов Н5 и Н7. Важно отметить, что формирование в организме млекопитающих предадаптированного к человеку аппарата внутренних белков вируса (NP, PB2, PB1, M1, NS1), отвечающих за репликацию вируса, может дать начало не только эпидемии высоковирулентного вируса субтипов H5 и/или H7, но и способствовать возникновению нового антигенного типа эпидемии у людей, вызванной низковирулентными вирусами данных субтипов.
Предпосылки появления пандемии вирусов гриппа А
Эпидемическая ситуация последних 2–3 лет характеризуется резким ростом заболеваемости птиц и млекопитающих в природе и сельском хозяйстве высокопатогенным штаммом вируса гриппа H5N1, относящимся к клайду h2.3.4.4b [77–82]. Регистрируемый рост заболеваемости имеет три особенности.
- Подъем заболеваемости связан с появлением генетического клайда h2.3.4.4b высокопатогенного субтипа вируса птиц H5N1.
- Распространение данного штамма среди домашних и диких птиц носит планетарный характер и затрагивает все континенты. За последние 2 года суммарная смертность птиц и животных от этого вируса на планете приближается к 1 млрд особей [79, 83].
Рис. 4. Причины и последствия проникновения высоковирулентного вируса гриппа A/H5N1 в Северную Евразию (весна 2005 ‒ весна 2008) [5].
Fig. 4. Reasons and consequences of the penetration of the highly pathogenic influenza virus A/H5N1 into Northern Eurasia (spring 2005 ‒ spring 2008) [5].
Вирус субтипа Н5 появился в КНР в феврале 2003 г., т.е. в начале сезона весенней миграции. Он является реассортантом вирусов гриппа А диких птиц. В 2005 г. этот вирус был занесен на территорию России дикими птицами с образованием природных очагов (рис. 4) [5, 84–86]. В последние годы в РФ вспышки, связанные с заражением диких и домашних птиц вирусами A/H5N8, A/H5N1, A/H5N5, выявлены в Северной Осетии – Алании, Краснодарском крае, Астраханской и Томской областях. В 2020 г. во время вспышек птичьего гриппа H5N8 в Астраханской области были отмечены первые случаи заражения людей этим вирусом – на птицефабрике заразились 7 сотрудников. По данным Россельхознадзора, в 2022 г. в РФ были зарегистрированы 54 вспышки птичьего гриппа, из них 5 связаны с дикими птицами, 42 – с домашними, 7 – с птицеводческими хозяйствами. Заражение птиц выявлено в Магаданской, Сахалинской областях, в Хабаровском крае, Калужской, Ивановской, Орловской, Курской, Белгородской, Самарской, Челябинской областях.
- Подъем заболеваемости птиц высоковирулентным вирусом птиц H5N1 сопровождался участившимися и масштабными вспышками птичьего гриппа среди разных видов млекопитающих: морских львов, тюленей, норок, кошек, лис, медведей, песцов и др. (всего 26 видов млекопитающих [87, 88]). Наиболее значимые эпизоды вспышек H5N1 зарегистрированы среди тюленей в Южной и Северной Америке [89], норок в Испании [90], кошек в Польше [91], пушных зверей в Финляндии [92], морских львов в Перу [93]. Клинически вспышки сопровождались генерализованной мультиорганной инфекцией и высокой смертностью, а масштаб вспышек охватывал десятки тысяч особей (табл. 4).
Таблица 4. Смертельные случаи среди млекопитающих при вспышках высокопатогенного птичьего гриппа H5N1 в 2021‒2023 гг.
Table 4. Fatal cases among mammals during outbreaks of highly pathogenic avian influenza H5N1 occurred in 2021‒2023
Регионы в мире Regions in the world | Вид погибших животных Species of dead animals | Число погибших животных Number of dead animals |
Северная Америка North America | Тюлени Seals | > 121 |
Южная Америка South America | Морские львы Sea lions | > 634 |
Испания Spain | Норки Minks | 51 986 |
Польша Poland | Кошки Cats | > 98 |
Финляндия Finland | Пушные с/х животные Fur-bearing farm animals | > 200 000 |
Примечание. В таблице суммированы данные публикаций [84‒89].
Note. In the table, the data of references [84‒89] are summarized.
В перечисленных эпизоотиях была зарегистрирована передача вируса от животного к животному, что указывало на повышенную контагиозность вируса H5N1 для норок [87, 90, 92]. Это свойство вируса формируется как генами, обеспечивающими достаточный уровень репликации вируса в данном виде животного (или человека), так и генами, обеспечивающими рецепторную специфичность к клеточным рецепторам и стабильность вирусных частиц к факторам внешней и внутриклеточной среды. Важнейшую роль в приобретении вирионной устойчивости к таким факторам, как повышенная температура и рН внешней и внутриклеточной среды, играет конформация вирусного белка НА, и особенно структура его стеблевого домена [94–98]. Изолированные штаммы птичьего вируса H5N1 от тюленей, кошек характеризовались появлением ряда примечательных мутаций в генах NP, PB2, HA, NA. Обнаруженные мутации отражали частичную адаптацию исходного птичьего вируса к организму животных и имели сходство с генетическими штампами, характерными для вирусов гриппа человека. В частности, в полимеразном белке РВ2 обнаруживались наиболее характерные мутации Е627К и А701К; в белке NP – G16D, V/I100R, V/Y313F, K357Q; в белке НА была выявлена сцепленная двойная мутация 163Ala и 193Arg, которая специфична для вирусного белка НА, имеющего сродство к сиалосодержащим рецепторам с типом связи СК-α2-6-Гал [38].
В гене нейраминидазы NA вирусов H5N1, изолированных во время испанской вспышки среди норок в 2021 г., обнаруживались мутации Ser369Ile и Ile396Met во втором дополнительном сайте связывания рецепторов с типом СКα2-3 (так называемый «гемадсорбирующий центр № 2»), инактивирующие данный рецепторный центр [65]. Наличие рецепторного центра № 2 характерно только для вирусов гриппа птиц и отсутствует у вируса гриппа человека. Вполне очевидно, что утрата этого сайта в белке NA снижает аффинитет к сиалорецепторам СКα2-3 и повышает характерное для вирусов человека сродство к рецепторам СКα2-6 [38, 66]. Приведенные данные показывают, что высоковирулентные вирусы гриппа H5N1, вызывающие заболевания млекопитающих, приобрели ряд адаптивных мутаций, отличающих эти штаммы от вирусов птиц и сближающих с вирусом гриппа, тропного к организму человека. Таким образом, вирус птичьего гриппа H5N1 имеет смешанный (птичье-человеческий) штамп мутаций, показывающий его частичную предадаптацию к организму человека. Такая продолжающаяся предадаптация птичьего вируса в организме млекопитающих создает очевидные предпосылки его последующего облегченного перехода от млекопитающих на людей. Экспериментально показана такая возможность [95, 96].
Как отмечено выше, помимо адаптационных мутаций, переход птичьего вируса от одного вида хозяина к другому может проходить путем шифта, т.е. обмена сегментами геномных РНК с вирусами, уже адаптированными к человеку. Такой механизм возникновения пандемических вирусов гриппа А имел место ранее, в 1957, 1968 и 2009 гг., при возникновении и развитии эпидемий, вызванных вирусами H2N2, H3N2, H1N1/09pdm соответственно [11, 12]. Участившиеся в 2020–2022 гг. вспышки птичьего гриппа H5N1 у млекопитающих создают предпосылки для реализации аналогичного процесса для данного высоковирулентного субтипа H5N1 с учетом интенсивного обмена вирусами и большого количества бессимптомных форм гриппозной инфекции у людей, имеющих контакт с животными [99, 100]. Переход высоковирулентного субтипа H5N1 на млекопитающих увеличивает вероятность совместного инфицирования данных животных вирусами гриппа птиц и человека и последующего обмена между ними сегментами РНК, отвечающими за тропизм вируса к человеку, и тем самым создает реальную возможность возникновения химерного высоковирулентного вируса, который окажется способным преодолеть межтаксонный барьер от птиц к человеку.
Глобализация распространения «птичьего гриппа» в мире
С начала XXI в. отмечен волнообразный рост заболеваемости и распространения высоковирулентных вирусов гриппа птиц среди как домашних, так и диких птиц [79]. Проведенный нами мониторинг на территории Северной Евразии выявил существование природных очагов, связанных с биоценозами водного и околоводного комплексов, 15 из известных в мире антигенных 18 субтипов гриппа А, в том числе A/H5N1, с которым связана возникшая в 2003 г. обширная эпизоотия, а затем панзоотия среди диких и домашних птиц [19–21, 101–103]. В апреле 2005 г. во время весенней миграции птиц вирус H5N1 проник на территорию Западной Сибири с дальнейшим распространением в западном направлении. В апреле 2008 г. другая генетическая линия вируса попала на территорию Приморского края с дальнейшим охватом более северных территорий Дальнего Востока (рис. 4) [5]. Возникшие на огромных пространствах севера Евразии природные очаги разных вариантов вируса A/H5N1 и других вирусов гриппа птиц продолжают активно функционировать и в настоящее время. Современные пути распространения высоковирулентных штаммов вируса птичьего гриппа Н5 (клайд h.2.3.4.4b) также совпадают с установленными ранее миграционными руслами птиц [83, 88, 101, 104]. Эти наблюдения подтверждают общую концепцию формирования стойких природных очагов слабовирулентного вируса Н5, наряду с другими антигенными субтипами вируса гриппа А, в Юго-Восточной Азии и Сибирском и Дальневосточном регионах России, которые служат природным пулом и источником для возникновения высоковирулентных штаммов и их последующего внутри- и межконтинентального распространения, создающего перманентную угрозу опасных пандемий среди млекопитающих и людей (рис. 4). Данная концепция сформировалась на основе обширных фундаментальных исследований экологии вирусных популяций [8, 19–21, 26–29, 101–103].
Как уже отмечалось выше, в период 2021–2023 гг. имели место масштабный подъем заболеваемости птичьим гриппом и глобальное распространение высоковирулентного вируса H5N1 (клайд h2.3.4.4b) среди птиц на всех континентах планеты [78–82, 88]. Как показывает генетический анализ вирусных геномов [105], установившийся в природе реципрокный обмен высоковирулентным вирусом H5N1 между млекопитающими и птицами привел к распространению и накоплению в природе вирусных вариантов, имеющих признаки частичной адаптации к млекопитающим, включая человека. Также вызывает тревогу и пристальное внимание тот факт, что глобальный подъем заболеваемости птиц сопровождается участившимися вспышками высоковирулентного птичьего гриппа H5N1 среди млекопитающих. В период 2021–2023 гг. зарегистрированы 5 масштабных эпизоотий опасного гриппа на 3 континентах: среди тюленей в Северной Америке, среди норок в Испании, среди кошек в Польше, среди пушных зверей на фермах Финляндии и морских львов в Перу [87–93]. Масштабы вспышек среди животных суммированы в табл. 4. Вирусы гриппа H5N1, изолированные от животных при этих вспышках, имели часть генетических маркеров и фенотипических признаков высокой вирулентности, характерных для вирусов гриппа человека и млекопитающих. Признаки высокой вирулентности птичьих вирусов H5N1 для млекопитающих проявлялись в тяжелом клиническом течении болезни, которая сопровождалась высокой смертностью, достигающей 50–60%, развитием первичной вирусной пневмонии, фатальных симптомов тотального геморрагического воспаления легких (т.н. легочное опеченение), поражением нервной системы с тяжелыми нарушениями моторных функций и параличами, массивного геморрагического поражения кишечника, тяжелой общей интоксикаций и т.п. [90, 92, 93].
Обнаруженное широкое распространение среди млекопитающих, имеющих тесный контакт с человеком, частично предадаптированного к человеку вируса субтипа H5N1 создает реальную угрозу его облегченного перехода на человека с эпидемическим потенциалом за счет респираторного пути передачи среди людей. Дополнительно такой предадаптированный у животных птичий вирус H5N1 может быстро усилить и окончательно сформировать свой эпидемический потенциал и контагиозность путем реассортации сегментами геномной РНК (главным образом NP, PB2, HA, NA) с вирусами гриппа человека либо уже при переходе на организм человека, либо еще в организме млекопитающих и птиц, имеющих тесный контакт с человеком (свиньи, утки и другие с/х и домашние животные). У свиней и уток инфекция, вызванная высоковирулентным вирусом птиц H5N1, может протекать в субклинической форме без выраженных симптомов, что может благоприятствовать возникновению у них смешанного инфицирования вирусами человека и птиц и реализации процесса реассортации их геномов [106–108]. По информации Avian Influenza Weekly update № 919 от 03 октября 2023 г., с января 2003 г. по настоящее время в 23 странах зарегистрировано 878 случаев заражения людей вирусом H5N1 при 50% летальности, 1568 случаев инфицирования вирусом A/H7N9 при летальности 39%, 88 и 90 случаев заражения A/H5N6 и A/H9N2 соответственно при 39% летальности, а также единичные случаи инфекции, вызванной вирусами субтипов A/H13N8, A/H7N4, A/H10N3 [88].
Меры предупреждения и готовности при угрозе «птичьего гриппа»
Возрастающая угроза человечеству вследствие резкого подъема заболеваемости и распространения высоковирулентного вируса птичьего гриппа среди животных обусловливает повышенное внимание к проблеме. В этой связи три организации: Продовольственная и сельскохозяйственная Организация Объединенных Наций (The Food and Agriculture Organization of the United Nations, FAO), Всемирная организация здравоохранения (The World Health Organization, WHO), Всемирная организация охраны здоровья животных (The World Organization for Animal Health, WOAH) обратились с совместным заявлением, в котором указано, что «продолжающиеся вспышки птичьего гриппа среди животных представляют опасность для человека» [87, 88]. В этом заявлении сформулирована общая повестка вопросов, требующих координации со стороны государственных органов на этапе ожидания и подготовки к возможной опасной эпидемии «птичьего гриппа». В частности, было рекомендовано обратить внимание на высоковирулентный птичий грипп как на источник риска для здоровья человека, усилить и регламентировать меры профилактики, включая меры биологической безопасности на фермерских хозяйствах и на этапах производственно-сбытовой цепи птицеводства, проведение эпидемиологических расследований по фактам вспышек, обеспечение оперативной передачи и обмена данными о геноме вирусов и поощрение сотрудничества между секторами охраны здоровья животных и человека.
Конкретизируя меры предупреждения и необходимой готовности в условиях масштабной угрозы эпидемии «птичьего гриппа», предлагаются следующие главные направления действий:
Предлагаемые меры позволят прогнозировать начало опасных пандемий и их риски, а также минимизировать последствия в случае возникновения эпидемий, вызванных вирусами гриппа птиц.
Заключение
На современном этапе отмечен глобальный подъем инфицирования и заболеваемости диких и домашних птиц высоковирулентным вирусом птичьего гриппа H5N1 (клайд h2.3.4.4b) и другими вирусами гриппа А. На этом фоне участились вспышки среди млекопитающих, вызванные данным клайдом вируса, и продолжается в природе циркуляция высоковирулентного вируса гриппа H5N1 и ряда других вирусов гриппа птиц, имеющих признаки частичной адаптации к организму человека. Переход высоковирулентного вируса H5N1 на млекопитающих создает реальную угрозу его дальнейшей адаптации к организму человека и повышению его эпидемического потенциала с последующим формированием пандемической ситуации. Складывающаяся предэпидемическая ситуация обусловливает необходимость выработки стратегии предупредительных мер для минимизации последствий возможного угрожающего сценария возникновения пандемии. Для снижения рисков и последствий такого развития событий в статье указаны неотложные меры, включая формирование концепции эффективной патогенетической терапии.
_______
*Фраза «По ком звонит колокол» заимствована из романа Эрнеста Хемингуэя, имеющего в английском оригинале название «For whom the bell tolls».
Примечание: Работа была представлена на международном конгрессе «Молекулярная диагностика и биобезопасность – 2024» (Москва, 16‒17 апреля 2024 г.): Жирнов О.П., Львов Д.К. Угроза пандемии птичьего гриппа: механизмы формирования популяционного генофонда вируса. Сборник тезисов под ред. акад. РАН В.Г. Акимкина, М., 2024, стр. 69‒70.
Об авторах
Олег Петрович Жирнов
Институт вирусологии имени Д.И. Ивановского ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи», Минздрава России; Русско-немецкая академия медико-социальных и биотехнологических наук, Инновационный центр Сколково
Email: zhirnov@inbox.ru
ORCID iD: 0000-0002-3192-8405
чл.-корр. РАН, доктор биол. наук, профессор, руководитель лаборатории вирусного патогенеза
Россия, 123098, Москва; 109029, МоскваДмитрий Константинович Львов
Институт вирусологии имени Д.И. Ивановского ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи», Минздрава России
Автор, ответственный за переписку.
Email: dk_lvov@mail.ru
ORCID iD: 0000-0001-8176-6582
академик РАН, доктор мед. наук, профессор, главный научный сотрудник
Россия, 123098, МоскваСписок литературы
- Walker P.J., Siddell S.G., Lefkowitz E.J., Mushegian A.R., Adriaenssens E.M., Alfenas-Zerbini P., et al. Recent changes to virus taxonomy ratified by the International Committee on Taxonomy of Viruses (2022). Arch. Virol. 2022; 167(11): 2429–40. https://doi.org/10.1007/s00705-022-05516-5
- Klemm C., Boergeling Y., Ludwig S., Ehrhardt C. Immunomodulatory Nonstructural Proteins of Influenza A Viruses. Trends. Microbiol. 2018; 26(7): 624–36. https://doi.org/10.1016/j.tim.2017.12.006
- Abdelwhab E.M., Mettenleiter T.C. Zoonotic Animal Influenza Virus and Potential Mixing Vessel Hosts. Viruses. 2023; 15(4): 980. https://doi.org/10.3390/v15040980
- Sanjuán R., Domingo-Calap P. Mechanisms of viral mutation. Cell Mol. Life Sci. 2016; 73(23): 4433–48. https://doi.org/10.1007/s00018-016-2299-6
- Львов Д.К., Гулюкин М.Ю., Забережный А.Д., Гулюкин А.М. Формирование популяционного генофонда потенциально угрожающих биобезопасности зоонозных вирусов. Вопросы вирусологии. 2020; 65(5): 243–58. https://doi.org/10.36233/0507-4088-2020-65-5-1 https://elibrary.ru/kprmam
- Brüssow H. The beginning and ending of a respiratory viral pandemic-lessons from the Spanish flu. Microb. Biotechnol. 2022; 15(5): 1301–17. https://doi.org/10.1111/1751-7915.14053
- Romero-Tejeda A., Capua I. Virus-specific factors associated with zoonotic and pandemic potential. Influenza Other Respir. Viruses. 2013; 7(Suppl. 2): 4–14. https://doi.org/10.1111/irv.12075
- Lvov D.K. Circulation of Influenza viruses in natural biocenosis. In: Viruses and Environment. Academic Press; 1978; 18: 351–80.
- Lvov D.K., Zhdanov V.M. Circulation of influenza viruses genes in the biosphere. Sov. Med. Rev. Virol. 1987; (1): 129–52.
- Lvov D.K. Influenza A viruses – a sum of populations with a common protected gene pool. Sov. Med. Rev. Virol. 1987; (2): 15–37.
- Palese P., Shaw M.L. Orthomyxoviridae: the viruses and their replication. In: Knipe D.M., Howley P.M., eds. Fields Virology. Lippincott Williams & Wilkins; 2007: 1648–89.
- Wille M., Holmes E.C. The ecology and evolution of influenza viruses. Cold Spring Harb. Perspect. Med. 2020; 10(7): a038489. https://doi.org/10.1101/cshperspect.a038489
- Suarez D.L. Evolution of avian influenza viruses. Vet. Microbiol. 2000; 74(1-2): 15–27. https://doi.org/10.1016/s0378-1135(00)00161-9
- Swayne D.E. Changing face of avian influenza ecology and its control : from wild birds to poultry and back again. In: Abstract Book of the 15th World Veterinary Poultry Congress. Beiging; 2007: 98–104.
- Krammer F., Smith G.J.D., Fouchier R.A.M., Peiris M., Kedzierska K., Doherty P.C., et al. Influenza. Nat. Rev. Dis. Primers. 2018; 4(1): 3. https://doi.org/10.1038/s41572-018-0002-y
- Garten W., Klenk H.D. Cleavage activation of the influenza virus hemagglutinin and its role in pathogenesis. In: Klenk H.D., Matrosovich M.N., Stech J., eds. Avian Influenza. Basel: Karger; 2008: 156–67.
- Львов Д.К., Ильичев В.Д. Миграции птиц и перенос возбудителей инфекций. М.: Наука; 1979.
- Львов Д.К., Жданов В.М. Персистенция генов эпидемических вирусов гриппа А в природных популяциях. Успехи современной биологии. 1982; 93(3): 323–37.
- Львов Д.К., Щелканов М.Ю. Птичий грипп H5N1. В кн.: Д.К. Львов (ред.): Вирусы и вирусные инфекции человека и животных. Руководство по вирусологии. М.: МИА; 2013: 554–77. https://elibrary.ru/tlzmhf
- Львов Д.К., Щелканов М.Ю., Алипер Т.И. Грипп диких птиц. В кн.: Львов Д.К., ред. Вирусы и вирусные инфекции человека и животных. Руководство по вирусологии. М.: МИА; 2013: 1086–94.
- Lvov D.K., Kaverin N.V. Avian influenza in Northern Eurasia. In: Klenk H.D., Matrosovich M.N., eds. Avian Influenza. Basel: Karger; 2008: 41–58.
- Львов Д.К. Возможное значение природных биоценозов в изменчивости вируса гриппа А. Вопросы вирусологии. 1974; 19(6): 740–4.
- Stallknect D., Brown J.D. Ecology of avian influenza in wild birds. In: Swayne D.E., ed. Avian Influenza. Oxford: Blackwell Publ.; 2008: 43–8.
- Pereira H.G., Tůmová B., Law V.G. Avian influenza A viruses. Bull. World Health Organ. 1965; 32(6): 855–60.
- Becker W.B. The isolation and classification of Tern virus: influenza A-Tern South Africa – 1961. J. Hyg. (Lond.). 1966; 64(3): 309–20. https://doi.org/10.1017/s0022172400040596
- Laver W.G., Webster R.G. Ecology of influenza viruses in lower mammals and birds. Br. Med. Bull. 1979; 35(1): 29–33. https://doi.org/10.1093/oxfordjournals.bmb.a071537
- Webster R.G., Laver W.G. Further evidence to support a recommendational events in the origin of new pandemic influenza viruses. In: Kilbourne E., ed. The Influenza. NY-San-Francisco: Academic Press; 1975; (7): 299–308.
- Львов Д.К. Популяционные взаимодействия в биологической системе: вирус гриппа а – дикие и домашние птицы – люди; причины и последствия проникновения на территорию России высоко вирулентного вируса гриппа A/H5N1. Журнал микробиологии эпидемиологии и иммунобиологии. 2006; 93(3): 96–100. https://elibrary.ru/htqbwt
- Львов Д.К., Альховский С.Д. 55 лет отделу экологии вирусов с научно-практическим центром по экологии и эпидемиологии гриппа (Институт вирусологии им. Д.И. Ивановского ФГБУ «НИЦЭМ им. Н.Ф. Гамалеи» Минздрава России). Вопросы вирусологии. 2024; 69(1): 7–21. https://doi.org/10.36233/0507-4088-217 https://elibrary.ru/xdikxk
- Böttcher-Friebertshäuser E., Garten W., Matrosovich M., Klenk H.D. The hemagglutinin: a determinant of pathogenicity. Curr. Top. Microbiol. Immunol. 2014; 385: 3–34. https://doi.org/10.1007/82_2014_384
- Klenk H.D., Rott R., Orlich M., Blödorn J. Activation of influenza A viruses by trypsin treatment. Virology. 1975; 68(2): 426–39. https://doi.org/10.1016/0042-6822(75)90284-6
- Lazarowitz S.G., Choppin P.W. Enhancement of the infectivity of influenza A and B viruses by proteolytic cleavage of the hemagglutinin polypeptide. Virology. 1975; 68(2): 440–54. https://doi.org/10.1016/0042-6822(75)90285-8
- Жирнов О.П. Феномен протеолитической активации миксовирусов и новая стратегия в лечении вирусных заболеваний. Вопросы вирусологии. 1983; 28(4): 9–21.
- Zhirnov O.P., Klenk H.D., Wright P.F. Aprotinin and similar protease inhibitors as drugs against influenza. Antiviral. Res. 2011; 92(1): 27–36. https://doi.org/10.1016/j.antiviral.2011.07.014
- Zhirnov O.P., Ikizler M.R., Wright P.F. Cleavage of influenza a virus hemagglutinin in human respiratory epithelium is cell associated and sensitive to exogenous antiproteases. J. Virol. 2002; 76(17): 8682–9. https://doi.org/10.1128/jvi.76.17.8682-8689.2002
- Zhirnov O.P., Matrosovich T.Y., Matrosovich M.N., Klenk H.D. Aprotinin, a protease inhibitor, suppresses proteolytic activation of pandemic H1N1v influenza virus. Antivir. Chem. Chemother. 2011; 21(4): 169–74. https://doi.org/10.3851/IMP1715
- Garten W., Hallenberger S., Ortmann D., Schäfer W., Vey M., Angliker H., et al. Processing of viral glycoproteins by the subtilisin-like endoprotease furin and its inhibition by specific peptidylchloroalkylketones. Biochimie. 1994; 76(3-4): 217–25. https://doi.org/10.1016/0300-9084(94)90149-x
- Chauhan R.P., Gordon M.L. An overview of influenza A virus genes, protein functions, and replication cycle highlighting important updates. Virus Genes. 2022; 58(4): 255–69. https://doi.org/10.1007/s11262-022-01904-w
- Lai S., Qin Y., Cowling B.J., Ren X., Wardrop N.A., Gilbert M., et al. Global epidemiology of avian influenza A H5N1 virus infection in humans, 1997–2015: a systematic review of individual case data. Lancet Infect. Dis. 2016; 16(7): e108–18. https://doi.org/10.1016/S1473-3099(16)00153-5
- Haller O., Kochs G. Mx genes: host determinants controlling influenza virus infection and trans-species transmission. Hum. Genet. 2020; 139(6–7): 695–705. https://doi.org/10.1007/s00439-019-02092-8
- Turan K., Mibayashi M., Sugiyama K., Saito S., Numajiri A., Nagata K. Nuclear MxA proteins form a complex with influenza virus NP and inhibit the transcription of the engineered influenza virus genome. Nucleic. Acids Res. 2004; 32(2): 643–52. https://doi.org/10.1093/nar/gkh192
- Peacock T.P., Sheppard C.M., Lister M.G., Staller E., Frise R., Swann O.C., et al. Mammalian ANP32A and ANP32B proteins drive differential polymerase adaptations in avian influenza virus. J. Virol. 2023; 97(5): e0021323. https://doi.org/10.1128/jvi.00213-23
- Sheppard C.M., Goldhill D.H., Swann O.C., Staller E., Penn R., Platt O.K., et al. An influenza A virus can evolve to use human ANP32E through altering polymerase dimerization. Nat. Commun. 2023; 14(1): 6135. https://doi.org/10.1038/s41467-023-41308-4
- Tome-Amat J., Ramos I., Amanor F., Fernández-Sesma A., Ashour J. Influenza A virus utilizes low-affinity, high-avidity interactions with the nuclear import machinery to ensure infection and immune evasion. J. Virol. 2018; 93(1): e01046–18. https://doi.org/10.1128/JVI.01046-18
- Zhang B., Xu S., Liu M., Wei Y., Wang Q., Shen W., et al. The nucleoprotein of influenza A virus inhibits the innate immune response by inducing mitophagy. Autophagy. 2023; 19(7): 1916–33. https://doi.org/10.1080/15548627.2022.2162798
- Ninpan K., Suptawiwat O., Boonarkart C., Phuangphung P., Sathirareuangchai S., Uiprasertkul M., et al. Expression of importin-α isoforms in human nasal mucosa: implication for adaptation of avian influenza A viruses to human host. Virol. J. 2016; 13: 90. https://doi.org/10.1186/s12985-016-0546-y
- Morris A.K., Wang Z., Ivey A.L., Xie Y., Hill P.S., Schey K.L., et al. Cellular mRNA export factor UAP56 recognizes nucleic acid binding site of influenza virus NP protein. Biochem. Biophys. Res. Commun. 2020; 525(2): 259–64. https://doi.org/10.1016/j.bbrc.2020.02.059
- Mänz B., Dornfeld D., Götz V., Zell R., Zimmermann P., Haller O., et al. Pandemic influenza A viruses escape from restriction by human MxA through adaptive mutations in the nucleoprotein. PLoS Pathog. 2013; 9(3): e1003279. https://doi.org/10.1371/journal.ppat.1003279
- Zhirnov O.P. The host origin of influenza A viruses can be assessed by the intracellular cleavage of the viral nucleocapsid protein. Brief report. Arch. Virol. 1988; 99(3-4): 277–84. https://doi.org/10.1007/BF01311077
- Zhirnov O., Bukrinskaya A.G. Nucleoproteins of animal influenza viruses, in contrast to those of human strains, are not cleaved in infected cells. J. Gen. Virol. 1984; 65 (Pt. 6): 1127–34. https://doi.org/10.1099/0022-1317-65-6-1127
- Finkelstein D.B., Mukatira S., Mehta P.K., Obenauer J.C., Su X., Webster R.G., et al. Persistent host markers in pandemic and H5N1 influenza viruses. J. Virol. 2007; 81(19): 10292–9. https://doi.org/10.1128/JVI.00921-07
- Worobey M., Han G.Z., Rambaut A. Genesis and pathogenesis of the 1918 pandemic H1N1 influenza A virus. Proc. Natl. Acad. Sci. USA. 2014; 111(22): 8107–12. https://doi.org/10.1073/pnas.1324197111
- Chen G.W., Gong Y.N., Shih S.R. Influenza A virus plasticity-A temporal analysis of species-associated genomic signatures. J. Formos. Med. Assoc. 2015; 114(5): 456–63. https://doi.org/10.1016/j.jfma.2015.01.015
- Long J.S., Idoko-Akoh A., Mistry B., Goldhill D., Staller E., Schreyer J., et al. Species-specific differences in use of ANP32 proteins by influenza A virus. Elife. 2019; 8: e45066. https://doi.org/10.7554/eLife.45066
- Subbarao E.K., London W., Murphy B.R. A single amino acid in the PB2 gene of influenza A virus is a determinant of host range. J. Virol. 1993; 67(4): 1761–4. https://doi.org/10.1128/jvi.67.4.1761-1764.1993
- Mitnaul L.J., Matrosovich M.N., Castrucci M.R., Tuzikov A.B., Bovin N.V., Kobasa D., et al. Balanced hemagglutinin and neuraminidase activities are critical for efficient replication of influenza A virus. J. Virol. 2000; 74(13): 6015–20. https://doi.org/10.1128/jvi.74.13.6015-6020.2000
- Kaverin N.V., Matrosovich M.N., Gambaryan A.S., Rudneva I.A., Shilov A.A., Varich N.L., et al. Intergenic HA-NA interactions in influenza A virus: postreassortment substitutions of charged amino acid in the hemagglutinin of different subtypes. Virus Res. 2000; 66(2): 123–9. https://doi.org/10.1016/s0168-1702(99)00131-8
- Wagner R., Matrosovich M., Klenk H.D. Functional balance between haemagglutinin and neuraminidase in influenza virus infections. Rev. Med. Virol. 2002; 12(3): 159–66. https://doi.org/10.1002/rmv.352
- Гамбарян А.С., Матросович М.Н. Какие адаптивные изменения в гемагглютинине и нейраминидазе необходимы для возникновения вируса пандемического гриппа из его птичьего предшественника? Биохимия. 2015; 80(7): 872–80. https://doi.org/10.1134/S000629791507007X
- Eggink D., Spronken M., van der Woude R., Buzink J., Broszeit F., McBride R., et al. Phenotypic effects of substitutions within the receptor binding site of highly pathogenic avian influenza H5N1 virus observed during human infection. J. Virol. 2020; 94(13): e00195-20. https://doi.org/10.1128/JVI.00195-20
- Guo H., de Vries E., McBride R., Dekkers J., Peng W., Bouwman K.M., et al. Highly pathogenic influenza A (H5Nx) viruses with altered H5 receptor-binding specificity. Emerg. Infect. Dis. 2017; 23(2): 220–31. https://doi.org/10.3201/eid2302.161072
- Gao R., Gu M., Liu K., Li Q., Li J., Shi L., et al. T160A mutation-induced deglycosylation at site 158 in hemagglutinin is a critical determinant of the dual receptor binding properties of clade 2.3.4.4 H5NX subtype avian influenza viruses. Vet. Microbiol. 2018; 217: 158–66. https://doi.org/10.1016/j.vetmic.2018.03.018
- Leguia M., Garcia-Glaessner A., Muñoz-Saavedra B., Juarez D., Barrera P., Calvo-Mac C., et al. Highly pathogenic avian influenza A (H5N1) in marine mammals and seabirds in Peru. Nat. Commun. 2023; 14(1): 5489. https://doi.org/10.1038/s41467-023-41182-0
- Scheibner D., Salaheldin A.H., Bagato O., Zaeck L.M., Mostafa A., Blohm U., et al. Phenotypic effects of mutations observed in the neuraminidase of human origin H5N1 influenza A viruses. PLoS Pathog. 2023; 19(2): e1011135. https://doi.org/10.1371/journal.ppat.1011135
- Du W., de Vries E., van Kuppeveld F.J.M., Matrosovich M., de Haan C.A.M. Second sialic acid-binding site of influenza A virus neuraminidase: binding receptors for efficient release. FEBS J. 2021; 288(19): 5598–612. https://doi.org/10.1111/febs.15668
- de Vries E., de Haan C.A. Letter to the editor: Highly pathogenic influenza A(H5N1) viruses in farmed mink outbreak contain a disrupted second sialic acid binding site in neuraminidase, similar to human influenza A viruses. Euro Surveill. 2023; 28(7): 2300085. https://doi.org/10.2807/1560-7917.ES.2023.28.7.2300085
- Bender C., Hall H., Huang J., Klimov A., Cox N., Hay A., et al. Characterization of the surface proteins of influenza A (H5N1) viruses isolated from humans in 1997-1998. Virology. 1999; 254(1): 115–23. https://doi.org/10.1006/viro.1998.9529
- Zhou H., Yu Z., Hu Y., Tu J., Zou W., Peng Y., et al. The special neuraminidase stalk-motif responsible for increased virulence and pathogenesis of H5N1 influenza A virus. PLoS One. 2009; 4(7): e6277. https://doi.org/10.1371/journal.pone.0006277
- Zhirnov O.P., Vorobjeva I.V., Saphonova O.A., Poyarkov S.V., Ovcharenko A.V., Anhlan D., et al. Structural and evolutionary characteristics of HA, NA, NS and M genes of clinical influenza A/H3N2 viruses passaged in human and canine cells. J. Clin. Virol. 2009; 45(4): 322–33. https://doi.org/10.1016/j.jcv.2009.05.030
- Young S.G., Kitchen A., Kayali G., Carrel M. Unlocking pandemic potential: prevalence and spatial patterns of key substitutions in avian influenza H5N1 in Egyptian isolates. BMC Infect. Dis. 2018; 18(1): 314. https://doi.org/10.1186/s12879-018-3222-6
- Nogales A., Villamayor L., Utrilla-Trigo S., Ortego J., Martinez-Sobrido L., DeDiego M.L. Natural selection of H5N1 avian influenza A viruses with increased PA-X and NS1 shutoff activity. Viruses. 2021; 13(9): 1760. https://doi.org/10.3390/v13091760
- Taft A.S., Ozawa M., Fitch A., Depasse J.V., Halfmann P.J., Hill-Batorski L., et al. Identification of mammalian-adapting mutations in the polymerase complex of an avian H5N1 influenza virus. Nat. Commun. 2015; 6: 7491. https://doi.org/10.1038/ncomms8491
- Elgendy E.M., Arai Y., Kawashita N., Daidoji T., Takagi T., Ibrahim M.S., et al. Identification of polymerase gene mutations that affect viral replication in H5N1 influenza viruses isolated from pigeons. J. Gen. Virol. 2017; 98(1): 6–17. https://doi.org/10.1099/jgv.0.000674
- Wang C., Qu R., Zong Y., Qin C., Liu L., Gao X., et al. Enhanced stability of M1 protein mediated by a phospho-resistant mutation promotes the replication of prevailing avian influenza virus in mammals. PLoS Pathog. 2022; 18(7): e1010645. https://doi.org/10.1371/journal.ppat.1010645
- Cheung P.H., Lee T.T., Chan C.P., Jin D.Y. Influenza A virus PB1-F2 protein: An ambivalent innate immune modulator and virulence factor. J. Leukoc. Biol. 2020; 107(5): 763–71. https://doi.org/10.1002/JLB.4MR0320-206R
- Rashid F., Xie Z., Li M., Xie Z., Luo S., Xie L. Roles and functions of IAV proteins in host immune evasion. Front. Immunol. 2023; 14: 1323560. https://doi.org/10.3389/fimmu.2023.1323560
- WHO. Cumulative number of confirmed human cases for avian influenza A(H5N1) reported to WHO, 2003–2020. Available at: https://www.who.int/influenza/human_animal_interface/2020_10_07_tableH5N1.pdf
- Lewis N.S., Banyard A.C., Whittard E., Karibayev T., Al Kafagi T., Chvala I., et al. Emergence and spread of novel H5N8, H5N5 and H5N1 clade 2.3.4.4 highly pathogenic avian influenza in 2020. Emerg. Microbes. Infect. 2021; 10(1): 148–51. https://doi.org/10.1080/22221751.2021.1872355
- Shi J., Zeng X., Cui P., Yan C., Chen H. Alarming situation of emerging H5 and H7 avian influenza and effective control strategies. Emerg. Microbes. Infect. 2023; 12(1): 2155072. https://doi.org/10.1080/22221751.2022.2155072.
- Sobolev I., Sharshov K., Dubovitskiy N., Kurskaya O., Alekseev A., Leonov S., et al. Highly pathogenic avian influenza A(H5N8) virus clade 2.3.4.4b, Western Siberia, Russia, 2020. Emerg. Infect. Dis. 2021; 27(8): 2224–7. https://doi.org/10.3201/eid2708.204969
- Tian J., Bai X., Li M., Zeng X., Xu J., Li P., et al. Highly Pathogenic Avian Influenza Virus (H5N1) Clade 2.3.4.4b Introduced by Wild Birds, China, 2021. Emerg. Infect. Dis. 2023; 29(7): 1367–75. https://doi.org/10.3201/eid2907.221149
- Isoda N., Onuma M., Hiono T., Sobolev I., Lim H.Y., Nabeshima K., et al. Detection of new H5N1 high pathogenicity avian influenza viruses in winter 2021-2022 in the Far East, which are genetically close to those in Europe. Viruses. 2022; 14(10): 2168. https://doi.org/10.3390/v14102168
- Adlhoch C., Fusaro A., Gonzales J.L., Kuiken T., Mirinavičiūtė G., Niqueux É., et al. Avian influenza overview June-September 2023. EFSA J. 2023; 21(10): e08328. https://doi.org/10.2903/j.efsa.2023.8328
- WHO (Western Pacific Ocean Region). Human infection with avian influenza A/H5 viruses. Human infection with avian influenza A(H5N1) virus. Wkly Update Number. 852; 2022.
- WHO (Western Pacific Ocean Region). Human infection with avian influenza A(H5) viruses. Human infection with avian influenza A(H5N1) virus. Avian Influ. Wkly Update Number. 921 (Pt. 1); 2023.
- Львов Д.К., Борисевич С.В., Альховский С.В., Бурцева Е.И. Актуальные подходы к анализу вирусных геномов в интересах биобезопасности. Инфекционные болезни: новости, мнения, обучение. 2019; 8(2): 96–101. https://doi.org/10.24411/2305-3496-2019-12012 https://elibrary.ru/xbkmpl
- WHO: Ongoing avian influenza outbreaks in animals pose risk to humans; 2023. Available at: https://who.int/news/item/12-07-2023-ongoing-avian-influenza-outbreaks-in-animals-pose-risk-to-humans
- Adlhoch C., Fusaro A., Gonzales J.L., Kuiken T., Marangon S., Mirinaviciute G., et al. Avian influenza overview December 2022 – March 2023. EFSA J. 2023; 21(3): e07917. https://doi.org/10.2903/j.efsa.2023.7917
- Puryear W., Sawatzki K., Hill N., Foss A., Stone J.J., Doughty L., et al. Highly pathogenic avian influenza A(H5N1) virus outbreak in New England Seals, United States. Emerg. Infect. Dis. 2023; 29(4): 786–91. https://doi.org/10.3201/eid2904.221538
- Agüero M., Monne I., Sánchez A., Zecchin B., Fusaro A., Ruano M.J., et al. Highly pathogenic avian influenza A(H5N1) virus infection in farmed minks, Spain, October 2022. Euro Surveill. 2023; 28(3): 2300001. https://doi.org/10.2807/1560-7917.ES.2023.28.3.2300001
- Rabalski L., Milewska A., Pohlmann A., Gackowska K., Lepionka T., Szczepaniak K., et al. Emergence and potential transmission route of avian influenza A (H5N1) virus in domestic cats in Poland, June 2023. Euro. Surveill. 2023; 28(31): 2300390. https://doi.org/10.2807/1560-7917.ES.2023.28.31.2300390
- Lindh E., Lounela H., Ikonen N., Kantala T., Savolainen-Kopra C., Kauppinen A., et al. Highly pathogenic avian influenza A(H5N1) virus infection on multiple fur farms in the South and Central Ostrobothnia regions of Finland, July 2023. Euro. Surveill. 2023; 28(31): 2300400. https://doi.org/10.2807/1560-7917.ES.2023.28.31.2300400
- Gamarra-Toledo V., Plaza P.I., Gutiérrez R., Inga-Diaz G., Saravia-Guevara P., Pereyra-Meza O., et al. Mass mortality of sea lions caused by highly pathogenic avian influenza A(H5N1) virus. Emerg. Infect. Dis. 2023; 29(12): 2553–6. https://doi.org/10.3201/eid2912.230192
- Russier M., Yang G., Rehg J.E., Wong S.S., Mostafa H.H., Fabrizio T.P., et al. Molecular requirements for a pandemic influenza virus: An acid-stable hemagglutinin protein. Proc. Natl Acad. Sci. USA. 2016; 113(6): 1636–41. https://doi.org/10.1073/pnas.1524384113
- Herfst S., Schrauwen E.J., Linster M., Chutinimitkul S., de Wit E., Munster V.J., et al. Airborne transmission of influenza A/H5N1 virus between ferrets. Science. 2012; 336(6088): 1534–41. https://doi.org/10.1126/science.1213362
- Imai M., Watanabe T., Hatta M., Das S.C., Ozawa M., Shinya K., et al. Experimental adaptation of an influenza H5 HA confers respiratory droplet transmission to a reassortant H5 HA/H1N1 virus in ferrets. Nature. 2012; 486(7403): 420–8. https://doi.org/10.1038/nature10831
- Tosheva I.I., Saygan K.S., Mijnhardt S.M., Russell C.J., Fraaij P., Herfst S. Hemagglutinin stability as a key determinant of influenza A virus transmission via air. Curr. Opin. Virol. 2023; 61: 101335. https://doi.org/10.1016/j.coviro.2023.101335
- Richard M., Fouchier R.A. Influenza A virus transmission via respiratory aerosols or droplets as it relates to pandemic potential. FEMS Microbiol. Rev. 2016; 40(1): 68–85. https://doi.org/10.1093/femsre/fuv039
- Furuya-Kanamori L., Cox M., Milinovich G.J., Magalhaes R.J., Mackay I.M., Yakob L. Heterogeneous and dynamic prevalence of asymptomatic influenza virus infections. Emerg. Infect. Dis. 2016; 22(6): 1052–6. https://doi.org/10.3201/eid2206.151080
- Nguyen T.T.K., Ngo T.T., Tran P.M., Pham T.T.T., Vu H.T.T., Nguyen N.T.H., et al. Respiratory viruses in individuals with a high frequency of animal exposure in southern and highland Vietnam. J. Med. Virol. 2020; 92(8): 971–81. https://doi.org/10.1002/jmv.25640
- Lvov D.K., Shchelkanov M.Y., Alkhovsky S.V., Deryabin P.G. Zoonotic Viruses of northern Eurasia: Taxonomy and Ecology. London: Academic Press, Elsevier; 2015.
- Lvov D.K., Shchelkanov M.Y., Prilipov A.G., Vlasov N.A., Fedyakina I.T., Deryabin P.G., et al. Evolution of highly pathogenic avian influenza H5N1 virus in natural ecosystems of northern Eurasia (2005-08). Avian. Dis. 2010; 54(1 Suppl.): 483–95. https://doi.org/10.1637/8893-042509-Review.1
- Alexander D.J. Summary of avian influenza activity in Europe, Asia, Africa, and Australasia, 2002–2006. Avian. Dis. 2007; 51(1 Suppl.): 161–6. https://doi.org/10.1637/7602-041306R.1
- Yang Q., Wang B., Lemey P., Dong L., Mu T., Wiebe R.A., et al. Synchrony of bird migration with avian influenza global spread; implications for vulnerable bird orders. bioRxiv. Preprint. https://doi.org/10.1101/2023.05.22.541648
- Huang P., Sun L., Li J., Wu Q., Rezaei N., Jiang S., et al. Potential cross-species transmission of highly pathogenic avian influenza H5 subtype (HPAI H5) viruses to humans calls for the development of H5-specific and universal influenza vaccines. Cell Discov. 2023; 9(1): 58. https://doi.org/10.1038/s41421-023-00571-x
- Soda K., Tomioka Y., Usui T., Ozaki H., Ito H., Nagai Y., et al. Susceptibility of common dabbling and diving duck species to clade 2.3.2.1 H5N1 high pathogenicity avian influenza virus: an experimental infection study. J. Vet. Med. Sci. 2023; 85(9): 942–9. https://doi.org/10.1292/jvms.23-0122
- Lee S.H., Lee J., Noh J.Y., Jeong J.H., Kim J.B., Kwon J.H., et al. Age is a determinant factor in the susceptibility of domestic ducks to H5 clade 2.3.2.1c and 2.3.4.4e high pathogenicity avian influenza viruses. Front. Vet. Sci. 2023; 10: 1207289. https://doi.org/10.3389/fvets.2023.1207289
- Graaf A., Piesche R., Sehl-Ewert J., Grund C., Pohlmann A., Beer M., et al. Low susceptibility of pigs against experimental infection with HPAI virus H5N1 Clade h2.3.4.4b. Emerg. Infect. Dis. 2023; 29(7): 1492–5. https://doi.org/10.3201/eid2907.230296
- Subbiah J., Oh J., Kim K.H., Shin C.H., Park B.R., Bhatnagar N., et al. A chimeric thermostable M2e and H3 stalk-based universal influenza A virus vaccine. NPJ Vaccines. 2022; 7(1): 68. https://doi.org/10.1038/s41541-022-00498-6
- He X., Zhang T., Huan S., Yang Y. Novel influenza vaccines: from Research and Development (R&D) challenges to regulatory responses. Vaccines (Basel). 2023; 11(10): 1573. https://doi.org/10.3390/vaccines11101573
- Misplon J.A., Lo C.Y., Crabbs T.A., Price G.E., Epstein S.L. Adenoviral-vectored universal influenza vaccines administered intranasally reduce lung inflammatory responses upon viral challenge 15 months post-vaccination. J. Virol. 2023; 97(10): e0067423. https://doi.org/10.1128/jvi.00674-23
- Tripp R.A. Understanding immunity to influenza: implications for future vaccine development. Expert. Rev. Vaccines. 2023; 22(1): 871–5. https://doi.org/10.1080/14760584.2023.2266033
- WHO. Antigenic and genetic characteristics of zoonotic influenza viruses and development of candidate vaccine viruses for pandemic preparedness. Available at: https://who.int/influenza/vaccines/virus/characteristicsvirusvaccines/en
- WHO. Antigenic and genetic characteristics of zoonotic influenza A viruses and development of candidate vaccine viruses for pandemic preparedness in the 2024 southern hemisphere influenza season. Available at: https://cdn.who.int/media/docs/default-source/influenza/who-influenza-recommendations/vcm-southern-hemisphere-recommendation-2024/202309_zoonotic_vaccinvirusupdate.pdf?sfvrsn=e78676a0_5
- Ludwig S., Pleschka S., Planz O. MEK inhibitors as novel host-targeted antivirals with a dual-benefit mode of action against hyperinflammatory respiratory viral diseases. Curr. Opin. Virol. 2023; 59: 101304. https://doi.org/10.1016/j.coviro.2023.101304
- Chakraborty S., Chauhan A. Fighting the flu: a brief review on anti-influenza agents. Biotechnol. Genet. Eng. Rev. 2023; 1–52. https://doi.org/10.1080/02648725.2023.2191081
- Blake M.E., Kleinpeter A.B., Jureka A.S., Petit C.M. Structural Investigations of Interactions between the Influenza a Virus NS1 and Host Cellular Proteins. Viruses. 2023; 15(10): 2063. https://doi.org/10.3390/v15102063
- Львов Д.К., Альховский С.Д., Жирнов О.П. 130 лет вирусологии. Вопросы вирусологии. 2022; 67(5): 357–84. https://doi.org/10.36233/0507-4088-140 https://elibrary.ru/qhembl
Дополнительные файлы