Филогенетический анализ вариантов вируса Пуумала (Hantaviridae: Orthohantavirus), циркулирующих на территории Саратовской области
- Авторы: Краснов Я.М.1, Найденова Е.В.1, Гусева Н.П.1, Полунина Т.А.1, Шарапова Н.А.1, Соседова Е.А.1, Котова Н.В.1, Захаров К.С.1, Казанцев А.В.1, Доманина И.В.1, Чекашов В.Н.1, Шилов М.М.1, Кондратьев Е.Н.1, Осина Н.А.1, Кутырев В.В.1
-
Учреждения:
- ФКУН «Российский научно-исследовательский противочумный институт «Микроб» Роспотребнадзора
- Выпуск: Том 69, № 2 (2024)
- Страницы: 162-174
- Раздел: ОРИГИНАЛЬНЫЕ ИССЛЕДОВАНИЯ
- URL: https://virusjour.crie.ru/jour/article/view/16616
- DOI: https://doi.org/10.36233/0507-4088-224
- EDN: https://elibrary.ru/ijmubu
- ID: 16616
Цитировать
Аннотация
Цель работы – определение полной нуклеотидной последовательности и проведение филогенетического анализа вариантов геномов вируса Пуумала, выделенных на территории Саратовской области.
Материалы и методы. Образцами для исследования послужил полевой материал, собранный в Гагаринском (бывшем Саратовском), Энгельсском, Новобурасском и Хвалынском районах Саратовской области в период с 2019 по 2022 г. Для специфического обогащения генома вируса Пуумала в образцах использовали ПЦР и панель праймеров, подготовленную для данного исследования. Далее проводили секвенирование полученных продуктов реакции и сборку фрагментов в одну последовательность для каждого из сегментов генома вируса. При построении филогенетических деревьев применяли алгоритм maximum parsimony.
Результаты. Показано, что генетические варианты вируса Пуумала, выделенные в Саратовской области, имеют высокую степень подобия генома, что говорит о единстве их происхождения. По данным филогенетического анализа, все выделенные варианты вируса (за исключением изолятов вируса из Хвалынского района) образуют обособленную ветвь в кластере, сформированном хантавирусами из других субъектов Приволжского федерального округа. Самыми близкими к образцам из Саратовской области являются варианты вируса из республик Удмуртия и Татарстан, а также из Самарской и Ульяновской областей.
Заключение. Полученные данные указывают на наличие выраженной территориальной приуроченности штаммов к определенным регионам или областям, являющимся природными биотопами их носителей. Этот факт позволяет довольно точно определять территорию возможного инфицирования заболевших и/или циркуляцию переносчиков данных вариантов вируса по последовательности отдельных сегментов их генома.
Ключевые слова
Полный текст
Введение
Известно, что на Приволжский федеральный округ (ПФО) Российской Федерации приходится более 80% всех ежегодно зарегистрированных случаев заболевания геморрагической лихорадкой с почечным синдромом (ГЛПС) [1, 2], и Саратовская область не является исключением. Так, по данным, изложенным в письме Главного государственного санитарного врача Российской Федерации А.Ю. Поповой от 11.03.2024 № 02/4030-2024-32 «Об эпидемиологической ситуации по геморрагической лихорадке с почечным синдромом в 2023 году и прогнозе на 2024 год», в 2023 г. в России зарегистрировано 5093 случая ГЛПС (3,47 на 100 тыс. населения; среднемноголетний показатель (2012–2023 гг., без учета 2020 и 2021 гг.) – 5,34 на 100 тыс. населения), из которых 84,7% случаев приходится на ПФО.
Районирование территории Саратовской области показало, что в 16 из 38 административных районов за период с 2010 по 2022 г. была отмечена высокая интенсивность эпидемических проявлений ГЛПС. К максимально неблагополучным относится в том числе Гагаринский район (бывший Саратовский), а также природный парк «Кумысная поляна», расположенный на территории г. Саратова. Площадь парка составляет 4417 га [3]. В рамках организации местного самоуправления территория Гагаринского района с 1 января 2022 г. включена в городской округ г. Саратова, при этом не входя в черту города.
В европейской части России, в том числе и Саратовской области, наиболее распространенным возбудителем ГЛПС является вирус Пуумала (Puumala orthohantavirus), основной природный носитель (хозяин) которого – рыжая полевка (Myodes glareolus Schreber, 1780) [4, 5]. Вирус Пуумала, как и другие хантавирусы, принадлежит к роду Orthohantavirus семейства Hantaviridae [6]. Вирусный геном представлен сегментированной одноцепочечной РНК отрицательной полярности. Большой сегмент генома (L) кодирует вирусную РНК-полимеразу RdRp, средний (М) – два поверхностных гликопротеина G1 и G2, а малый сегмент (S) – нуклеокапсидный белок (N) [7].
В предыдущей работе [8], совместно с коллегами из ФБУН Государственный научный центр вирусологии и биотехнологии «Вектор», было проведено секвенирование полных геномов (сегменты S, M и L) трех образцов, взятых от рыжих полевок, пойманных в природном парке «Кумысная поляна» (г. Саратов) в середине 2019 г. Анализ полученных результатов позволил подтвердить, что вспышку ГЛПС, отмеченную в то время в Саратове, вызвал именно вирус Пуумала. Полученные нуклеотидные последовательности оказались близкими к ранее описанным вариантам из республик Татарстан и Удмуртия, а также Самарской области. Проведены филогенетический анализ вариантов вируса Пуумала, циркулирующих у рыжих полевок, отловленных на территории Республики Татарстан [9, 10], и сравнение с образцами из Удмуртской Республики и Ульяновской области [11]. Анализ показал близость вирусных геномов из исследуемых регионов с вариантами, изолированными в Татарстане. Установлено, что выявленные изоляты вируса Пуумала из Тюменской, Архангельской и Омской областей образуют общую восточно-финскую ветвь, что вызывает дополнительные вопросы о путях распространения основного носителя. Кроме этого, приводятся данные сравнения генетических вариантов из Татарстана, Удмуртии, Самары и Саратова, при этом авторы делают вывод о вероятном возникновении вариантов вируса Пуумала в данных регионах в результате реассортации, поскольку они содержали сегменты S и L, относящиеся к башкирской ветви, и сегмент M, возможно полученный от предков сублинии из Курской, Московской и Ивановской областей [12]. Также предпринята попытка смоделировать филогеографическое распространение хантавируса Пуумала по всей Европе в течение последнего послеледникового периода [13].
Таким образом, целью представленной работы являются определение полной нуклеотидной последовательности генома разных вариантов вируса Пуумала, выделенных на территории Саратовской области, и проведение филогенетического анализа полученных данных.
Материалы и методы
Образцами для исследования послужили пробы органов мелких млекопитающих, в основном рыжей полевки, собранных в 2019–2022 гг. в пригородной и городской зоне Саратова (Природный парк «Кумысная поляна»), а также в Гагаринском, Энгельсском, Новобурасском и Хвалынском районах Саратовской области. Зоологическая группа осуществляла регулярные выезды в точки, определенные планом ежегодного мониторинга, где и проводился отлов.
Авторы подтверждают соблюдение институциональных и национальных стандартов по использованию лабораторных животных в соответствии с «Consensus author guidelines for animal use» (IAVES 23 July 2010). Исследование одобрено комиссией по биоэтике ФКУН «Российский противочумный институт «Микроб» Роспотребнадзора (протокол № 8 от 21.11.2023).
Выделение нуклеиновых кислот осуществляли с помощью набора для экстракции РНК/ДНК «АмплиПрайм РИБО-преп», а обратную транскрипцию – с комплектом регентов «Реверта-L» (ЦНИИ Эпидемиологии Роспотребназора, Россия). Присутствие генетического материала хантавирусов определяли методом полимеразной цепной реакции с обратной транскрипцией (ОТ-ПЦР) с набором реагентов «ОМ-Скрин-ГЛПС-РВ» (НПК «Синтол», Россия).
Для исследований была составлена панель из 38 образцов суспензий легких мелких млекопитающих, в которых методом ОТ-ПЦР была выявлена РНК вируса Пуумала. Для специфического обогащения вирусного генома (сегменты S, M и L) в образцах из полевого материала использовали набор праймеров, рассчитанный авторами (табл. 1).
Таблица 1. Последовательности олигонуклеотидных праймеров, используемых в работе
Table 1. Oligonucleotide primer used in the work
Праймер Primer | Структура праймера Primer sequence | Температура отжига, °C Annealing T, °C | Длина ампликона, п.н. Amplicon length, bp |
Сегмент S Segment S | |||
Pum_S_1f | TAGTAGTAGACTCCTTGAAAAAGCTAC | 50 | 370 |
Pum_S_1r | CAATGTCAATGGCGTTCAC | ||
Pum_S_2f | GAAGAATGGCAGATGCTGTGTCCC | 58 | 454 |
Pum_S_2r | ACGGTCTGTCTTCCACGAGTTGAC | ||
Pum_S_3f | CGACTGGGATTGAACCTGATGATC | 56 | 344 |
Pum_S_3r | CGGGTGTAAGTTCCTCAGCTTTC | ||
Pum_S_4f | CATTTGAGGATATTAATGGCTTTAGG | 54 | 376 |
Pum_S_4r | AATCAACTTATCAATGTCTGCCAC | ||
Pum_S_5f | TGCGTAATATCATGAGTCCAGTGATG | 56 | 355 |
Pum_S_5r | AGCCATCCCAGCAACATAAATG | ||
Pum_S_6f | ACATCGAATCTCCTAATGCACC | 54 | 354 |
Pum_S_6r | TGATCTATGAGTGACTGAGCAAGG | ||
Pum_S_7f | TGCTCAGTCACTCATAGATCAGAAAG | 53 | 400 |
Pum_S_7r | AGCTCAGTTTCACATTCTTGGG | ||
Pum_S_7-2f | AATCAGGAGCCCTTAAAGATATG | 53 | 467 |
Pum_S_7-2r | TCAGCATGTTGAGGTAGTATGTTGTG | ||
Pum_S_fin_f | GTTTTGAATTAATGCACTAATCAGGG | 50 | 350 |
Pum_S_fin_r | TAGTAGTATGCTCCTTGAAAAGCAATC | ||
Сегмент М Segment M | |||
Pum_М_1f | TAGTAGTAGACTCCGCAAGAAGAAGC | 54 | 386 |
Pum_М_1r | ATCCTCTCAAATTCACTTCACTGC | ||
Pum_М_2f | CATGGGAAATTAAAGGTGATCTTG | 54 | 309 |
Pum_М_2r | AATTGCCCTGAAACACAGTATG | ||
Pum_М_3f | GTTTGATCCCTACTTTAGTGGTTG | 53 | 349 |
Pum_М_3r | CTTCATACTATCACCAGATGTCACC | ||
Pum_М_4f | ATACTGTGTTTCAGGGCAATTGG | 56 | 386 |
Pum_М_4r | CCTGTTACTTTCCCAGCAATACGTAG | ||
Pum_М_5f | ATCGTTCTGCTGAAGTTCTTTCAAG | 56 | 345 |
Pum_М_5r | AAGACTGTGCATTGTGTTGTCTTCTC | ||
Pum_М_6f | ACTGGGTTCATCTCATTACCTGG | 54 | 341 |
Pum_М_6r | TAACTCTACGGCAAGAGAGTGTG | ||
Pum_М_7f | GAGTTATGTGTACCAGGTCTTCACGG | 55 | 340 |
Pum_М_7r | GCTTGAAGGGCAGATGTTGTTG | ||
Pum_М_8f | ATGGGCTCGATGGTCTGTGAGG | 58 | 380 |
Pum_М_8r | TATCCCAGACCCGTGTGCTGTGTC | ||
Pum_М_9f | CTCTCTTTAGGTATCGGAGTCGG | 55 | 392 |
Pum_М_9r | CAGCAGTTTGCCAAGGATAAGC | ||
Pum_М_10f | AAACTGCATTTCATTGCTATGGTTC | 55 | 326 |
Pum_М_10r | TTGAAATTGTCCCTATCAAACACAC | ||
Pum_М_10-2f | GGCTGTAACCCACTTGATTGCC | 58 | 430 |
Pum_М_10-2r | CCATCAAACTGGCACACAGGTGTTG | ||
Pum_М_11f | TAATATTTAAGCAATGGTGCACTAC | 53 | 452 |
Pum_М_11r | CCTTAATTGAAGTAAGAAATGCAG | ||
Pum_М_12f | GATGGGAATACAATTTCAGGATAC | 53 | 307 |
Pum_М_12r | GCCTTAATTGAAGTAAGAAATGCAG | ||
Pum_М_13f | GAATGGATTGATCCTGACAGTTCAC | 56 | 502 |
Pum_М_13r | ATCCAATTTCCATTGAGGACCC | ||
Pum_М_13-2f | CAATGTGTTACGGATCTACTACAGC | 54 | 393 |
Pum_М_13-2r | GCAACTATCTACTAAGGCTTATGCTC | ||
Pum_М_fin_f | GACAGGATACAACCAAGCAGATAGTG | 54 | 417 |
Pum_М_fin_r | TAGTAGTAGACTCCGCAAGAACAAAAG | ||
Сегмент L Segment L | |||
Pum_L_1f | TAGTAGTAGACTCCGAGATAGAGAAG | 51 | 468 |
Pum_L_1r | TGTGCTTCTACCTGTAGTTGTTGCTC | ||
Pum_L_2f | ATGATGTGATACAAAGCATGGAG | 53 | 453 |
Pum_L_2r | TCCTAAAGCCAGATTGACAATTAC | ||
Pum_L_3f | GTTTATTGAGCAACAACTACAGGTAG | 53 | 394 |
Pum_L_3r | GCCCAGTTACTTCTTTAAATGC | ||
Pum_L_4f | TGTAAGAATTGGCTCGGAACTGATC | 56 | 421 |
Pum_L_4r | TCAATCAATGCCTTCGACTTAGGATC | ||
Pum_L_5f | ATCTCAAAGGATTCAAAGAAAGGG | 54 | 391 |
Pum_L_5r | TCAAGTGATTTAGATGGCAGGATAC | ||
Pum_L_6f | CGTGATATTACTGAATCTCTTATTGC | 53 | 406 |
Pum_L_6r | TGAGATGTGGAAAGAAAGAAATGG | ||
Pum_L_7f | CTTCTTGCTACAGCTACATGGTTTC | 54 | 410 |
Pum_L_7r | ACGATAATGCTTATACACGACCC | ||
Pum_L_8f | AAGACACTATTAGTTAGCTTAGCCC | 52 | 354 |
Pum_L_8r | GCTGTTCAACAACTACCTGATTG | ||
Pum_L_9f | TTCATCTTGAAACAGTTGAATGGG | 54 | 438 |
Pum_L_9r | CTTTGGTATTTCCTAACAATTCTTGC | ||
Pum_L_9-2f | TCATCTTGAAACAGTTGAATGGG | 54 | 341 |
Pum_L_9-2r | AAACGAATTGCCTCAATGAGAG | ||
Pum_L_10f | GTCAGGAGCATTACAAGAAGATGGTC | 56 | 369 |
Pum_L_10r | CAACGAGGACTGGATTTCACTTTCTC | ||
Pum_L_11f | ATTCAACAGGCTTTAGAGAAGGC | 54 | 450 |
Pum_L_11r | GATATAATTTGGCCCACACACG | ||
Pum_L_12f | AATATGGGTGAATTGTCTGATGAAG | 54 | 440 |
Pum_L_12r | GAATGTAGATAGAAACTCCGCATTTG | ||
Pum_L_13f | GGCAGGAAACTTTCATTGGCATG | 57 | 440 |
Pum_L_13r | GACATAGAACCATCACCACCTAACGG | ||
Pum_L_14f | TTATATGGTACTGCTCCTGGTATGG | 54 | 383 |
Pum_L_14r | AGGATGTTGTTCACTCCAAAGCTC | ||
Pum_L_15f | TTTGTAGGTAAAGTTCAGTGGAAAG | 53 | 459 |
Pum_L_15r | TTGGTCTATGAATTTGTCTTGTTG | ||
Pum_L_15-2f | CTTTGGAGTGAACAACATCCTGTC | 54 | 320 |
Pum_L_15-2r | ATCCCTCCAGGCATATTCTTTAG | ||
Pum_L_16f | GGATATGGAGCTCTTTCAAACACTTG | 55 | 387 |
Pum_L_16r | CACAATTACAATCCTCGACTTTCC | ||
Pum_L_17f | TTATTGGTTGAAGACTATGTCTCTTG | 52 | 441 |
Pum_L_17r | ATTAAATGTTACCCTCAAATCTCC | ||
Pum_L_18f | CTTTATGAAGGAGATTTGAGGGTAAC | 53 | 423 |
Pum_L_18r | CCTTTAGATTATGGTATGCATGG | ||
Pum_L_19f | CTAATGGATTTAGGGCTATGGC | 54 | 385 |
Pum_L_19r | TGACCATTGAGTACTAGAGATTGTGC | ||
Pum_L_20f | AGCACAATCTCTAGTACTCAATGGTC | 53 | 405 |
Pum_L_20r | CGAACTCTGTTAAATCATACGGATC | ||
Pum_L_fin_f | GTCCAAGCATTACAATTTCCATAC | 52 | 304 |
Pum_L_fin_r | TAGTAGTATGCTCCGAGAAAAGAGC | ||
Далее секвенирование полученных продуктов ПЦР проводили с помощью генетического анализатора АВ 3500xl и/или Ion S5, в соответствии с рекомендациями производителя (Thermo Fisher Scientific, США). Для сборки секвенированных фрагментов в одну последовательность использовали программное обеспечение MEGA7 (http://www.megasoftware.net/) и UGENE (http://ugene.net).
При построении филогенетических деревьев в работу брали последовательность сегмента S (центральная часть в 1071 нуклеотид), а также бо́льшую часть последовательности сегментов M и L генома вируса Пуумала, представленных в международной базе данных NCBI GeneBank. Данный раздел работы проводили с использованием алгоритма maximum parsimony в программном пакете BioNumerics 7.6 (Applied Maths, Бельгия).
Результаты и обсуждение
В результате проведенного исследования были получены нуклеотидные последовательности генома вируса Пуумала, циркулирующего на территории Саратовской области, из которых 33 с наиболее качественным уровнем прочтения депонированы в международной базе данных NCBI GenBank (табл. 2).
Таблица 2. Нуклеотидные последовательности вируса Пуумала, полученные в результате исследования
Table 2. List of Puumala orthohantavirus sequences obtained in this study and deposited in the NCBI GenBank database
№ No. | Образец Sample | Место получения образца Place of the sample collection | Год сбора материала Year of collection | Номер в базе NCBI GenBank GenBank Accession Number | ||
сегмент S segment S | сегмент M segment M | сегмент L segment L | ||||
1. | 114 | Гагаринский район Gagarinsky district | 2019 | OL343591.1 | OL343569.1 | OL343547.1 |
2. | 131 | 2019 | OL343593.1 | OL343571.1 | OL343549.1 | |
3. | 296 | 2019 | OL343594.1 | OL343572.1 | OL343550.1 | |
4. | 348 | 2019 | OL343595.1 | OL343573.1 | OL343551.1 | |
5. | 420 | 2019 | OL343596.1 | OL343574.1 | OL343552.1 | |
6. | 422 | 2019 | OL343597.1 | OL343575.1 | OL343553.1 | |
7. | 439 | 2019 | OL343598.1 | OL343576.1 | OL343554.1 | |
8. | 525 | 2019 | OL343599.1 | OL343577.1 | OL343555.1 | |
9. | 645 | 2019 | OL343600.1 | OL343578.1 | OL343556.1 | |
10. | 656 | 2019 | OL343601.1 | OL343579.1 | OL343557.1 | |
11. | 696 | 2019 | OL343602.1 | OL343580.1 | OL343558.1 | |
12. | 701 | 2019 | OL343603.1 | OL343581.1 | OL343559.1 | |
13. | 836 | 2019 | OL343604.1 | OL343582.1 | OL343560.1 | |
14. | 988 | 2019 | OL343605.1 | OL343583.1 | OL343561.1 | |
15. | 989 | 2019 | OL343606.1 | OL343584.1 | OL343562.1 | |
16. | 1039 | 2019 | OL343585.1 | OL343563.1 | OL343541.1 | |
17. | 1042 | 2019 | OL343586.1 | OL343564.1 | OL343542.1 | |
18. | 1059 | 2019 | OL343587.1 | OL343565.1 | OL343543.1 | |
19. | 1081 | 2019 | OL343588.1 | OL343566.1 | OL343544.1 | |
20. | 1107 | 2019 | OL343589.1 | OL343567.1 | OL343545.1 | |
21. | 1109 | 2019 | OL343590.1 | OL343568.1 | OL343546.1 | |
22. | 1186 | 2019 | OL343592.1 | OL343570.1 | OL343548.1 | |
23. | 3 | 2022 | OQ032670.1 | * | * | |
24. | 29 | 2022 | OQ032672.1 | * | * | |
25. | 30 | 2022 | OQ032673.1 | * | * | |
26. | 31 | 2022 | OQ032671.1 | * | * | |
27. | 99 | Энгельсский район Engelssky district | 2022 | OQ032667.1 | * | * |
28. | 100 | 2022 | OQ032669.1 | * | * | |
29. | 101 | 2022 | OQ032668.1 | * | * | |
30. | 117 | Хвалынский район Khvalynsky district | 2021 | OR999067 | * | * |
31. | 122 | 2021 | OR999068 | * | * | |
32. | 129 | 2021 | OR999069 | * | * | |
33. | 272 | Новобурасский район Novoburassky district | 2021 | OR999070 | * | * |
Примечание. * – данные на этапе анализа.
Note. * – Data at the analysis stage.
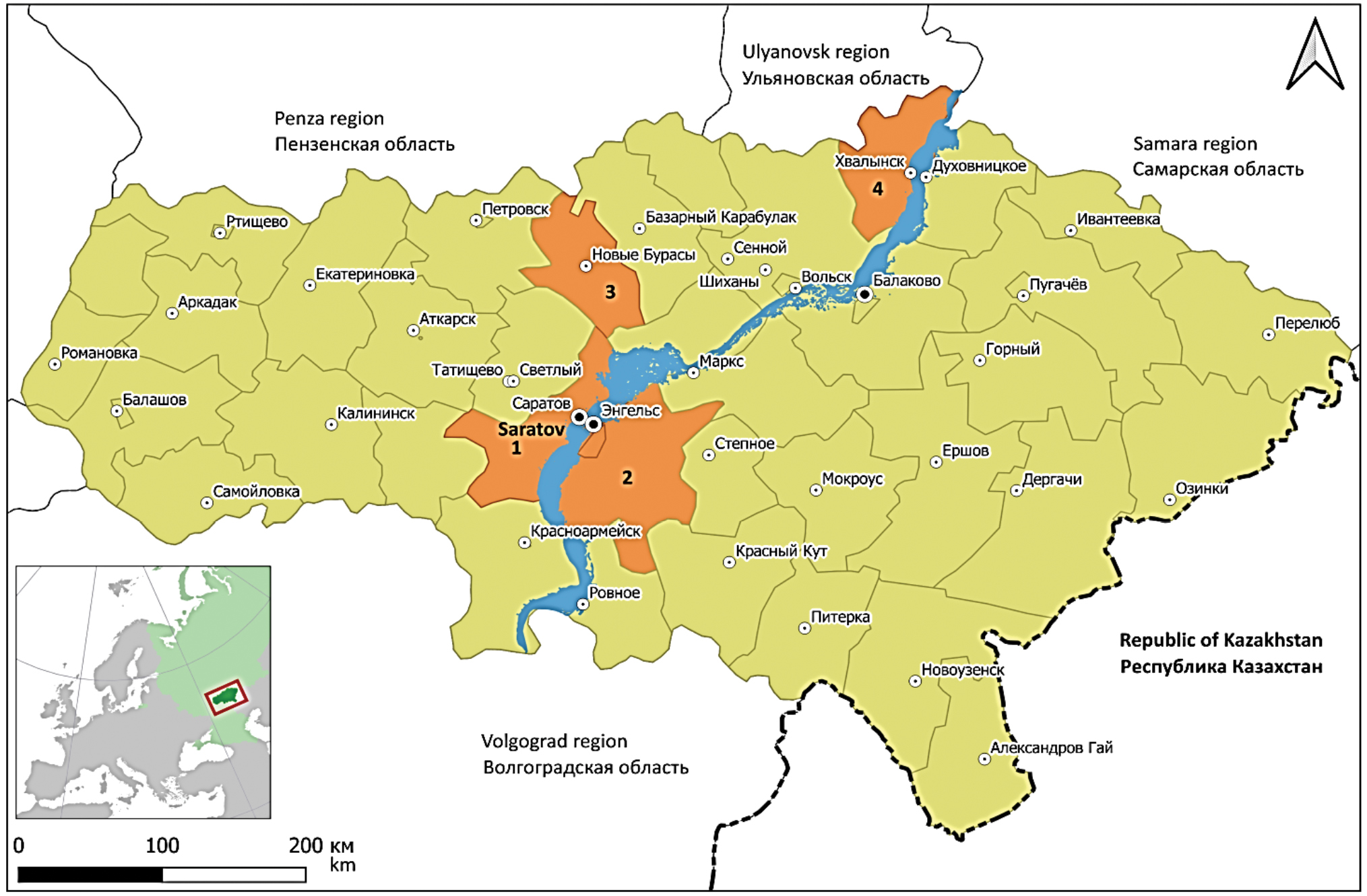
Сравнительный анализ 26 нуклеотидных последовательностей полного генома вируса Пуумала из пригородной и городской зон Саратова (Гагаринский район) показал их высокую идентичность между собой (98,6–100%), независимо от года изоляции. Образцы из Новобурасского (№ 272) и Энгельсского (№№ 99, 100, 101) районов имели идентичность генома 98,4–99,1% с 26 образцами из Гагаринского района. Три образца из Хвалынского района Саратовской области (№№ 117, 122, 129) были идентичны на 95,5% со всеми вариантами из Гагаринского района. Таким образом, выявлено достаточно большое количество отличий среди вариантов генома вируса из Хвалынского района по сравнению с образцами из Энгельсского, Гагаринского и Новобурасского районов, граничащих друг с другом (рис. 1).
Рис. 1. Административное деление Саратовской области. Цветом выделены Гагаринский район и городской округ Саратова (1), Энгельсский (2), Новобурасский (3) и Хвалынский (4) районы.
Fig. 1. Administrative division of the Saratov region. The city district of Saratov (1), Engelssky (2), Novoburassky (3) and Khvalynsky (4) districts are highlighted in color.
С использованием имеющихся нуклеотидных последовательностей S-сегмента генома вируса Пуумала, представленных в международной генетической базе, был проведен анализ, включающий 368 вирусных вариантов (рис. 2, а) и построено общее филогенетическое дерево. Сравнение последовательностей М- или L-сегментов генома хантавирусов ограничивается гораздо меньшим их количеством относительно сегмента S, представленным в базе данных, но при этом картина кластеризации изменяется незначительно, сохраняя, как правило, то же взаимное расположение.
Рис. 2. Филогенетическое дерево, показывающее близость родства между 368 вариантами генома (сегмент S) вируса Пуумала с территории России, стран ближнего и дальнего зарубежья. а – по трем кластерам; б – увеличенный фрагмент филогенетического дерева – кластер 1 на рис. 2 а. На рис. 2 б образцы, обведенные красным цветом, остались единственными вариантами из республик Башкортостан и Татарстан, Самарской и Ульяновской областей в случае филогенетических деревьев по сегментам M и L.
Fig. 2. Phylogenetic tree showing the close relationship between 368 genome variants (segment S) strains of the Puumala orthohantavirus from Russia, near and far abroad countries. a – for three clusters; b – enlarged fragment of the phylogenetic tree – cluster 1 in Figure 2 a. The close relationship between genome variants (segment S) of 151 strains of the Puumala orthohantavirus from the Volga Federal District is shown. In Figure 2 b, samples circled in red remained the only variants from the republics of Bashkortostan and Tatarstan, Samara and Ulyanovsk regions in the case of phylogenetic trees for segments M and L.
При установлении филогенетического родства варианты генома вируса Пуумала из Саратовской области по сегменту S образовали общий кластер с вирусными последовательностями из Ульяновской, Самарской и Пензенской областей, а также республик Татарстан, Башкортостан и Удмуртия. Варианты последовательностей этого же сегмента генома, полученные на других территориях России (Омская, Тюменская, Архангельская, Московская, Ивановская и Курская области, Республика Карелия), оказались значительно удалены от кластера, сформированного из образцов с территории перечисленных выше субъектов ПФО. Стоит отметить, что на одной ветви, образованной геномами из Самарской области, находятся два образца (NCBI, MT822195 и MT822196), полученные из клинического материала, собранного в январе 2020 г. в Швейцарии (рис. 2 а, б). Но в аннотации к этим последовательностям нет информации о возможном месте заражения больных ГЛПС, в результате чего четкие выводы сделать сложно. В остальном в кластер, образованный из последовательностей генома вируса (сегмент S) с территории ПФО, не попало ни одного варианта из других федеральных округов России, стран ближнего или дальнего зарубежья.
Внутри кластера 1 (рис. 2 а, б), образованного геномами вируса Пуумала из образцов с территории ПФО, степень идентичности последовательностей составляет 91,4–100%. Несмотря на то что в сравнительном анализе использовалась только центральная часть последовательности сегмента S (1071 пн), каждый вариант из данного кластера имеет не менее 62 уникальных единичных мутаций, являющихся общими для всех образцов из кластера 1 и отличающих эти варианты от всех других (рис. 2 а, б ). На рис. 2 б показано, что варианты генома хантавируса Пуумала из городского округа Саратова и Энгельсского района и один – из Новобурасского района образуют компактную группу в виде отдельного ответвления. Места отлова носителей вируса Пуумала в пределах городского округа Саратова находились друг от друга от нескольких сотен метров до 9 км. Между местами отлова носителей вируса Пуумала в пределах городского округа Саратова и Энгельсского района расстояние по прямой составляет 14–19 км, а для образца из Новобурасского района – около 46 км. Несмотря на значительное удаление, для вариантов генома вируса Пуумала с данных территорий наблюдается высокая степень идентичности (98% и более), что может свидетельствовать о едином биотопе основных носителей и свободном их перемещении в пределах указанных регионов. Хвалынский район находится на севере Саратовской области (расстояние до других обследованных регионов более 100 км) и граничит с Ульяновской и Самарской областями (рис. 1). Данный факт четко отражается на расположении в кластере 1 (рис. 2 б) вариантов вируса Пуумала. Показано, что образцы из Хвалынского района находятся на разных ветвях с вариантами из Гагаринского, Энгельсского и Новобурасского районов, отличаясь от них на 55–78 однонуклеотидных полиморфизмов (ОНП) (Single Nucleotide Polymorphism, SNP), т.е. имеют идентичность с последними 92,7–96,9%. Ближайшими к вариантам хантавируса из Хвалынского района являются геномы вируса Пуумала из Самарской области, затем его варианты из Республики Татарстан и Ульяновской области (рис. 2 б).
Детальное рассмотрение расположения вариантов генома (сегмент S) вируса Пуумала из Саратовской области (рис. 2 б) показывает очень близкое родство между образцами с территории Гагаринского района, а также их большое сходство с образцами из Энгельсского и Новобурасского районов. Отличие между последовательностью сегмента S (1071 пн) у варианта вируса Пуумала, выделенного в августе 2021 г. в Новобурасском районе, от ближайшего к нему варианта Пуумала, полученного в мае 2019 г. на территории Гагаринского района, составляет 5 ОНП. Отличие между последовательностями сегмента S (1071 пн) у варианта вируса Пуумала, обнаруженного в июле 2022 г. в Энгельсском районе, от ближайшего к нему, полученного в феврале 2020 г. при обследовании Гагаринского района, составляет 15 ОНП. Между всеми исследованными вариантами вируса Пуумала, циркулирующими на территории Гагаринского района Саратовской области, максимальное отличие в последовательности сегмента S (1071 пн) составляет 22 ОНП (идентичность 98% и более).
Анализ молекулярных часов, проведенный группами исследователей [14, 15], показал, что S-сегмент генома хантавирусов эволюционирует со скоростью около 6,7 × 10−4 единичных замен в год. Эти данные свидетельствуют о довольно медленном изменении генома вируса Пуумала в природе. В целом кластеризация по сегменту S показывает общее происхождение от одного предкового вируса или нескольких близких друг другу его вариантов для всех использованных в анализе образцов с территории ПФО.
Сравнительный филогенетический анализ вируса Пуумала по сегментам М или L образцов, полученных на территории Саратовской области, показывает некоторые изменения в кластеризации, сохраняя, однако, общие тенденции взаимного расположения.
Следует отметить, что по сегментам М или L данных о последовательностях генома вируса Пуумала значительно меньше, чем по сегменту S. На рис. 2 б образцы, обведенные красным цветом, остались единственными вариантами из республик Башкортостан и Татарстан, Самарской и Ульяновской областей в случае филогенетических деревьев по сегментам M и L.
Рис. 3. Филогенетическое дерево, показывающее близость родства между 94 вариантами генома (сегмент М) вируса Пуумала с территории России, стран ближнего и дальнего зарубежья.
Примечание: Башкортостан (AB297666, AF442614, KT885051, L08754, MH251332, MK496160, MZ673553, NC_077666); Курская обл. (MZ580943, MZ580946, MZ580949, MZ580952); Ивановская обл. (OP561838); Московская обл. (OP561841); Самарская обл. (AB433850, AB433852); Ульяновская обл. (OP561826); Татарстан (Z84205); Удмуртия (OP561835); Пензенская обл. (OP561847); Швейцария (MT822194); Омская обл. (AF367061, AF442615, AF442616, AF442617); Архангельская обл. (OP561850); Тюменская обл. (OP561853).
Fig. 3. Phylogenetic tree showing the close relationship between 94 genome variants (segment M) of Puumala orthohantavirus strains from Russia, near and far abroad countries.
Note: Bashkortostan (NCBI: AB297666, AF442614, KT885051, L08754, MH251332, MK496160, MZ673553, NC_077666); Kursk region (MZ580943, MZ580946, MZ580949, MZ580952); Ivanovo region (OP561838); Moscow region (OP561841); Samara region (AB433850, AB433852); Ulyanovsk region (OP561826); Tatarstan (Z84205); Udmurtia (OP561835); Penza region (OP561847); Switzerland (MT822194); Omsk region (AF367061, AF442615, AF442616, AF442617); Arkhangelsk region (OP561850); Tyumen region (OP561853).
На рис. 3 представлен результат филогенетической кластеризации между 94 вариантами генома (сегмент M, локус в 2920 пн) вируса Пуумала с территории России, стран ближнего и дальнего зарубежья. Полученное филогенетическое дерево показывает, что образцы из Гагаринского района (Saratov ar.) по-прежнему образуют однородную группу и находятся в одном кластере с вариантами из субъектов ПФО, при этом ближайшими по подобию к ним являются варианты вируса из республик Удмуртия (OP561835, идентичность 94,9–95,6%), Татарстан (Z84205, идентичность 94,6–95,3%) и Самарской области (AB433850, идентичность 91,3–92,0% и AB433852, идентичность 90,6–91,3%). Обращает на себя внимание то, что при кластеризации по сегменту М последовательности генома вируса, полученные из Московской, Курской и Ивановской областей, располагаются ближе к вариантам с территории ПФО, нежели варианты из Республики Башкортостан входящей в ПФО. По сегменту М варианты из Башкирии образуют отдельную ветвь, не входящую в кластеры 1 и 2. Напротив, при филогенетической кластеризации вируса по сегменту S все последовательности из ПФО были в одном кластере и значительно удалены от вариантов из Центральной части России. Вероятно, у хантавируса, полученного в Республике Башкортостан, произошла реассортация по сегменту М с каким-то вариантом данного вируса из региона, не входящего в состав ПФО.
Заключение ряда авторов [12] о вероятном возникновении вариантов вируса Пуумала на территории Татарстана, Удмуртии, Самары и Саратова в результате реассортации, поскольку они содержали сегменты S и L, относящиеся к башкирской ветви, и сегмент M, полученный от предков сублинии из Курской, Московской и Ивановской областей, не получило подтверждения в настоящем исследовании. Очевидно стабильное сохранение единства филогенетической обособленности геномов вируса Пуумала на территории субъектов ПФО, за исключением вариантов с территории Башкортостана по сегменту М (рис. 3) и образца из Пензенской области – по сегменту L (рис. 4). На рис. 3, где представлено сродство по сегменту М, любой из вариантов генома вируса Пуумала в кластере 1 отличается как минимум на 433 ОНП (идентичность 85,2%) от любого из вариантов генома вируса Пуумала в кластере 2. Отличие вариантов из Башкортостана составляет не менее 531 ОНП (идентичность 81,8%) от любого из вариантов как в кластере 1, так и кластере 2. Ветвь с ближайшими вариантами вируса Пуумала из Финляндии имеет около 580 ОНП (идентичность 80,1%) от вариантов из Башкортостана.
Рис. 4. Филогенетическое дерево, показывающее близость родства между 76 вариантами генома (сегмент L) вируса Пуумала с территории России, стран ближнего и дальнего зарубежья.
Примечание: Башкортостан (AB297667, KT885050, MK496161, MK496164, MH251333, NC_077667); Самарская обл. (AB574183, AB574184); Ульяновская обл. (OP561825); Удмуртия (OP561834); Татарстан (EF405801); Швейцария (MT822193); курская обл. (MZ580944, MZ580947, MZ580953, MZ580950); Московская обл. (OP561840, OP561843); Ивановская обл. (OP561837); Пензенская обл. (OP561846); Архангельская обл. (OP561849); Тюменская обл. (OP561852).
Fig. 4. Phylogenetic tree showing the close relationship between 76 genome variants (segment L) of Puumala orthohantavirus strains from the territory of Russia, near and far abroad countries.
Note: Bashkortostan (NCBI: AB297667, KT885050, MK496161, MK496164, MH251333, NC_077667); Samara region (AB574183, AB574184); Ulyanovsk region (OP561825); Udmurtia (OP561834); Tatarstan (EF405801); Switzerland (MT822193); Kursk region (MZ580944, MZ580947, MZ580953, MZ580950); Moscow region (OP561840, OP561843); Ivanovo region (OP561837); Penza region (OP561846); Arkhangelsk region (OP561849); Tyumen region (OP561852).
Отсутствие данных с большинства областей России о циркулирующих на них вариантах вируса Пуумала очень затрудняет возможность сделать выводы о возможных путях возникновения реассортантов.
На рис. 4 представлен результат филогенетической кластеризации между 76 вариантами генома (сегмент L, локус в 6405 пн) вируса Пуумала с территории России, стран ближнего и дальнего зарубежья. Все варианты из Гагаринского района (Saratov ar.) Саратовской области неизменно находятся в одном кластере с другими вирусами Пуумала из субъектов ПФО. Ближайшими к вариантам из Саратовской области, как и в случае сегмента М, являются образцы вируса Пуумала из республик Удмуртия (OP561834, идентичность 95,2–95,8%) и Татарстан (EF405801, идентичность 94,5–95,1%), а также Самарской области (AB574183 и AB574184, идентичность 91,7–92,7%). При анализе сегмента L варианты вируса Пуумала из Республики Башкортостан, хотя и располагаются в одном кластере (кластер 1) вместе вариантами данного вируса из других субъектов ПФО, но значительно от них обособлены, максимальная идентичность составляет 86,1% (около 890 ОНП от ближайшего к ним варианта из Ульяновской обл. в этом кластере). Результат филогенетической кластеризации вариантов генома вируса Пуумала по сегменту L показывает, что образец из Пензенской области не располагается в кластере 1 вместе с вариантами вируса из ПФО, а находится в кластере 2 с вариантами из Ивановской, Московской и Курской областей. Как видно из рис. 2 б и рис. 3, образец из Пензенской области по сегментам S и M относится к кластеру 1, состоящему только из вариантов вируса с территории ПФО. Таким образом, показана возможная реассортация по сегменту L варианта вируса Пуумала из Пензенской области с вариантами этого вируса из Ивановской, Московской или Курской областей, видимо, в результате миграции рыжей полевки. В опубликованных ранее материалах авторы отмечали, что данный вариант генома мог возникнуть в результате двухэтапной рекомбинации [12].
Заключение
Полученные результаты филогенетического анализа разных вариантов вируса Пуумала из Саратовской области с последовательностями, представленными в базе NCBI GenBank, позволяют сделать следующие выводы:
- – все варианты штаммов вируса Пуумала, циркулирующие в Саратовской области, имеют высокую степень подобия геномов, что может свидетельствовать о единстве их происхождения;
- – генетические последовательности вируса Пуумала из Хвалынского района имеют значительные отличия от вариантов, полученных из центральных регионов Саратовской области;
- – все генетические последовательности вируса Пуумала, полученные из городского округа Саратова, в том числе и по сегментам генома S, M и L, не имеют реассортации.
Обобщая полученные данные можно отметить наличие выраженной территориальной приуроченности штаммов вируса Пуумала (точнее их носителей) к определенным регионам или областям. Это позволяет по последовательности сегментов вирусного генома довольно точно определять район возможного инфицирования заболевших и места обитания переносчиков данных геновариантов вируса Puumala orthohantavirus.
Материал подготовлен к публикации на основании исследований, выполненных в рамках НИР 92-2-21 «Разработка системы верификации результатов индикации и идентификации возбудителей опасных инфекционных болезней бактериальной и вирусной природы с помощью молекулярно-генетических методов» (2021–2023 гг.).
Об авторах
Ярослав Михайлович Краснов
ФКУН «Российский научно-исследовательский противочумный институт «Микроб» Роспотребнадзора
Email: rusrapi@microbe.ru
ORCID iD: 0000-0002-4909-2394
кандидат хим. наук, ведущий научный сотрудник
Россия, 410005, СаратовЕкатерина Владимировна Найденова
ФКУН «Российский научно-исследовательский противочумный институт «Микроб» Роспотребнадзора
Автор, ответственный за переписку.
Email: katim2003@mail.ru
ORCID iD: 0000-0001-6474-3696
кандидат биологических наук, ведущий научный сотрудник
Россия, 410005, СаратовНаталья Петровна Гусева
ФКУН «Российский научно-исследовательский противочумный институт «Микроб» Роспотребнадзора
Email: rusrapi@microbe.ru
ORCID iD: 0000-0003-3763-9708
кандидат биол. наук, старший научный сотрудник
Россия, 410005, СаратовТатьяна Алексеевна Полунина
ФКУН «Российский научно-исследовательский противочумный институт «Микроб» Роспотребнадзора
Email: rusrapi@mail.ru
ORCID iD: 0000-0002-2234-2760
кандидат мед. наук, старший научный сотрудник
Россия, 410005, СаратовНаталья Анатольевна Шарапова
ФКУН «Российский научно-исследовательский противочумный институт «Микроб» Роспотребнадзора
Email: rusrapi@microbe.ru
ORCID iD: 0000-0002-5289-7783
кандидат биол. наук, научный сотрудник
Россия, 410005, СаратовЕкатерина Анатольевна Соседова
ФКУН «Российский научно-исследовательский противочумный институт «Микроб» Роспотребнадзора
Email: rusrapi@microbe.ru
ORCID iD: 0009-0004-4443-2646
научный сотрудник
Россия, 410005, СаратовНина Владимировна Котова
ФКУН «Российский научно-исследовательский противочумный институт «Микроб» Роспотребнадзора
Email: rusrapi@microbe.ru
ORCID iD: 0000-0002-9270-523X
научный сотрудник
Россия, 410005, СаратовКирилл Сергеевич Захаров
ФКУН «Российский научно-исследовательский противочумный институт «Микроб» Роспотребнадзора
Email: zaharov_ks@mail.ru
ORCID iD: 0000-0002-4726-309X
кандидат биол. наук, старший научный сотрудник
Россия, 410005, СаратовАндрей Владимирович Казанцев
ФКУН «Российский научно-исследовательский противочумный институт «Микроб» Роспотребнадзора
Email: andreikazancev@mail.ru
ORCID iD: 0000-0003-1790-0411
научный сотрудник
Россия, 410005, СаратовИрина Владимировна Доманина
ФКУН «Российский научно-исследовательский противочумный институт «Микроб» Роспотребнадзора
Email: rusrapi@microbe.ru
ORCID iD: 0000-0002-4731-8089
научный сотрудник
Россия, 410005, СаратовВладимир Николаевич Чекашов
ФКУН «Российский научно-исследовательский противочумный институт «Микроб» Роспотребнадзора
Email: rusrapi@microbe.ru
ORCID iD: 0000-0002-9593-4353
кандидат биол. наук, старший научный сотрудник
Россия, 410005, СаратовМихаил Михайлович Шилов
ФКУН «Российский научно-исследовательский противочумный институт «Микроб» Роспотребнадзора
Email: rusrapi@microbe.ru
ORCID iD: 0000-0002-0083-8212
кандидат биол. наук, научный сотрудник
Россия, 410005, СаратовЕвгений Николаевич Кондратьев
ФКУН «Российский научно-исследовательский противочумный институт «Микроб» Роспотребнадзора
Email: rusrapi@microbe.ru
ORCID iD: 0000-0002-7508-4355
научный сотрудник
Россия, 410005, СаратовНаталья Александровна Осина
ФКУН «Российский научно-исследовательский противочумный институт «Микроб» Роспотребнадзора
Email: davidova_n_work@mail.ru
ORCID iD: 0000-0003-0954-5683
кандидат биол. наук, заведующий отделом микробиологии
Россия, 410005, СаратовВладимир Викторович Кутырев
ФКУН «Российский научно-исследовательский противочумный институт «Микроб» Роспотребнадзора
Email: rusrapi@mail.ru
ORCID iD: 0000-0003-3788-3452
академик РАН, профессор, директор
Россия, 410005, СаратовСписок литературы
- Савицкая Т.А., Иванова А.В., Исаева Г.Ш., Решетникова И.Д., Трифонов В.А., Зиатдинов В.Б. и др. Анализ эпидемиологической ситуации по геморрагической лихорадке с почечным синдромом в Российской Федерации в 2022 г. и прогноз ее развития на 2023 г. Проблемы особо опасных инфекций. 2023; (1): 85–95. https://doi.org/10.21055/0370-1069-2023-1-85-95 https://elibrary.ru/mgxnza
- Иванова А.В., Сафронов В.А., Попов Н.В., Куклев Е.В. Эпидемиологическое районирование территории Приволжского федерального округа по уровню потенциальной эпидемической опасности природных очагов геморрагической лихорадки с почечным синдромом. Проблемы особо опасных инфекций. 2020; (1): 91–6. https://doi.org/10.21055/0370-1069-2020-1-91-96 https://elibrary.ru/jrywjk
- Чумачкова Е.А., Иванова А.В., Поршаков А.М., Вяткин И.Н., Форостяная М.В., Чумачков К.Я. и др. Районирование территории Саратовской области по интенсивности эпидемических проявлений ГЛПС с использованием ГИС-анализа. Проблемы особо опасных инфекций. 2023; (3): 156–63. https://doi.org/10.21055/0370-1069-2023-3-156-163 https://elibrary.ru/dwrlpq
- Tkachenko E.A., Ishmukhametov A.A., Dzagurova T.K., Bernshtein A.D., Morozov V.G., Siniugina A.A., et al. Hemorrhagic fever with renal syndrome, Russia. Emerg. Infect. Dis. 2019; 25(12): 2325–8. https://doi.org/10.3201/eid2512.181649
- Ишмухаметов А.А., Дзагурова Т.К., Морозов В.Г., Курашова С.С., Баловнева М.В., Соцкова С.Е. и др. Характеристика хантавирусов – возбудителей зоонозных геморрагических лихорадок. Эпидемиология и вакцинопрофилактика. 2017; 16(3): 26–32. https://doi.org/10.31631/2073-3046-2017-16-3-26-32 https://elibrary.ru/yrhmch
- ICTV. Taxonomy Browser. Available at: https://ictv.global/taxonomy
- Kabwe E., Davidyuk Y., Shamsutdinov A., Garanina E., Martynova E., Kitaeva K., et al. Orthohantaviruses, emerging zoonotic pathogens. Pathogens. 2020; 9(9): 775. https://doi.org/10.3390/pathogens9090775
- Яшина Л.Н., Трегубчак Т.В., Малышев Б.С., Сметанникова Н.А., Грищенко И.В., Дольский А.А. и др. Возбудитель вспышки ГЛПС в Саратовской области, 2019 г. Проблемы особо опасных инфекций. 2021; (4): 150–6. https://doi.org/10.21055/0370-1069-2021-4-150-156 https://elibrary.ru/dxxsey
- Davidyuk Y.N., Kabwe E., Shamsutdinov A.F., Knyazeva A.V., Martynova E.V., Ismagilova R.K., et al. The Distribution of Puumala orthohantavirus genome variants correlates with the regional landscapes in the Trans-Kama area of the Republic of Tatarstan. Pathogens. 2021; 10(9): 1169. https://doi.org/10.3390/pathogens10091169
- Kabwe E., Shamsutdinov A.F., Suleimanova S., Martynova E.V., Ismagilova R.K., Shakirova V.G., et al. Puumala orthohantavirus reassortant genome variants likely emerging in the watershed forests. Int. J. Mol. Sci. 2023; 24(2): 1018. https://doi.org/10.3390/ijms24021018
- Kabwe E., Al Sheikh W., Shamsutdinov A.F., Ismagilova R.K., Martynova E.V., Ohlopkova O.V., et al. Analysis of Puumala orthohantavirus genome variants identified in the territories of Volga Federal District. Trop. Med. Infect. Dis. 2022; 7(3): 46. https://doi.org/10.3390/tropicalmed7030046
- Blinova E., Deviatkin A., Makenov M., Popova Y., Dzagurova T. Evolutionary formation and distribution of Puumala virus genome variants, Russia. Emerg. Infect. Dis. 2023; 29(7): 1420–4. https://doi.org/10.3201/eid2907.221731
- Castel G., Chevenet F., Razzauti M., Murri S., Marianneau P., Cosson J.F., et al. Phylogeography of Puumala orthohantavirus in Europe. Viruses. 2019; 11(8): 679. https://doi.org/10.3390/v11080679
- Souza W.M., Bello G., Amarilla A.A., Alfonso H.L., Aquino V.H., Figueiredo L.T. Phylogeography and evolutionary history of rodent-borne hantaviruses. Infect. Genet. Evol. 2014; 21: 198–204. https://doi.org/10.1016/j.meegid.2013.11.015
- Ramsden C., Holmes E.C., Charleston M.A. Hantavirus evolution in relation to its rodent and insectivore hosts: no evidence for codivergence. Mol. Biol. Evol. 2009; 26(1): 143–53. https://doi.org/10.1093/molbev/msn234
Дополнительные файлы