Occurrence in sick animals and genetic heterogeneity of Siberian isolates of bovine respiratory syncytial virus (Pneumoviridae: Orthopneumovirus; BRSV) identified in the territories of the Ural, Siberian Federal District and the Republic of Kazakhstan
- Authors: Glotov A.G.1, Yuzhakov A.G.2, Glotova T.I.1, Nefedchenko A.V.1, Koteneva S.V.1, Komina A.K.2, Zhukova E.V.2
-
Affiliations:
- Siberian Federal Scientific Centre of Agro-Biotechnologies of the Russian Academy of Science, Institute of Experimentally Veterinary Medicine of Siberia and Far East
- Federal Scientific Center All-Russian Research Institute of Experimental Veterinary named after the honorary K.I. Scriabin and Y.R. Kovalenko of the Russian Academy of Sciences
- Issue: Vol 69, No 1 (2024)
- Pages: 76-87
- Section: ORIGINAL RESEARCH
- URL: https://virusjour.crie.ru/jour/article/view/16602
- DOI: https://doi.org/10.36233/0507-4088-216
- EDN: https://elibrary.ru/eborho
- ID: 16602
Cite item
Full Text
Abstract
Introduction. Bovine respiratory syncytial virus (Pneumoviridae: Orthornavirae, Orthopneumovirus; Bovine orthopneumovirus, Bovine respiratory syncytial virus, BRSV) is one of causative agents of respiratory diseases in animals. The study of the occurrence and genetic diversity of this pathogen is of particular importance.
Objective. To study the frequency of virus in animals using RT-PCR and genetic heterogeneity of isolates based on determining the complete nucleotide sequence of glycoprotein G gene.
Materials and methods. A 381-bp region of glycoprotein F gene was used for identification of virus genome, while complete nucleotide sequences of G gene were used for phylogenetic analysis. Phylogenetic trees were constructed using the maximum likelihood method in MEGA 7.0 software.
Results. During outbreaks of respiratory diseases, BRSV RNA was detected in animals of all ages in samples of lungs, nasal secretions, pulmonary lymph nodes. Complete nucleotide sequences of glycoprotein G gene, 771 bp in length were obtained for five isolates and 789 bp in length ‒ for two isolates. Nucleotide similarity between them was 87–100%. Phylogenetic analysis assigned the isolates to subgroups II and III, each of which included two isolates. A separate clade formed by K18 isolate from animals imported from Canada and sequences from vaccines containing the attenuated «375» strain.
Conclusion. The virus genome was identified in cows and heifers (20.0 and 14.3%), in calves up 1 month of age (3.05%), and in calves from 1 to 6 months of age (6.7%). Complete G gene nucleotide sequence analysis is a useful tool for studying the molecular epidemiology of BRSV on particular territories.
Keywords
Full Text
Введение
Респираторные болезни причиняют значительный экономический ущерб молочному и мясному скотоводству, вызывая гибель или снижение скорости роста животных, увеличивая затраты на лечение, проведение диагностических и профилактических мероприятий [1, 2]. Одним из этиологических агентов, вызывающих инфекционную патологию органов дыхания, является респираторно-синцитиальный вирус крупного рогатого скота (Bovine respiratory syncytial virus, BRSV, Bovine orthopneumovirus), широко распространенный во всех странах мира с развитым типом ведения животноводства [3‒6].
BRSV относится к семейству Pneumoviridae, роду Orthopneumovirus и представляет собой полиморфный оболочечный вирус с одноцепочечной РНК отрицательной полярности размером ~15,2 kb, которая кодирует не менее 10 протеинов [7, 8].
Гликопротеин G ‒ самый большой среди известных белков вируса, он является основным вирусным антигеном, который индуцирует иммунный ответ хозяина и используется в качестве мишени для филогенетических и молекулярно-эпидемиологических исследований. Считается, что белки F и G играют важную роль в инфекционности возбудителя и являются основными белками, участвующими в репликации вируса [9].
К инфицированию вирусом BRSV восприимчив крупный рогатый скот всех пород и возрастов, однако чаще болеют телята от 4 нед до года, иногда – с рождения до 2-недельного возраста. Описано синергетическое взаимодействие агента с бактериями семейства Pasteurellaceae, а также другими респираторными вирусами [10, 11]. Штаммы вируса различаются по вирулентности [9].
BRSV непрерывно эволюционирует в ответ на воздействие иммунной системы организма животного и вакцинации, а патогенез болезни и характер эпизоотической ситуации во многом зависят от антигенной изменчивости вируса, обусловленной мутациями в составе протеинов, особенно гликопротеина G [12‒17].
Молекулярные исследования подтвердили существование антигенной дивергенции и генетической вариабельности среди полевых изолятов вируса. Штаммы вируса, выделенные от животных одного стада во время первичных вспышек болезни, идентичны, в отличие от штаммов, выделенных при повторных вспышках болезни в стадах, куда новые животные не вводились 10 лет. Различия в генетической структуре между ними могут достигать 11% [12].
Белок G содержит три домена: цитоплазматический, расположенный между аминокислотами (а.к.) 1‒37; трансмембранный (38‒65 а.к.); внеклеточный, или эктодомен (66‒257 а.к.) [18]. Этот последний домен содержит высококонсервативную гидрофобную центральную область из 32 a.к. и четырех цистеинов, которые образуют два дисульфидных мостика [18, 19]. Из-за высокой генетической изменчивости (до 8%) G-белок может быть использован для эволюционного анализа штаммов BRSV [20]. Первоначально на основе анализа гена G штаммы BRSV были разделены на четыре подгруппы, обозначенные как A, B, промежуточные (или AB) и нетипированные [9, 12].
В настоящее время все известные штаммы вируса классифицируются на подгруппы I‒X [12, 18, 21‒23]. Подгруппа I состоит из европейских штаммов, выделенных до 1976 г. [12]. Штаммы из Италии, Бельгии, Нидерландов, Дании и Швеции сгруппированы в подгруппу II [13, 21, 23, 24]. Подгруппа III изначально включала исключительно вирусы из США [4, 14], однако позднее штаммы этой подгруппы были обнаружены в 8 провинциях Китая [25, 26], в Турции и Бразилии [14, 27]. Представители подгруппы IV циркулируют в Германии, Бельгии, Дании, Америке и других европейских странах [21, 28, 29]. Штаммы из Бельгии и Франции включены в подгруппы V и VI [18]. Изоляты из Хорватии были отнесены к подгруппам VII и VIII [30]. В недавних исследованиях были выявлены подгруппа IX в Бразилии [14, 31] и подгруппа X в Японии [32].
Несмотря на то что респираторно-синцитиальная инфекция крупного рогатого скота имеет широкое распространение на территории Российской Федерации, особенно на молочных комплексах с наличием высокопродуктивного импортированного поголовья, исследования по филогенетическому анализу изолятов вируса не проводились.
В связи с этим целью настоящей работы являлось изучение частоты выявления BRSV у больных животных и генетического полиморфизма его изолятов, циркулирующих среди высокопродуктивного молочного скота в Сибири, на основе секвенирования полной нуклеотидной последовательности гена гликопротеина G.
Материалы и методы
Исследования проводили в течение 5 лет (2018‒2022 гг.) на крупных молочных комплексах и молочно-товарных хозяйствах, расположенных в Тюменской, Омской, Томской, Новосибирской, Иркутской областях, Алтайском и Красноярском краях РФ и Республике Казахстан в период вспышек массовых желудочно-кишечных и респираторных болезней животных. На момент исследований вакцинация против респираторно-синцитиальной инфекции крупного рогатого скота не проводилась. Работу выполняли во время эпизоотологических обследований на наличие возбудителей вирусных и бактериальных инфекций крупного рогатого скота. Всего исследовали 1012 проб биоматериала от вынужденно убитых и павших телят в возрасте до 6 мес, а также коров и нетелей с признаками острых респираторных заболеваний.
От каждого животного отбирали пробы органов респираторного тракта, доставляли в лабораторию в замороженном состоянии или транспортной среде в течение не более 12 ч с момента отбора, где хранили при −80 °С. Перед исследованием образцы гомогенизировали и готовили 10% суспензии на физиологическом растворе, которые центрифугировали в течение 15 мин при 3000 об/мин. Для выделения РНК использовали по 100 мкл осветленного супернатанта.
Исследованный материал включал: 493 пробы легких, 99 – легочных лимфатических узлов, 83 – носовых выделений, 32 – трахеального и бронхиального экссудатов, 120 – слизистой оболочки носа, 165 – слизистой оболочки трахеи и бронхов, 20 – бронхов от больных и вынужденно убитых животных.
В настоящей работе определяли полную нуклеотидную последовательность гена G 5 изолятов, выделенных от больных животных, а также двух вакцинных штаммов, входящих в состав вакцин BoviSchieldGoldFP5 и CattleMaster GoldFP5. Характеристика штаммов приведена в табл. 1.
Таблица 1. Изоляты BRSV, использованные для филогенетического анализа
Table 1. BRSV isolates used for phylogenetic analysis
Название (номер в GenBank) Name (GenBank number) | Биоматериал Biomaterial | Возрастная группа животных Animal age group | Регион Region | Дата поступления Receipt date |
NSO1 (OR426499) | Легкие, бронхи Lungs, bronchi | Теленок Calf | Новосибирская область Novosibirsk | 15.05.2018 |
NSO2 (OR426500) | Слизистая носа, Легкие Nasal mucosa, lungs | Теленок Calf | Новосибирская область Novosibirsk | 18.04.2018 |
Alt3 (OR426501) | Легкие, трахея Lungs, trachea | Теленок Calf | Алтайский край Altai | 25.03.2021 |
Alt4 (OR426502) | Носовые выделения Nasal discharge | Корова Cow | Алтайский край Altai | 11.04.2020 |
K18 (OR426503) | Легкие Lungs | Изолят К18. Выделен от нетели в первично-трипсинизированной культуре клеток текстикул бычков Isolate K18. Isolated from a heifer in a primary TB cell culture | Республика Казахстан The Republic of Kazakhstan | Хранился в лиофилизированном виде с мая 2008 г. Stored in lyophilized form since May 2008 |
FP5L5HB (OR426504) | Вакцина Бови-Шилд Голд FP5L5 HB Vaccine Bovi‒Shield Gold FP5L5 HB | Аттенуированный штамм 375, выделен от теленка Attenuated strain 375, isolated from a calf | США, 1979 USA, 1979 | Положительный контроль. Хранился в лиофилизированном виде с мая 2015 г. Positive control. Stored in lyophilized form since May 2015 |
FP5L5 (OR426505) | Вакцина Кэтлмастер Голд FP5L5 Vaccine CattleMaster Gold FP5L5 HB | Аттенуированный штамм 375, выделен от теленка Attenuated strain 375, isolated from a calf | США, 1979 USA, 1979 | Положительный контроль. Хранился в лиофилизированном виде с мая 2015 г. Positive control. Stored in lyophilized form since May 2015 |
Экстракция РНК и обратная транскрипция
РНК выделяли из 100 мкл гомогената органов с помощью набора «РИБО-преп» (ЦНИИ Эпидемиологии Роспотребнадзора, Россия) в соответствии с рекомендациями производителя. Очищенную РНК ресуспендировали в 50 мкл РНК-буфера. Для проведения обратной транскрипции (ОТ) использовали 10 мкл выделенной РНК. Реакцию проводили с помощью набора «Реверта-L» (ЦНИИ Эпидемиологии Роспотребнадзора, Россия) в соответствии с рекомендациями производителя. После проведения реакции объем пробы составлял 40 мкл.
Выявление BRSV с помощью ПЦР
Пробы биоматериала исследовали на наличие генома BRSV методом полимеразной цепной реакции (ПЦР) с использованием тест-системы, разработанной нами ранее [33].
ПЦР-амплификация гена G
Для амплификации и последующего секвенирования полной нуклеотидной последовательности гена гликопротеина G разработали две перекрывающиеся пары праймеров:
F1 (GTTACATACAGATGTTGGGGC) и R1 (GTTTGGGAGTTGTTGTGGTC); F2 (GGGAAATGCTAAAGCCAAGCC) и R2 (CCATCCTTATTTGCCCCAG).
Состав реакционной смеси: ПЦР-буфер (Bioron), 0,6 мкл 50× dNTP, по 0,5 мкг каждого праймера в концентрации 100 мкМ, 1 мкл 100 мМ MgCl2, 1,5 еа Taq-ДНК-полимеразы (Bioron), 5 мкл ДНК. Общий объем – 30 мкл. Температурный режим для ПЦР: 95 °С – 5 мин, 1 цикл; 95 °С – 30 с, 57 °С – 60 с, 72 °С – 60 с, 45 циклов; 72°С – 5 мин, 1 цикл. Выделение ампликонов из 1% агарозного геля проводили по методике производителя коммерческого набора для очистки ДНК из агарозного геля и реакционных смесей «Евроген» («Евроген», Россия).
Нуклеотидную последовательность определяли при помощи секвенирования по методу Сэнгера с использованием набора Big Dye Terminator v. 3.1 Cycle Sequencing Kit (Applied Biosystems, США) согласно инструкции изготовителя. Полученные нуклеотидные последовательности анализировали при помощи пакетов программ UGENE (v. 45.1). Выравнивание последовательностей проводили с использованием метода ClustalW. Филогенетическая дендрограмма была построена по методу максимального правдоподобия в программе MEGA 7.0. Топологию ветвей дендрограммы подтверждали методом бутстрэп-анализа (1000 шагов репликации). Для построения деревьев использовали модель General Time Reversible (GTR), гамма-распределение вариации частот между сайтами (G + I).
Авторы подтверждают соблюдение институциональных и национальных стандартов по использованию лабораторных животных в соответствии с Consensus author guidelines for animal use (IAVES 23 July 2010). Протокол исследования одобрен Этическим комитетом СФНЦА РАН (Протокол № 4 от 14.06. 2023).
Результаты
Результаты исследований представлены в табл. 2. Наиболее часто геном BRSV выявляли у коров и нетелей (20 и 14,3%) с признаками острых респираторных заболеваний, интерстициальной пневмонией и эмфиземой легких. У телят РНК вируса обнаруживали чаще в возрастной группе от 1 до 6 мес (6,7%), до 1 мес – 3,05%. У BRSV-позитивных телят в возрасте от одного до 6 мес при клиническом осмотре нередко отмечали признаки сверхострой формы инфекции: угнетение, отказ от корма, повышение температуры тела, учащенное дыхание брюшного типа с открытым ртом и высунутым языком, опущенные вниз шея и голова, выделение обильной слюны из ротовой полости. При патологоанатомическом вскрытии у них регистрировали интерстициаль- ную и легочную эмфизему и разрушение паренхимы легкого.
Таблица 2. Частота выявления BRSV в пробах биоматериала от животных разных половозрастных групп (n = 1012)
Table 2. BRSV detection rates in biomaterial samples from animals of different sex and age groups (n = 1012)
Половозрастная группа животных Sex and age group of animals | Число исследованных/положительных проб Number of samples/positive examined | Количество положительных проб от числа исследованных, % Number of positive samples out of the number of tested samples, % |
Телята / Calves: | ||
от 10 дней до 1 мес from 10 days to 1 month | 328/10 | 3,05 |
от 1 до 6 мес from 1 up to 6 months | 555/37 | 6,7 |
Нетели Heifers | 14/2 | 14,3 |
Коровы Cows | 115/23 | 20,0 |
Всего Total | 1012/72 | 7,1 |
Результаты исследования частоты выявления возбудителя в пробах биоматериала разного происхождения приведены в табл. 3. Вирус чаще выявляли в пробах трахеального и бронхиального экссудатов (40,6%) и бронхов (25,0%), реже – в пробах легких (7,5%), носовых выделений (7,2%), слизистой оболочки трахеи и бронхов (3,6%), легочных лимфатических узлов (3,0%) и слизистой оболочки носа (1,7%) животных с наличием признаков острых респираторных болезней.
Таблица 3. Частота выявления BRSV методом ПЦР в пробах биоматериала различного происхождения (n = 1012)
Table 3. Frequency of BRSV detection by PCR in biomaterial samples of various origin (n = 1012)
Вид биоматериала Type of biomaterial | Количество исследованных/положительных проб Number of samples/positive examined | Количество положительных проб от числа исследованных, % Number of positive samples out of the number of tested samples, % |
Легкие Lungs | 493/37 | 7,5 |
Легочные лимфатические узлы Lungs lymph nodes | 99/3 | 3,0 |
Носовые выделения Nasal discharges | 83/6 | 7,2 |
Экссудат из трахеи и бронхов Exudate from the trachea and bronchi | 32/13 | 40,6 |
Слизистая оболочка носа Nasal mucosa | 120/2 | 1,7 |
Слизистая оболочка трахеи и бронхов Trachea and bronchi mucosa | 165/6 | 3,6 |
Бронхи Bronchi | 20/5 | 25,0 |
Всего Total | 1012/72 | 7,1 |
Результаты проведенных исследований показали, что к инфицированию вирусом восприимчивы все половозрастные группы животных. В среднем вирус присутствовал в 7,1% исследованных проб биоматериала, полученных от крупного рогатого скота при вспышках болезни. Были получены полные нуклеотидные последовательности гена гликопротеина G изолятов NSO1, NSO2, K18, FP5L5HB, FP5L5 размером 771 п.н. и изолятов Alt3, Alt4 размером 789 п.н. Нуклеотидное сходство 7 исследуемых изолятов варьировало от 87‒100%. Полученные последовательности были депонированы в базу данных GenBank под номерами OR426499‒OR426505.
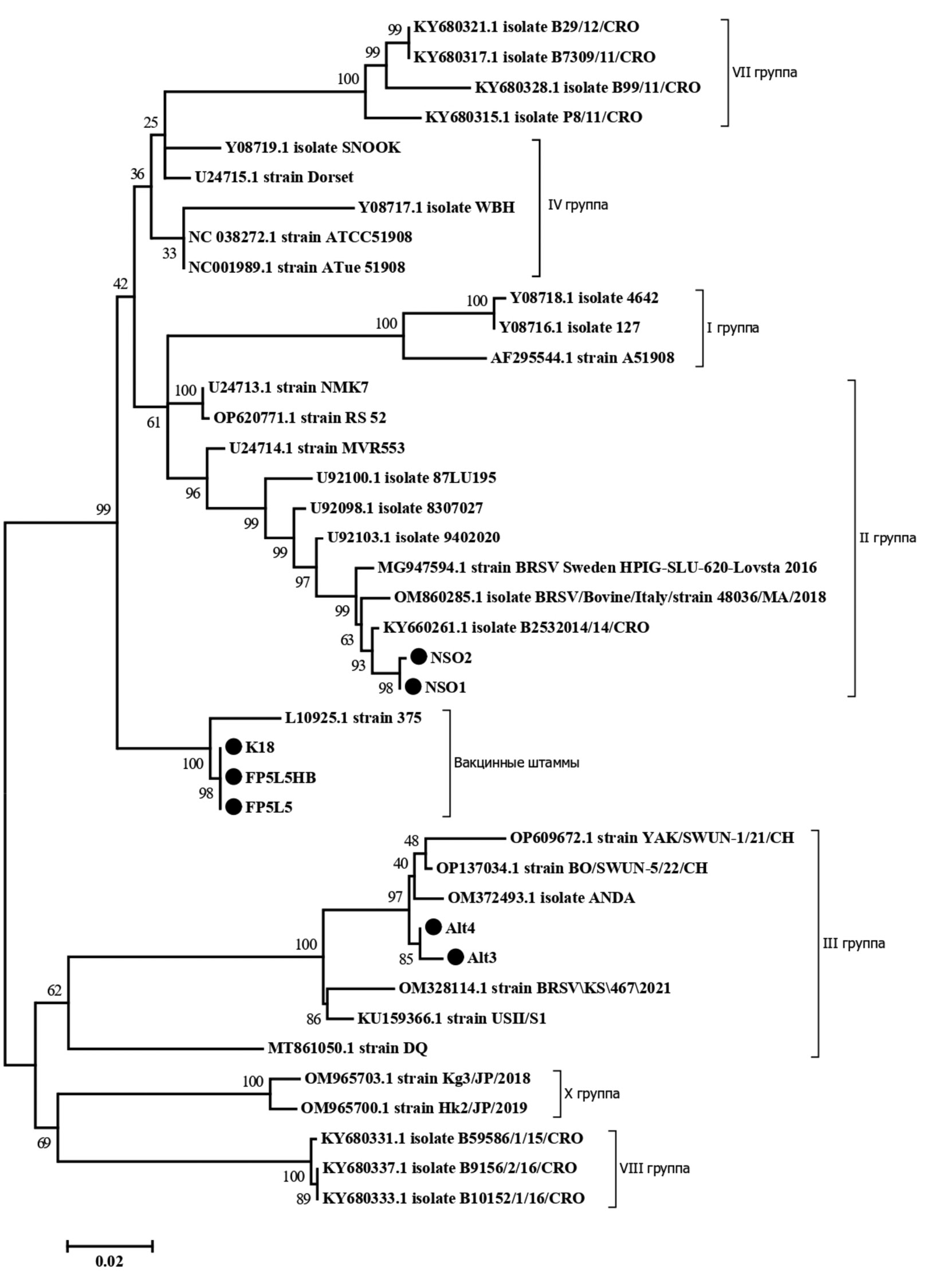
На основе полученных последовательностей, а также последовательностей из базы данных GenBank было построено филогенетическое дерево (рис. 1).
Рис. 1. Филогенетическое дерево, построенное на основе полной нуклеотидной последовательности гена G BRSV. Последовательности, полученные в данном исследовании, отмечены ●.
Fig. 1. Phylogenetic tree based on the complete nucleotide sequence of the BRSV G gene. The sequences obtained in this study are marked with ●
Результаты филогенетического анализа показали, что 7 полученных последовательностей входят в две разные подгруппы: II и III. Изоляты NSO1 и NSO2 сгруппировались в одну кладу с европейскими изолятами из Хорватии ‒ B2532014/14/CRO (KY660261), Италии ‒ 48036/MA/2018 (OM860285), Швеции ‒ HPIG-SLU-620-Lovsta 2016 (MG947594), относящимися к подгруппе II. Нуклеотидное сходство изолятов NSO1 и NSO2 с хорватским штаммом составило 99,09%, со шведским ‒ 98,44%, а с итальянским ‒ 98,31%. В последовательности гена G изолятов NSO1 и NSO2 обнаружены нуклеотидные мутации относительно других представителей подгруппы II, приводящие к ряду уникальных аминокислотных замен 4 (His→Arg), 55 (Val→Ile), 181 (Ala→Thr). Также у изолята NSO2 присутствует замена 66 (Asn→Lys) (рис. 2).
Рис. 2. Множественное выравнивание аминокислотных последовательностей G-белка между изолятами NSO1, NSO2, Alt3, Alt4, K18, FP5L5HB, FP5L5 и эталонными штаммами BRSV, опубликованными в GenBank.
Fig. 2. Multiple alignment of G protein amino acid sequences of isolates NSO1, NSO2, Alt3, Alt4, K18, FP5L5HB, FP5L5 and BRSV reference strains deposited in GenBank.
Отдельную кладу образовали изолят K18, выделенный от животных, завезенных из Канады, а также образцы вакцин FP5L5HB (вакцина Бови-Шилд Голд) и FP5L5 (вакцина Кэтлмастер Голд) и штамм 375 (L10925), входящий в состав разных вакцин. Нуклеотидное сходство между изолятами K18, FP5L5HB и FP5L5 составило 100%, а со штаммом 375 – 98%. Нуклеотидные мутации привели к аминокислотным заменам относительно эталонного штамма 375: 13 (Gly→Leu), 19 (Pro→Ala), 41 (Thr→Lys), 66 (Lys→Asn), 67 (Ser→Ala), 139 (Ala→Thr), 171 (Glu→Val), 213 (Arg→Lys). Данные представлены на рис. 2.
Изоляты Alt3 и Alt4, выделенные нами в Алтайском крае, были отнесены к подгруппе III. Наиболее близкими к алтайским изолятам оказались китайские штаммы BO/SWUN-5/22/CH (OP137034), YAK/SWUN-1/21/CH (OP609672) и американский изолят ANDA (OM372493), нуклеотидное сходство которых составило 98,73‒97,34%. Кроме того, отдельными соседними ветками в этой кладе выделяются американский штамм USII/S1 (KU159366) и китайский штамм DQ (MT861050). В последовательностях изолята Alt3 были обнаружены уникальные замены остатков 39 (Val→Gly) и 42 (Ala→Asp). Относительно других штаммов BRSV у изолятов Alt3 и Alt4 обнаружена аминокислотная замена 116 (Thr→Ile). Данные представлены на рис. 2.
Обсуждение
Нами исследованы образцы из 8 регионов Уральского, Сибирского ФО РФ и Республики Казахстан, отобранные от животных из хозяйств с различным типом ведения животноводства при массовых вспышках острых респираторных заболеваний. В часть из этих регионов осуществлялся завоз высокопродуктивных животных из стран Европы и Северной Америки, но большая часть проб была получена от аборигенного скота. Результаты исследования показали, что к инфицированию вирусом восприимчивы все половозрастные категории крупного рогатого скота. Участие респираторно-синцитиального вируса в этиологии массовых респираторных болезней было подтверждено в 20 и 14,3% случаев у коров и нетелей, в 3,05% случаев у телят в возрасте до 1 мес и в 6,7% случаев у телят в возрасте 1—6 мес.
В результате работы были выделены 5 изолятов вируса, которые использовали для секвенирования полной нуклеотидной последовательности гена гликопротеина G.
Изоляты NSO1, NSO2 были выявлены в 2018 г. в органах респираторного тракта телят с признаками острого респираторного заболевания в двух хозяйствах Новосибирской области и отнесены нами к подгруппе II штаммов BRSV, которую представляют референтные штаммы, обнаруженные, в частности, в Швеции (MG947594) и Дании во время вспышек респираторных болезней, а также в Швеции и Норвегии в 2010‒2011 гг., сопровождавшихся высокой заболеваемостью и летальностью животных [24]. Сюда же относится высоковирулентный итальянский штамм 48036/MA/2018, вызвавший вспышку болезни в невакцинированном стаде животных. Штамм был ассоциирован с тяжелым течением респираторной инфекции, что привело к значительному экономическому ущербу. Экстренная иммунизация животных живой вакциной, вводимой интраназально, к успеху не привела [21]. Наши изоляты также были выявлены у животных, которые не подвергались иммунизации против данной BRSV-инфекции.
К отдельной кладе мы отнесли высоковирулентный изолят K18, выделенный от больных нетелей, завезенных из Канады, при вспышке массового респираторного заболевания после смешивания их с местным скотом. Вспышка болезни характеризовалась высокими показателями заболеваемости и летальности животных всех возрастов. По данным J. Valarcher и соавт. (2000), R. Leme и соавт. (2020), S. Jia и соавт. (2021) штамм 375 Lehmkuhl и его аттенуированные варианты 375.1 и 375.2, входящие в состав использованных нами вакцин, относятся к подгруппе III [14, 18, 25]. Наши результаты не согласуются с этими утверждениями, так как упомянутые вакцинные штаммы и изолят К18 распределились в отдельную кладу и значительно отличаются от штаммов подгрупп II и III. Возможно, это связано с нуклеотидными мутациями, которые привели к аминокислотным заменам относительно эталонного штамма 375: 13 (Gly→Leu), 19 (Pro→Ala), 41 (Thr→Lys), 66 (Lys→Asn), 67 (Ser→Ala), 139 (Ala→Thr), 171 (Glu→Val), 213 (Arg→Lys).
В ряде научных публикаций сообщали о широком распространении штаммов, относящихся к подгруппе III, в Америке, Турции, Бразилии и Китае [4, 22, 25, 31]. Штамм USII/S1 связывают со вспышками респираторных заболеваний в Америке в 2015 г., которые принесли огромные экономические потери животноводству [4]. Также при вспышке острого респираторного заболевания среди телят на китайских фермах в образцах легочной ткани был обнаружен штамм DQ, относящийся к подгруппе III [26]. Последовательности, кодирующие белок G изолятов Alt3 и Alt4 длиной 789 п.н., кодировали 263 аминокислоты, что характерно для всех представителей подгруппы III. Замены аминокислот относительно эталонного американского штамма USII/S1 (KU159366) были идентичны мутациям, обнаруженным у китайских изолятов (OM372493, OP137034, OP609672).
Антигенные вариации в основном поверхностном гликопротеине белка прикрепления G могут иметь важные последствия в эпизоотологии и патогенезе BRSV-инфекций [34]. Исследования показали, что аминокислотные остатки 158‒189 представляют центральную консервативную область белка G BRSV, а остатки 174‒187 центральной консервативной области являются иммунодоминантными [19, 23]. Уникальная аминокислотная замена у изолятов NSO1 и NSO2 в иммунодоминантной области 181 (Ala→Thr) отмечена только у высоковирулентного штамма DQ (MT861050), однако они относятся к разным подгруппам. По данным J.P. Langedijk и соавт. [35], Ala181 является важной аминокислотой для связывания антител, поэтому влияние этой аминокислотной мутации на антигенность белка G нуждается в дальнейшем изучении.
Все изоляты вируса, использованные нами в нстоящей работе, были выделены от больных животных с клиническими признаками острых респираторных заболеваний. У больных животных при клиническом осмотре наблюдали признаки острой формы инфекции: угнетение, отказ от корма, повышение температуры тела, учащенное дыхание брюшного типа с открытым ртом и высунутым языком, опущенные вниз шея и голова, выделение обильной слюны из ротовой полости. При патологоанатомическом вскрытии у них регистрировали интерстициальную, легочную эмфизему, а в некоторых случаях разрушение паренхимы легкого.
Заключение
Популяция сибирских изолятов вируса BRSV представлена двумя подгруппами и одной независимой кладой. Полученные данные показывают, что полный анализ нуклеотидной последовательности гена G является полезным инструментом для изучения молекулярной эпизоотологии респираторно-синцитиальной инфекции крупного рогатого скота. Исследования по молекулярной эпизоотологии этого вируса в конкретном регионе можно использовать с целью оптимизации и выбора стратегии контрольных мероприятий на региональном уровне и решения вопроса о применении вакцин. Это особенно важно при реализации программ вакцинации животных, когда генетические типы вакцинных штаммов не совпадают с типами, циркулирующими среди животных на конкретной территории. Полученная в ходе таких исследований информация может быть полезной при изучении молекулярной эпизоотологии вирусов, разработке более точных диагностических тестов, эффективных вакцин и программ контроля инфекции. Незначительное количество изолятов, использованных в работе, связано с биологическими особенностями вируса: короткий «транзитный» период нахождения в органах респираторного тракта животных, низкие концентрации в тканях респираторного тракта, трудности культивирования в клеточных системах и т.д. Однако полученная в ходе исследований информация с использованием 7 изолятов вируса дает представления о гетерогенности популяции BRSV на территории Сибири и Республики Казахстан. Ранее нами было изучено распространение респираторно-синцитиальной инфекции на молочных комплексах этих регионов [36], разработаны различные варианты ПЦР для выявления и типирования возбудителя в пробах органов от животных [37, 38]. Это первое исследование по определению полной нуклеотидной последовательности гена G респираторно-синцитиального вируса крупного рогатого скота в России. Исследования в этом направлении будут продолжены.
About the authors
Alexander G. Glotov
Siberian Federal Scientific Centre of Agro-Biotechnologies of the Russian Academy of Science, Institute of Experimentally Veterinary Medicine of Siberia and Far East
Author for correspondence.
Email: glotov_vet@mail.ru
ORCID iD: 0000-0002-2006-0196
D. Sci. (Vet.), Professor, Chief Researcher – Head of Biotechnology Laboratory, Diagnostic Center, Siberian Federal Scientific Center of Agrobiotechnologies (SFSCA) of the Russian Academy of Sciences, Institute of Experimental Veterinary Medicine of Siberia and the Far
Russian Federation, 630501, Krasnoobsk, Novosibirsk regionAnton G. Yuzhakov
Federal Scientific Center All-Russian Research Institute of Experimental Veterinary named after the honorary K.I. Scriabin and Y.R. Kovalenko of the Russian Academy of Sciences
Email: anton_oskol@mail.ru
ORCID iD: 0000-0002-0426-9678
Ph.D. biol. Sciences, head of laboratory, Federal Scientific Center – All-Russian Research Institute of Experimental Veterinary Medicine named after K.I. Scriabin and Y.R. Kovalenko of the Russian Academy of Sciences
Russian Federation, 109428, MoscowTatyana I. Glotova
Siberian Federal Scientific Centre of Agro-Biotechnologies of the Russian Academy of Science, Institute of Experimentally Veterinary Medicine of Siberia and Far East
Email: t-glotova@mail.ru
ORCID iD: 0000-0003-3538-8749
SPIN-code: 7488-5915
Sci. (Biol.), Professor, Chief Researcher – Biotechnology Laboratory-Diagnostic Center Siberian Federal Scientific Center of Agrobiotechnologies (SFSCA) of the Russian Academy of Sciences, Institute of Experimental Veterinary Medicine of Siberia and the Far East
Russian Federation, 630501, Krasnoobsk, Novosibirsk regionAlexey V. Nefedchenko
Siberian Federal Scientific Centre of Agro-Biotechnologies of the Russian Academy of Science, Institute of Experimentally Veterinary Medicine of Siberia and Far East
Email: homeovet@narod.ru
ORCID iD: 0000-0002-4181-4268
SPIN-code: 1583-5776
D. Sci. (Vet.), Associate Professor, Leading Researcher of the laboratory of biotechnology – diagnostic center of the Siberian Federal Scientific Center of Agrobiotechnologies (SFSCA) of the Russian Academy of Sciences, Institute of Experimental Veterinary Medicine of Siberia and the Far East
Russian Federation, 630501, Krasnoobsk, Novosibirsk regionSvetlana V. Koteneva
Siberian Federal Scientific Centre of Agro-Biotechnologies of the Russian Academy of Science, Institute of Experimentally Veterinary Medicine of Siberia and Far East
Email: koteneva-sv@mail.ru
ORCID iD: 0000-0003-2649-7505
SPIN-code: 7545-7206
Ph.D. Sci (Vet.), leading researcher at the laboratory of biotechnology-diagnostic center of the Siberian Federal Scientific Center for Agrobiotechnologies (SFSCA) of the Russian Academy of Sciences, Institute of Experimental Veterinary Medicine of Siberia and the Far East
Russian Federation, 630501, Krasnoobsk, Novosibirsk regionAlina K. Komina
Federal Scientific Center All-Russian Research Institute of Experimental Veterinary named after the honorary K.I. Scriabin and Y.R. Kovalenko of the Russian Academy of Sciences
Email: komina.a.k@yandex.ru
ORCID iD: 0000-0002-7173-5501
SPIN-code: 3699-2612
graduate student, Federal Scientific Center – All-Russian Research Institute of Experimental Veterinary Medicine named after K.I. Scriabin and Y.R. Kovalenko of the Russian Academy of Sciences
Russian Federation, 109428, MoscowElena V. Zhukova
Federal Scientific Center All-Russian Research Institute of Experimental Veterinary named after the honorary K.I. Scriabin and Y.R. Kovalenko of the Russian Academy of Sciences
Email: evz-sk@mail.ru
ORCID iD: 0000-0001-7423-6102
Ph.D. biol. Sciences, leading researcher at the Laboratory of Biochemistry and Molecular Biology Federal Scientific Center – All-Russian Research Institute of Experimental Veterinary Medicine named after K.I. Scriabin and Y.R. Kovalenko of the Russian Academy of Sciences
Russian Federation, 109428, MoscowReferences
- Cummings D.B., Meyer N.F., Step D.L. Bovine respiratory disease considerations in young dairy calves. Vet. Clin. North Am. Food Anim. Pract. 2022; 38(1): 93–105. https://doi.org/10.1016/j.cvfa.2021.11.007
- Gorden P.J., Plummer P. Control, management, and prevention of bovine respiratory disease in dairy calves and cows. Vet. Clin. North Am. Food Anim. Pract. 2010; 26(2): 243–59. https://doi.org/10.1016/j.cvfa.2010.03.004
- Valarcher J.F., Taylor G. Bovine respiratory syncytial virus infection. Vet. Res. 2007; 38(2): 153–80. https://doi.org/10.1051/vetres:2006053
- Kirolos A., Christides A., Xian S., Reeves R., Nair H., Campbell H. A landscape review of the published research output relating to respiratory syncytial virus (RSV) in North & Central America and Europe between 2011–2015. J. Glob. Health. 2019; 9(1): 010425. https://doi.org/10.7189/jogh.09.010425
- Renault V., Damiaans B., Sarrazin S., Humblet M.F., Lomba M., Ribbens S., et al. Classification of adult cattle infectious diseases: A first step towards prioritization of biosecurity measures. Transbound. Emerg. Dis. 2018; 65(6): 1991–2005. https://doi.org/10.1111/tbed.12982
- Makoschey B., Berge A.C. Review on bovine respiratory syncytial virus and bovine parainfluenza – usual suspects in bovine respiratory disease – a narrative review. BMC Vet. Res. 2021; 17(1): 261. https://doi.org/10.1186/s12917-021-02935-5
- Amarasinghe G.K., Bào Y., Basler C.F., Bavari S., Beer M., Bejerman N., et al. Taxonomy of the order Mononegavirales: update 2017. Arch. Virol. 2017; 162(8): 2493–504. https://doi.org/10.1007/s00705-017-3311-7
- Rima B., Collins P., Easton A., Fouchier R., Kurath G., Lamb R.A., et al. ICTV Report Consortium. ICTV virus taxonomy profile: Pneumoviridae. J. Gen. Virol. 2017; 98(12): 2912–3. https://doi.org/10.1099/jgv.0.000959.
- Larsen L.E. Bovine respiratory syncytial virus (BRSV): a review. Acta Vet. Scand. 2000; 41(1): 1–24. https://doi.org/10.1186/bf03549652
- Fulton R.W., Purdy C.W., Confer A.W., Saliki J.T., Loan R.W., Briggs R.E., et al. Bovine viral diarrhea viral infections in feeder calves with respiratory disease: interactions with Pasteurella spp., parainfluenza-3 virus, and bovine respiratory syncytial virus. Can. J. Vet. Res. 2000; 64(3): 151–9.
- Guzman E., Taylor G. Immunology of bovine respiratory syncytial virus in calves. Mol. Immunol. 2015; 66(1): 48–56. https://doi.org/10.1016/j.molimm.2014.12.004
- Larsen L.E., Tjørnehøj K., Viuff B. Extensive sequence divergence among bovine respiratory syncytial viruses isolated during recurrent outbreaks in closed herds. J. Clin. Microbiol. 2000; 38(11): 4222–7. https://doi.org/10.1128/jcm.38.11.4222-4227.2000
- Sarmiento-Silva R.E., Nakamura-Lopez Y., Vaughan G. Epidemiology, molecular epidemiology and evolution of bovine respiratory syncytial virus. Viruses. 2012; 4(12): 3452–67. https://doi.org/10.3390/v4123452
- Leme R.A., Dall Agnol A.M., Balbo L.C., Pereira F.L., Possatti F., Alfieri A.F., et al. Molecular characterization of Brazilian wild-type strains of bovine respiratory syncytial virus reveals genetic diversity and a putative new subgroup of the virus. Vet Q. 2020; 40(1): 83–96. https://doi.org/10.1080/01652176.2020.1733704
- Sacco R.E., McGill J.L., Pillatzki A.E., Palmer M.V., Ackermann M.R. Respiratory syncytial virus infection in cattle. Vet. Pathol. 2014; 51(2): 427–36. https://doi.org/10.1177/0300985813501341
- Valentova V. The antigenic and genetic variability of bovine respiratory syncytial virus with emphasis on the G protein. Veterinární medicína. 2012; 48(9): 254–66. https://doi.org/10.17221/5778-VETMED18
- Doreleijers J.F., Langedijk J.P.M., Hård K., Boelens R., Rull- mann J.A.C., Schaaper W.M., et al. Solution structure of the immunodominant region of protein G of bovine respiratory syncytial virus. Biochemistry. 1996; 35(47): 14684–8. https://doi.org/10.1021/bi9621627
- Valarcher J.F., Schelcher F., Bourhy H. Evolution of bovine respiratory syncytial virus. J. Virol. 2000; 74(22): 10714–28. https://doi.org/10.1128/jvi.74.22.10714-10728.2000
- Furze J.M., Roberts S.R., Wertz G.W., Taylor G. Antigenically distinct G glycoproteins of BRSV strains share a high degree of genetic homogeneity. Virology. 1997; 231(1): 48–58. https://doi.org/10.1006/viro.1997.8490
- Krešić N., Bedeković T., Brnić D., Šimić I., Lojkić I., Turk N. Genetic analysis of bovine respiratory syncytial virus in Croatia. Comp. Immunol. Microbiol. Infect. Dis. 2018; 58: 52–7. https://doi.org/10.1016/j.cimid.2018.09.004
- Giammarioli M., Mangili P., Nanni A., Pierini I., Petrini S., Pirani S., et al. Highly pathogenic Bovine Respiratory Syncytial virus variant in a dairy herd in Italy. Vet. Med. Sci. 2020; 6(4): 740–5. https://doi.org/10.1002/vms3.312
- Klem T.B., Rimstad E., Stokstad M. Occurrence and phylogenetic analysis of bovine respiratory syncytial virus in outbreaks of respiratory disease in Norway. BMC Vet. Res. 2014; 10(1): 15. https://doi.org/10.1186/1746-6148-10-15
- Bertolotti L., Giammarioli M., Rosati S. Genetic characterization of bovine respiratory syncytial virus strains isolated in Italy: evidence for the circulation of new divergent clades. J. Vet. Diagn. Invest. 2018; 30(2): 300–4. https://doi.org/10.1177/1040638717746202
- Bidokti M.R., Tråvén M., Ohlson A., Zarnegar B., Baule C., Belák S., et al. Phylogenetic analysis of bovine respiratory syncytial viruses from recent oubreaks in feedlot and dairy cattle herds. Arch. Virol. 2012; 157(4): 601–7. https://doi.org/10.1007/s00705-011-1209-3
- Jia S., Yao X., Yang Y., Niu C., Zhao Y., Zhang X., et al. Isolation, identification, and phylogenetic analysis of subgroup III strain of bovine respiratory syncytial virus contributed to outbreak of acute respiratory disease among cattle in Northeast China. Virulence. 2021; 12(1): 404–14. https://doi.org/10.1080/21505594.2021.1872178
- Chang Y., Yue H., Tang C. Prevalence and molecular characteristics of bovine respiratory syncytial virus in beef cattle in China. Animals (Basel). 2022; 12(24): 3511. https://doi.org/10.3390/ani12243511
- Karayel Hacioğlu İ., Coşkun N., Duran Yelken S., Sevinç S., Alkan F. Phylogenetic analysis of bovine respiratory syncytial viruses from calves with respiratory disorders. Kafkas Univ. Vet. Fak. Derg. 2019; 25(2): 251–6. https://doi.org/10.9775/kvfd.2018.20819
- Ellis J., Marx J., Perumbakkam S., West K., Gow S., Lacoste S., et al. Genealogy of an in-vivo passaged isolate of western Canadian bovine respiratory syncytial virus. Can. J. Vet. Res. 2022; 86(3): 218–28.
- Nettleton P.F., Gilray J.A., Caldow G., Gidlow J.R., Durkovic B., Vilcek S. Recent isolates of bovine respiratory syncytial virus from Britain are more closely related to isolates from the USA than to earlier British and current mainland European isolates. J. Vet. Med. B Infect. Dis. Vet. Public Health. 2003; 50(4): 196–9. https://doi.org/10.1046/j.1439-0450.2003.00647.x
- Krešić N., Bedeković T., Brnić D., Šimić I., Lojkić I., Turk N. Genetic analysis of bovine respiratory syncytial virus in Croatia. Comp. Immunol. Microbiol. Infect. Dis. 2018; 58: 52–7. https://doi.org/10.1016/j.cimid.2018.09.004
- Almeida R.S., Domingues H.G., Spilki F.R., Larsen L.E., Hägglund S., Belák S., et al. Circulation of bovine respiratory syncytial virus in Brazil. Vet. Rec. 2006; 158(18): 632–4. https://doi.org/10.1136/vr.158.18.632
- Kumagai A., Kawauchi K., Andoh K., Hatama S. Sequence and unique phylogeny of G genes of bovine respiratory syncytial viruses circulating in Japan. J. Vet. Diagn. Investig. 2021; 33(1): 162–6. https://doi.org/10.1177/1040638720975364
- Glotov A.G., Glotova T.I., Koteneva S.V., Nefedchenko A.V. Synthetic oligonucleotide primers and method of bovine respiratory syncytial infection RNA virus detection by synthetic oligonucleotide primers in Polymerase Chain Reaction (PCR). Patent RF 2405039 C1; 2010. https://elibrary.ru/ttzyly (in Russian)
- Prozzi D., Walravens K., Langedijk J.P., Daus F., Kramps J.A., Letesson J.J. Antigenic and molecular analyses of the variability of bovine respiratory syncytial virus G glycoprotein. J. Gen. Virol. 1997; 78(Pt. 2): 359–66. https://doi.org/10.1099/0022-1317-78-2-359
- Langedijk J.P., Meloen R.H., Taylor G., Furze J.M., van Oirschot J.T. Antigenic structure of the central conserved region of protein G of bovine respiratory syncytial virus. J. Virol. 1997; 71(5): 4055–61. https://doi.org/10.1128/jvi.71.5.4055-4061.1997
- Glotov A.G., Glotova T.I., Koteneva S.V., Nefedchenko A.V., Voytova K.V. Features of epidemiological situation on bovine respiratory syncytial virus infection (BRSV) in dairy farms. Veterinariya. 2010; (7): 21–5. https://elibrary.ru/msrezd (in Russian)
- Nefedchenko A.V., Glotov A.G., Koteneva S.V., Glotova T.I. Developing and testing a real-time polymerase chain reaction to identify and quantify bovine respiratory syncytial viruses. Molekulyarnaya genetika, mikrobiologiya i virusologiya. 2020; 38(3): 145–50. https://doi.org/10.17116/molgen202038031145 https://elibrary.ru/obaant (in Russian)
- Nefedchenko A.V., Glotov A.G., Koteneva S.V., Glotova T.I. Detection and quantitative assessment of viral and bacterial pathogens in bovine respiratory diseases by real-time-qPCR. Sel’skokhozyaystvennaya biologiya. 2021; 56(4): 695–706. https://doi.org/10.15389/agrobiology.2021.4.695rus https://elibrary.ru/spttqp (in Russian)
Supplementary files