Генетическое разнообразие вируса иммунодефицита человека (ВИЧ-1) в Калининградской области
- Авторы: Щемелев А.Н.1, Семенов А.В.2, Останкова Ю.В.1, Найденова Е.В.3, Зуева Е.Б.1, Валутите Д.Э.1, Чурина М.А.4, Виролайнен П.А.1, Тотолян А.А.1
-
Учреждения:
- ФБУН «Санкт-Петербургский НИИ эпидемиологии и микробиологии имени Пастера» Федеральной службы по надзору в сфере защиты прав потребителей и благополучия человека (Роспотребнадзор)
- Екатеринбургский научно-исследовательский институт вирусных инфекций Федерального бюджетного учреждения науки «Государственный научный центр вирусологии и биотехнологии «Вектор» Федеральной службы по надзору в сфере защиты прав потребителей и благополучия человека
- ФКУН «Российский научно-исследовательский противочумный институт «Микроб» Федеральной службы по надзору в сфере защиты прав потребителей и благополучия человека (Роспотребнадзор)
- СПб ГБУЗ «Клиническая инфекционная больница имени С.П. Боткина»
- Выпуск: Том 67, № 4 (2022)
- Страницы: 310-321
- Раздел: ОРИГИНАЛЬНЫЕ ИССЛЕДОВАНИЯ
- URL: https://virusjour.crie.ru/jour/article/view/633
- DOI: https://doi.org/10.36233/0507-4088-119
- ID: 633
Цитировать
Аннотация
Введение. Как известно на сегодняшний день, эпидемия ВИЧ-инфекции в Калининградской области преимущественно была связана с распространением рекомбинантной формы вируса (CRF03_AB), однако регулярные заносы ВИЧ из других стран и частей света создали благоприятные условия для формирования и распространения его разнообразных рекомбинантных форм.
Наиболее полная информация о разнообразии рекомбинантных форм в регионе необходима для понимания структуры лекарственной устойчивости (ЛУ), так как влияние ассоциированных с ней мутаций на приспособленность вируса может быть неодинаковым для разных субтипов, причём рекомбинантные формы могут сочетать в своём геноме наиболее удачные паттерны мутаций, что позволит ВИЧ с большей эффективностью противостоять антиретровирусной терапии.
Цель работы. Изучение генетического разнообразия ВИЧ-1 в Калининградской области.
Материалы и методы. Исследованы 162 образца плазмы крови, полученные от пациентов из Калининградской области как с подтверждённой вирусологической неэффективностью антиретровирусной терапии, так и с впервые выявленной ВИЧ-инфекцией. Для обратной транскрипции и амплификации ВИЧ использовали диагностический набор «АмплиСенс HIVResist-Seq» (ЦНИИЭ, Россия).
Результаты и обсуждение. Доминирующими в группе являлись различные рекомбинанты между субтипами А и В (74%), в том числе CRF03_AB и субтипом А (33,95%) и рекомбинантная форма, схожая с СRF03_AB (CRF03_AB-like (13,58%). Среди «чистых» субтипов вируса доминирует характерный для территории Российской Федерации суб-субтип – А6 (16,67%), одновременно с ним циркулируют субтипы В (3,70%) и G (1,23%).
Были выявлены 96 пациентов (59,26%) хотя бы c одной мутацией, ассоциированной с ЛУ к антиретровирусным препаратам.
Заключение. Выявленное разнообразие субтипов и рекомбинантных форм вируса указывает на то, что в исследуемом регионе продолжается активный процесс формирования новых рекомбинантов, причём между как уже существующими рекомбинантными формами и «чистыми» субтипами, так и между «чистыми» субтипами.
Полный текст
Введение
Вирус иммунодефицита человека 1-го типа (ВИЧ-1), впервые выявленный в 1980-х гг., циркулирует в человеческой популяции около 100 лет. За это время сформировалась устойчивая филогенетическая структура, представленная тремя большими группами: N, O, M; последняя, в свою очередь, разделяется на 12 независимых ветвей-субтипов, включающих вирусные штаммы, более тесно связанные друг с другом, чем с другими субтипами [1, 2]. Первоначально классификация ВИЧ-1 была основана на последовательностях субгеномных областей или отдельных генов, однако совершенствование методов секвенирования дало возможность классифицировать ВИЧ-1 на основе полноразмерных геномов или последовательностей из нескольких субгеномных областей. Это позволило идентифицировать штаммы с характерными частями их геномов, соответствующих разным субтипам: подобные штаммы являются продуктами рекомбинации между родительскими штаммами, принадлежащими к разным субтипам. Когда ту или иную рекомбинантную форму идентифицируют у трёх или более человек без прямой эпидемиологической связи, её классифицируют как циркулирующую рекомбинантную форму (CRF). В настоящее время известно более 100 CRF [3], в совокупности их вклад в глобальную эпидемию ВИЧ составляет не менее 20% [4], в том числе потому, что рекомбинантные формы преобладают в нескольких регионах, таких как Западная и Центральная Африка (CRF02_AG) [5, 6] и Юго-Восточная Азия (CRF01_AE) [7, 8].
Рекомбинантные формы вируса являются результатом рекомбинации в ходе обратной транскрипции. В процессе синтеза антисмысловой цепи дезоксирибонуклеиновой кислоты (ДНК) обратная транскриптаза (ОТ) с высокой частотой смещается с одной цепи рибонуклеиновой кислоты (РНК) на другую, и можно предположить, что, по крайней мере, обе копии геномной РНК альтернативно используются в качестве матриц. Частоту смены выбора копии у ВИЧ-1 между очень похожими матричными РНК оценивают в 3 × 10–4 – 1,4 × 10–3 событий на нуклеотид, т.е. 3–12 переключений матрицы на репликацию генома [9–11]. Важно отметить, что образование вирионов, содержащих две разные геномные РНК, требует выполнения определённых условий: во-первых, два или более вируса с разными генотипами должны инфицировать одну и ту же клетку, во-вторых, геномные РНК разного происхождения должны быть впоследствии совместно упакованы. Подобная ситуация может быть связана либо с коинфекцией, либо с суперинфекцией пациента различными субтипами вируса.
Тот факт, что белки Nef и Vpu подавляют экспрессию CD4 и корецепторов во время инфекции ВИЧ-1, даёт основание предполагать крайне редкое возникновение суперинфекции [12]. Тем не менее гибридизация клеток пациентов in situ показала, что отдельные клетки могут содержать более четырех различных провирусов [13, 14]. Кроме того, темпы образования новых рекомбинантных форм и их распространение, показанные в настоящее время, свидетельствуют о высокой частоте коинфекции in vivo [4, 15]. Таким образом, для активного образования рекомбинантных форм вируса необходимо выполнение важного условия – совместной циркуляции разных субтипов ВИЧ в регионе.
В Российской Федерации доминирующим суб-субтипом вируса является А6, также называемый IDU-A (Injecting Drug Users), или A-FSU (former Soviet Union countries). Данный суб-субтип ранее классифицировали как А1, но в связи со значимыми отличиями от других вариантов ВИЧ-1 суб-субтипа А1 в строении и распространении его выделили в отдельную сравнительно однородную группу [16, 17]. Тем не менее в некоторых регионах показана благоприятная обстановка для совместной циркуляции нескольких субтипов. В число таких регионов входит Калининградская область, так как её центр является крупным транспортным узлом с железными и шоссейными дорогами, морским и речным портами, международным аэропортом.
Как известно на сегодняшний день, эпидемический процесс в Калининградской области на начальном этапе был связан с распространением рекомбинантной формы вируса (CRF03_AB) в среде потребителей инъекционных наркотиков. В дальнейшем ВИЧ-инфекция вышла за пределы уязвимых групп населения. Кроме того, регулярные заносы ВИЧ из других стран и частей света создали благоприятных условия для формирования новых разнообразных рекомбинантных форм вируса в регионе [18, 19].
Примечательно, что распространение субтипов и рекомбинантных форм в эпидемии ВИЧ-инфекции очень динамично: сейчас генетическое разнообразие вируса представлено смесью рекомбинантов, возникших на ранних этапах глобальной эпидемии, и других, более позднего происхождения, и все они способствуют созданию более сложных рекомбинантных форм, которые в дальнейшем внесут свой вклад в динамику глобальной популяции ВИЧ-1. Можно предположить, что подобный процесс образования всё более сложных рекомбинантных форм окажется основным направлением эволюционного развития вируса в Калининградской области.
Целью работы являлось изучение генетического разнообразия ВИЧ-1 в Калининградской области.
Материалы и методы
В ходе работы в 2014–2018 гг. был исследован клинический материал от 162 пациентов из Калининградской области как с подтверждённой вирусологической неэффективностью антиретровирусной терапии (АРТ), так и с впервые выявленной ВИЧ-инфекцией. Плазма крови для определения устойчивых штаммов ВИЧ была направлена в Северо-Западный окружной центр по профилактике и борьбе со СПИДом (СЗО Центр СПИД) на базе Санкт-Петербургского НИИ эпидемиологии и микробиологии имени Пастера.
В полученной плазме крови была определена вирусная нагрузка набором реагентов «АмплиСенс ВИЧ-Монитор-FRT» (ЦНИИЭ, Россия) с порогом чувствительности 500 копий/мл. Образцы с определяемой вирусной нагрузкой в дальнейшем подвергали полимеразной цепной реакции с обратной транскрипцией РНК в ДНК (ОТ-ПЦР) и секвенированию по Сэнгеру. Для обратной транскрипции и амплификации ВИЧ использовали диагностические наборы «ОТ-ПЦР-комплект-Pro/Rev» и «ПЦР-комплект-Pro/Rev» (ЦНИИЭ, Россия), секвенирующую реакцию проводили согласно инструкции к набору «АмплиСенс HIVResist-Seq» (ЦНИИЭ, Россия). Генотипирование ВИЧ-1 проводили на основе анализа нуклеотидных последовательностей участка гена pol протяженностью 1302 нт., кодирующего протеазу (PR) и часть обратной транскриптазы (RT/ОТ) в области 2253–3554 нт., координаты даны для представленного в международной базе данных GenBank ВИЧ HXB2 (K03455.1). Анализ продуктов секвенирующей реакции проводили с использованием генетического анализатора ABI Prism 3500 (Applied Biosystems, США).
Первичный анализ нуклеотидных последовательностей проводили с помощью программы NCBI Blast в сравнении с нуклеотидными последовательностями, представленными в международной базе данных GenBank. Выравнивание нуклеотидных последовательностей проводили в программе MEGA 7.0, используя алгоритм ClustalW [20]. Для построения филогенетических деревьев и последующего филогенетического анализа применяли алгоритм Neighbor Joining, позволяющий оптимизировать деревья в соответствии с критерием сбалансированной минимальной эволюции. При оценке достоверности филогенетических связей использовали многократную генерацию выборок методом Bootstrap для 1000 независимых построений каждого филогенетического дерева.
Генотипирование исследуемых штаммов проводили параллельно в программе REGA HIV-1 Subtyping Tool 3.0 [21] и на основании анализа их филогенетических отношений с референсными последовательностями из международной базы данных GenBank. Для выявления и анализа рекомбинантных форм применяли программу REGA HIV-1 Subtyping Tool 3.0, используя параметры, предустановленные в программе (размер окна 400, шаг 20). Анализ генетических последовательностей ВИЧ-1 на наличие мутаций лекарственной устойчивости (ЛУ) проводили при помощи Стэнфордской базы данных (Stanford HIV DB) [22]. Анализ мутационных профилей проводили путём построения линейных диаграмм при помощи ПО Linear Diagram Generator [23].
Статистическую обработку данных производили с помощью пакетов программ MS Excel Professional Plus 2013 (Microsoft), Prizm v5.0 (GraphPad Software Inc.). При оценке статистической погрешности использовали точный интервал Клоппера–Пирсона. Результаты представляли с указанием 95% доверительного интервала (ДИ). Для оценки достоверности различий численных данных, полученных при парных сравнениях, использовали (в зависимости от характеристик выборок) точный критерий Фишера или критерий χ2 с поправкой Йейтса. В качестве порога достоверности отличий значение вероятности определили как p < 0,05.
Исследование проводилось при информированном согласии пациентов. Протокол исследования одобрен Этическим комитетом Протоколы исследования № 3 от 07.04.2010 и № 47 от 25.12.2018 одобрены Этическим комитетом ФБУН «Санкт-Петербургский научно-исследовательский институт эпидемиологии и микробиологии им. Пастера».
Результаты и обсуждение
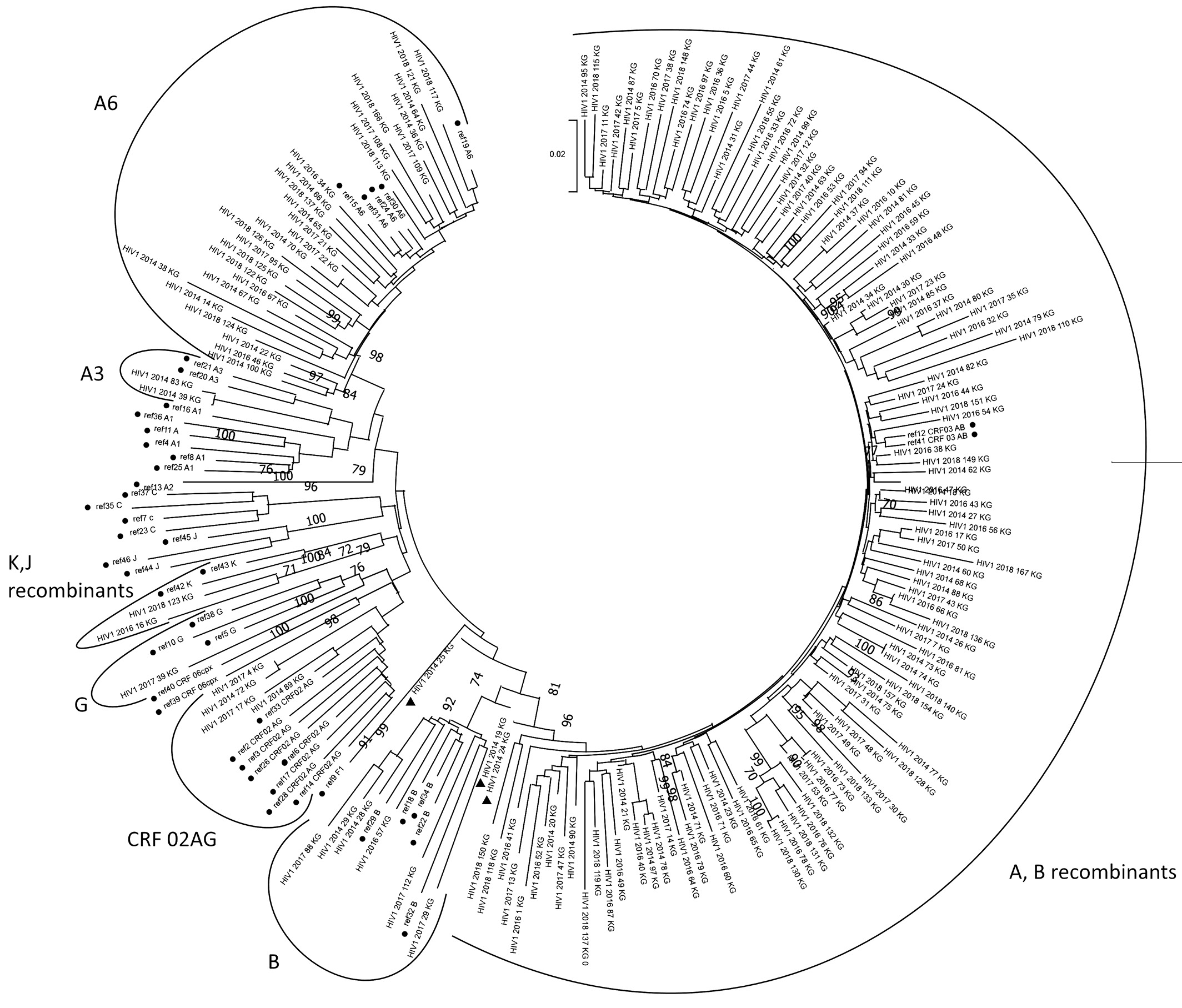
Получены последовательности 162 штаммов ВИЧ-1, которые были депонированы в GenBank под номерами ON367567 – ON367728. Для всех был определен их суб-субтип (табл. 1). При этом учитывали как данные, полученные при генотипировании при помощи инструментов REGA HIV Subtyping Tool 3.0 и jumping profile Hidden Markov Model (jpHMM) (Приложение А), так и результаты филогенетического исследования, проведенного в ПО Mega X (рис. 1).
Таблица 1. Распределение исследованных штаммов по субтипам ВИЧ-1
Table 1. Distribution of isolates by HIV-1 subtypes
Субтип Subtype | Количество штаммов Number of isolates | Доля в выборке, % Sample Share, % | 95% ДИ, % 95% CI, % | |
– | + | |||
HIV-1 Subtype A3 | 2 | 1,23 | 0,15 | 4,39 |
HIV-1 Subtype A6 | 27 | 16,67 | 11,28 | 23,31 |
HIV-1 Subtype B | 6 | 3,70 | 1,37 | 7,89 |
HIV-1 Subtype G | 2 | 1,23 | 0,15 | 4,39 |
HIV-1 CRF03_AB | 41 | 25,31 | 18,81 | 32,73 |
HIV-1 CRF03_AB-like | 22 | 13,58 | 8,71 | 19,84 |
Recombinant of 03_AB, A | 55 | 33,95 | 26,71 | 41,79 |
HIV-1 CRF02_AG | 4 | 2,47 | 0,68 | 6,20 |
Recombinant of A1, B | 2 | 1,23 | 0,15 | 4,39 |
Recombinant of K, J | 2 | 1,23 | 0,15 | 4,39 |
Рис. 1. Результаты филогенетического анализа при помощи алгоритма Neighbor Joining.
• – референсные последовательности (табл. 2) ;
▲ – рекомбинантный формы между субтипами А и В, не кластеризующиеся с другими рекомбинантами этой группы.
Таблица 2. Наименование референсных последовательностей из GenBank, использованных в филогенетическом анализе
Table 2. Names of reference sequences from GenBank used in phylogenetic analysis
Суб- субтип Sub- subtype | Номер последовательности на филогенетическом дереве Number sequence in phylogenetic tree | Номер последовательности из GenBank Number sequence in GenBank | Регион происхождения Region of origin |
A1 | ref4 | AF069670 | Сомали Somali |
A1 | ref8 | AB287376 | Руанда Ruanda |
A1 | ref16 | U51190 | Уганда Uganda |
A1 | ref25 | EU110087 | Кения Kenia |
A1 | ref27 | AF484509 | Уганда Uganda |
A1 | ref36 | AF107771 | Швеция Sweden |
A2 | ref13 | AF286237 | Кипр Cyprus |
A3 | ref1 | AY521631 | Сенегал Senegal |
A3 | ref20 | AY521629 | Швеция Sweden |
A6 | ref15 | HQ449397 | Россия, Краснодар Russia, Krasnodar |
A6 | ref19 | HQ161930 | Россия, Смоленск Russia, Smolensk |
A6 | ref24 | EF589043 | Казахстан Kazakhstan |
A6 | ref30 | AY500393 | Россия, Москва Russia, Moscow |
A6 | ref31 | AF413987 | Украина Ukraine |
B | ref18 | M17449 | США USA |
B | ref22 | KJ771697 | Германия Germany |
B | ref29 | HM586190 | Великобритания Great Britain |
B | ref32 | AY713409 | США USA |
B | ref34 | AY173951 | Таиланд Thailand |
C | ref7 | AF067155 | Индия India |
C | ref23 | U52953 | Бразилия Brazil |
C | ref35 | U46016 | Эфиопия Ethiopia |
C | ref37 | AY772699 | Африка Africa |
F1 | ref9 | AF075703 | Финляндия Finland |
G | ref5 | AF061641 | Финляндия Finland |
G | ref10 | U88826 | Нигерия Nigeria |
G | ref38 | AF084936 | Конго Congo |
J | ref44 | EF614151 | Конго Congo |
J | ref45 | GU237072 | Камерун Cameron |
J | ref46 | AF082394 | Швеция Sweden |
К | ref42 | AJ249235 | Камерун Cameron |
К | ref43 | AJ249239 | Камерун Cameron |
CRF02_AG | ref2 | AF063224 | Джибути Djibouti |
CRF02_AG | ref3 | GU201514 | Камерун Cameron |
CRF02_AG | ref6 | KT124792 | Германия Germany |
CRF02_AG | ref14 | AB231898 | Гана Ghana |
CRF02_AG | ref17 | EU786671 | Испания Spain |
CRF02_AG | ref26 | AB231896 | Гана Ghana |
CRF02_AG | ref28 | AY151001 | Эквадор Ecuador |
CRF02_AG | ref33 | AF377954 | Камерун Cameron |
CRF06_cpx | ref39 | HQ529257.1 | Гана Ghana |
CRF03_AB | ref12 | AF193276 | Россия, Калининград Russia, Kalinigrad |
CRF03_AB | ref41 | AF414006.1 | Беларусь Belarus |
CRF06_cpx | ref40 | MH605500.1 | Гвинея-Бисау Guinea-Bissau |
Доминирующими в группе являлись различные рекомбинанты между субтипами А и В (74%; 95% ДИ 66,61–80,63%), наиболее часто встречали рекомбинант между CRF03_AB и субтипом А (33,95%; 95% ДИ 26,71–41,79%). Кроме того, значительную долю (13,58%; 95% ДИ 8,71–19,84%) составляла рекомбинантная форма, схожая с СRF03_AB (CRF03_AB-like), но вклад «чистых» субтипов в формирование данной рекомбинантной формы не до конца ясен.
Доминирование в регионе рекомбинантов между субтипами А и В, в том числе CRF03_AB, согласуется с описанным в литературе генетическим разнообразием вируса в Калининградской области [16, 18, 19]. Тем не менее обращает на себя внимание неоднородность внутри клады рекомбинантов на филогенетическом дереве, что может быть связано как с особенностями эпидемиологических связей, так и с формированием в регионе новых циркулирующих рекомбинантных форм. Данный вопрос требует изучения полных геномов штаммов вируса из Калининградской области.
Среди рекомбинантов между субтипами А и В особого внимания заслуживают три штамма, отмеченные на филогенетическом дереве (рис. 1). На дендрограмме рекомбинанты между субтипами A и B образуют крупный гетерогенный кластер, а данные штаммы отделены от них, несмотря на то что в составе их гена pol присутствуют фрагменты, соответствующие субтипам А и В, что было подтверждено сравнительным рекомбинационным анализом в различном ПО (рис. 2). При этом их субтипирование в различных инструментах (REGA, Geno2Pheno, NCBI, Stanford HIV DB, RIP) не позволяет сделать окончательный вывод о генотипической принадлежности данных штаммов. Необходимо отметить, что все три образца имеют различное расположение на дендрограмме: штамм HIV1_2014_24_KG наиболее приближен к другим рекомбинантным формам между субтипами А и В; штамм HIV1_2014_19_KG кластеризуется со штаммами субтипа В; а штамм HIV1_2014_25_KG образует наиболее раннее ответвление – на уровне расхождения А-, В-рекомбинантов и других субтипов ВИЧ-1. Пациенты, от которых были получены все три штамма, инфицированы относительно недавно (срок инфекции менее одного года) и не получали АРТ. При анализе генетических последовательностей на хроматограммах были выявлены многочисленные вырожденные фрагменты, т.е. присутствие нескольких различных нуклеотидов в одних и тех же позициях генома. Наблюдаемая ситуация, как известно, может свидетельствовать о разнообразии вирусной популяции в организме пациента, в том числе о коинфекции разными субтипами ВИЧ [24]. Отмечая вышесказанное, можно предположить, что обозначенные выше штаммы находятся в начале процесса рекомбинации ретровирусов, т.е. представлены преимущественно не рекомбинантными формами, а вариантами вируса с совместно упакованными в капсиде геномами А- и В-штаммов.
Рис. 2. Сравнительный рекомбинационный анализ образцов 2014_80 (CRF03_AB) и 2014_19 (A + B recombinant) в Rega HIV Subtyping Tool v3.0 [21] и Recombinant Identification Program [25]. a – образец 2014_80 в Rega HIV Subtyping Tool v3.0; б – образец 2014_80 в Recombinant Identification Program; в – образец 2014_19 в Rega HIV Subtyping Tool v3.0; г – образец 2014_19 в Recombinant Identification Program [25].
Кроме рекомбинантов между субтипами А и В, были встречены более редкие для европейской части России CRF02_AG, а также рекомбинанты между субтипами K и J [26].
Среди «чистых» субтипов вируса доминирует характерный для территории Российской Федерации субтип А, представленный двумя суб-субтипами – А6 (16,67%; 95% ДИ 11,28–23,31%) и А3 (1,23%; 95% ДИ 0,15–4,39%); одновременно с ним циркулируют субтипы В (3,70%; 95% ДИ 1,37–7,89%) и G (1,23%; 95% ДИ 0,15–4,39%).
Исследуемый регион демонстрирует распределение субтипов ВИЧ-1, отличное от других регионов России в целом и Северо-Западного федерального округа в частности [27–29]. Для сравнения значимости различий генетического разнообразия между регионами Северо-Западного федерального округа были выбраны суб-субтип А6, субтип В и рекомбинантные формы между субтипами А и В, поскольку именно они встречаются не только среди изученных нами образцов, но также и в штаммах из Архангельской [28] и Ленинградской области [29]. Для оценки достоверности различий был использован критерий χ2 с поправкой Йейтса. При этом достоверных различий между встречаемостью субтипов ВИЧ-1 в Архангельской и Ленинградской областях не выявлено, но наблюдается статистически значимое различие генетического разнообразия между ними и Калининградской областью (χ2 составляет 254,277; критическое значение χ2 при уровне значимости p = 0,01 составляет 13,277).
Такие различия в генетическом разнообразии объясняются доминированием рекомбинантных форм ВИЧ-1 в Калининградской области, в то время как в Архангельской и Ленинградской областях они были встречены в единичных случаях. Одновременно с этим разнообразие «чистых» субтипов ВИЧ-1 соотносится с описанным в литературе [27–29], среди них также наблюдается преобладание субтипа А, преимущественно суб-субтипа А6.
Факт того, что в регионе преобладают варианты вируса, представляющие собой рекомбинант между CRF03_AB и субтипом А, а также рекомбинантная форма, схожая с CRF03_AB, но имеющая от неё ряд отличий (CRF03_AB-like), соотносится с представлением о том, что при длительной совместной циркуляции в популяции рекомбинантных форм и «чистых» субтипов вируса формируются новые, более сложные рекомбинантные формы с включением в геном новых фрагментов [4].
Помимо генотипического анализа, было проведено исследование встречаемости в данном регионе мутаций, ассоциированных с ЛУ. При этом были исследованы штаммы, полученные как от пациентов с неэффективностью АРТ (n = 107), так и с впервые выявленной инфекцией (n = 55). Первичная ЛУ была выявлена всего в двух случаях (3,64%; 95% ДИ 0,44–12,53%), поэтому дальнейший анализ объединяет всех пациентов с выявленными мутациями ЛУ.
Всего было встречено 80 различных мутаций, ассоциированных с ЛУ. Из них большая часть – мутации ЛУ к ингибиторам ОТ, в том числе к нуклеозидным ингибиторам обратной транскриптазы (НИОТ) – 31 замена (38,75%; 95% ДИ 28,06–50,30%) и к ненуклеозидным ингибиторам обратной транскриптазы (ННИОТ) – 35 мутаций (43,75%; 95% ДИ 32,68–55,30%); меньшая доля разнообразия мутаций приходится на замены, ассоциированные с ЛУ к ингибиторам протеазы (ИП) – 14 (17,50%; 95% ДИ 9,91–27,62%).
У 96 пациентов (59,26%; 95% ДИ 51,27–66,90%) были выявлены штаммы ВИЧ-1 хотя бы c одной мутацией, ассоциированной с ЛУ к антиретровирусным препаратам. Наиболее часто встречались мутации ЛУ к ингибиторам ОТ. В 13 случаях были встречены мутации ЛУ к НИОТ, в 4 – ННИОТ, в 66 – НИОТ + ННИОТ. Кроме того, у 13 пациентов были встречены мутации ЛУ к ИП: у 10 – ИП + НИОТ, у 3 – ИП + НИОТ + ННИОТ.
Среди мутаций ЛУ к НИОТ наиболее часто показаны мутации M184V [30] (65,63%; 95% ДИ 55,23–75,02%), L74V [31] (19,79%; 95% ДИ 12,36–29,17%), Y115F [32] (14,58%; 95% ДИ 8,21–23,26%), остальные замены встречались в 10% случаев и реже. При анализе множества мутационных профилей путём построения линейных диаграмм можно проследить образующиеся устойчивые паттерны мутаций ЛУ (рис. 3 а). Подробно описанные в литературе сочетания мутаций, ассоциированных с ЛУ к тимидиновым аналогам (thymidine analog mutations – ТАМ), встречены в полученных профилях в единичных случаях. Существует два основных пути формирования паттернов ТАМ: мутации, возникающие вместе с T215Y (включая M41L, L210W и иногда D67N), составляют кластер TAM-1; мутации, возникающие вместе с K70R (включая D67N, T215F и K219Q), составляют кластер TAM-2. Тем не менее в данном случае оба кластера мутаций ассоциированы с заменой T215Y, в то время как известно, что формирование паттерна по пути TAM-2 обладает наибольшими преимуществами с заменой T215F, которая также показана в изученных мутационных профилях, но не в составе паттернов TAM [33]. Доминирующими являлись профили, несущие не-TAM мутации, среди них обнаружена устойчивая связь замен L74V + Y115F. Данные мутации связаны преимущественно с ЛУ к абакавиру и диданозину, но существуют сведения об их ассоциации с ЛУ к тенофовиру [31, 32], который, в свою очередь, входит в большинство современных схем антиретровирусной терапии. Кроме того, во всех случаях данное сочетание встречено вместе с заменой M184V, что, скорее всего, связано с присутствием данной замены у большинства штаммов, обладающих ЛУ.
Рис. 3. Результаты исследования множества мутационных профилей путём построения линейных диаграмм: а – для мутаций устойчивости к НИОТ; б – для мутаций устойчивости к ННИОТ.
Анализ встречаемости мутаций ЛУ к ННИОТ показал, что наиболее распространены замены K103N [34] (36,46%; 95% ДИ 26,87–46,91%), K101E [35] (12,50%; 95% ДИ 6,63–20,82%), G190A [36] (11,46%; 95% ДИ 5,86–19,58%), P225H (15,63%; 95% ДИ 9,02–24,46%), Y18C [37] (12,50%; 95% ДИ 6,63–20,82%); остальные мутации встречались менее чем в 10% случаев. Изучение профилей мутаций ЛУ в полученных штаммах (рис. 3 б) позволило обнаружить связь между заменами K101E + G190A/S, причём данное сочетание встречается преимущественно без наиболее распространённой мутации K103N. Также выявлена зависимость замены в 190-й позиции на аланин (А) или серин (S) от субтипа вируса. Замена 190А встречалась только в рекомбинантах между субтипами А и В, а мутация 190S – преимущественно в штаммах субсубтипа А6 (в пяти из шести случаев). В литературе также описана распространённость замены в 190-й позиции ОТ на серин для субтипа А [38–40] и аланин для не-А субтипов [38, 41, 42].
Заключение
Полученные в исследовании результаты указывают на значительное разнообразие рекомбинантных форм в Калининградской области. При этом преобладание рекомбинантов между CRF03_AB и А указывает на то, что основным источником рекомбинации является совместная циркуляция варианта, характерного для исследуемого региона – CRF03_AB, и суб-субтипа А6, распространённого на остальной территории России. Вклад совместной циркуляции с субтипом В не выявлен.
Выявленное разнообразие субтипов и рекомбинантных форм вируса указывает на то, что в исследуемом регионе продолжается активный процесс формирования новых рекомбинантов, причём между как уже существующими рекомбинантными формами и «чистыми» субтипами, так и чистыми» субтипами. Подобная активность вируса указывает на необходимость изучения полных геномов штаммов, полученных в Калининградской области, для описания всех рекомбинантных форм, циркулирующих там на сегодняшний день.
Об авторах
Александр Николаевич Щемелев
ФБУН «Санкт-Петербургский НИИ эпидемиологии и микробиологии имени Пастера» Федеральной службы по надзору в сфере защиты прав потребителей и благополучия человека (Роспотребнадзор)
Автор, ответственный за переписку.
Email: tvildorm@gmail.com
ORCID iD: 0000-0002-3139-3674
младший научный сотрудник лаборатории иммунологии и вирусологии ВИЧ-инфекции, ФБУН «Санкт-Петербургский научно-исследовательский институт эпидемиологии и микробиологии имени Пастера» Федеральной службы по надзору в сфере защиты прав потребителей и благополучия человека (Роспотребнадзор)
Россия, 197101, г. Санкт-ПетербургАлександр В. Семенов
Екатеринбургский научно-исследовательский институт вирусных инфекций Федерального бюджетного учреждения науки «Государственный научный центр вирусологии и биотехнологии «Вектор» Федеральной службы по надзору в сфере защиты прав потребителей и благополучия человека
Email: tvildorm@gmail.com
ORCID iD: 0000-0003-3223-8219
Россия, 620030, г. Екатеринбург
Юлия В. Останкова
ФБУН «Санкт-Петербургский НИИ эпидемиологии и микробиологии имени Пастера» Федеральной службы по надзору в сфере защиты прав потребителей и благополучия человека (Роспотребнадзор)
Email: tvildorm@gmail.com
ORCID iD: 0000-0003-2270-8897
Россия, 197101, г. Санкт-Петербург
Екатерина В. Найденова
ФКУН «Российский научно-исследовательский противочумный институт «Микроб» Федеральной службы по надзору в сфере защиты прав потребителей и благополучия человека (Роспотребнадзор)
Email: tvildorm@gmail.com
ORCID iD: 0000-0001-6474-3696
Россия, 410005, г. Саратов
Елена Б. Зуева
ФБУН «Санкт-Петербургский НИИ эпидемиологии и микробиологии имени Пастера» Федеральной службы по надзору в сфере защиты прав потребителей и благополучия человека (Роспотребнадзор)
Email: tvildorm@gmail.com
ORCID iD: 0000-0002-0579-110X
Россия, 197101, г. Санкт-Петербург
Диана Э. Валутите
ФБУН «Санкт-Петербургский НИИ эпидемиологии и микробиологии имени Пастера» Федеральной службы по надзору в сфере защиты прав потребителей и благополучия человека (Роспотребнадзор)
Email: tvildorm@gmail.com
ORCID iD: 0000-0002-0931-102X
Россия, 197101, г. Санкт-Петербург
Мария А. Чурина
СПб ГБУЗ «Клиническая инфекционная больница имени С.П. Боткина»
Email: tvildorm@gmail.com
ORCID iD: 0000-0003-0424-4654
Россия, 191167, г. Санкт-Петербург
Павел А. Виролайнен
ФБУН «Санкт-Петербургский НИИ эпидемиологии и микробиологии имени Пастера» Федеральной службы по надзору в сфере защиты прав потребителей и благополучия человека (Роспотребнадзор)
Email: tvildorm@gmail.com
ORCID iD: 0000-0001-5918-9395
Россия, 197101, г. Санкт-Петербург
Арег А. Тотолян
ФБУН «Санкт-Петербургский НИИ эпидемиологии и микробиологии имени Пастера» Федеральной службы по надзору в сфере защиты прав потребителей и благополучия человека (Роспотребнадзор)
Email: tvildorm@gmail.com
ORCID iD: 0000-0003-4571-8799
Россия, 197101, г. Санкт-Петербург
Список литературы
- Korber B., Muldoon M., Theiler J., Gao F., Gupta R., Lapedes A., et al. Timing the ancestor of the HIV-1 pandemic strains. Science. 2000; 288(5472): 1789–96. https://doi.org/10.1126/science.288.5472.1789
- Kuiken C., Foley B., Hahn B., Marx P., McCutchan F., Mellors J.W., et al. A compilation and analysis of nucleic acid and amino acid sequences. In: Human Retroviruses and AIDS. Los Alamos; 1999.
- Los Alamos National Laboratory. HIV Circulating Recombinant Forms (CRFs). Available at: https://www.hiv.lanl.gov/content/sequence/HIV/CRFs/CRFs.html
- Simon-Loriere E., Rossolillo P., Negroni M. RNA structures, genomic organization and selection of recombinant HIV. RNA Biol. 2011; 8(2): 280–6. https://doi.org/10.4161/rna.8.2.15193
- McCutchan F.E., Carr J.K., Bajani M., Sanders-Buell E., Harry T.O., Stoeckli T.C., et al. Subtype G and multiple forms of A/G intersubtype recombinant human immunodeficiency virus type 1 in Nigeria. Virology. 1999; 254(2): 226–34. https://doi.org/10.1006/viro.1998.9505
- Montavon C., Toure-Kane C., Liegeois F., Mpoudi E., Bourgeois A., Vergne L., et al. Most env and gag subtype A HIV-1 viruses circulating in West and West Central Africa are similar to the prototype AG recombinant virus IBNG. J. Acquir. Immune. Defic. Syndr. 2000; 23(5): 363–74. https://doi.org/10.1097/00126334-200004150-00001
- Menu E., Truong T.X., Lafon M.E., Nguyen T.H., Müller-Trutwin M.C., Nguyen T.T., et al. HIV type 1 Thai subtype E is predominant in South Vietnam. AIDS Res. Hum. Retroviruses. 1996; 12(7): 629–33. https://doi.org/10.1089/aid.1996.12.629
- Piyasirisilp S., McCutchan F.E., Carr J.K., Sanders-Buell E., Liu W., Chen J., et al. A recent outbreak of human immunodeficiency virus type 1 infection in southern China was initiated by two highly homogeneous, geographically separated strains, circulating recombinant form AE and a novel BC recombinant. J. Virol. 2000; 74(23): 11286–95. https://doi.org/10.1128/jvi.74.23.11286-11295.2000
- Galetto R., Moumen A., Giacomoni V., Veron M., Charneau P., Negroni M. The structure of HIV-1 genomic RNA in the gp120 gene determines a recombination hot spot in vivo. J. Biol. Chem. 2004; 279(35): 36625–32. https://doi.org/10.1074/jbc.m405476200
- Zhuang J., Jetzt A.E., Sun G., Yu H., Klarmann G., Ron Y., et al. Human immunodeficiency virus type 1 recom-bination: rate, fidelity and putative hot spots. J. Virol. 2002; 76(22): 11273–82. https://doi.org/10.1128/jvi.76.22.11273-11282.2002
- Jetzt A.E., Yu H., Klarmann G.J., Ron Y., Preston B.D., Dougherty J.P. High rate of recombination throughout the human immunodeficiency virus type 1 genome. J. Virol. 2000; 74(3): 1234–40. https://doi.org/10.1128/jvi.74.3.1234-1240.2000
- Piantadosi A., Chohan B., Chohan V., McClelland R.S., Overbaugh J. Chronic HIV-1 infection frequently fails to protect against superinfection. PLoS Pathog 2007; 3(11): 177. https://doi.org/10.1371/journal.ppat.0030177
- Gratton S., Cheynier R., Dumaurier M.J., Oksenhendler E., Wain-Hobson S. Highly restricted spread of HIV-1 and multiply infected cells within splenic germinal centers. Proc. Natl. Acad. Sci. USA. 2000; 97(26): 14566–71. https://doi.org/10.1073/pnas.97.26.14566
- Jung A., Maier R., Vartanian J.P., Bocharov G., Jung V., Fischer U., et al. Recombination: Multiply infected spleen cells in HIV patients. Nature. 2002; 418(6894): 144. https://doi.org/10.1038/418144a
- Chen J., Dang Q., Unutmaz D., Pathak V.K., Maldarelli F., Powell D., et al. Mechanisms of nonrandom human immunodeficiency virus type 1 infection and double infection: preference in virus entry is important but is not the sole factor. J. Virol 2005; 79(7): 4140–9. https://doi.org/10.1128/jvi.79.7.4140-4149.2005
- Bobkov A.F., Kazennova E.V., Selimova L.M., Khanina T.A., Ryabov G.S., Bobkova M.R., et al. Temporal trends in the HIV-1 epidemic in Russia: predominance of subtype A. J. Med. Virol. 2004; 74(2): 191–6. https://doi.org/10.1002/jmv.20177
- Schlösser M., Kartashev V.V., Mikkola V.H., Shemshura A., Saukhat S., Kolpakov D., et al. HIV-1 sub-subtype A6: Settings for normalised identification and molecular epidemiology in the Southern Federal District, Russia. Viruses. 2020; 12(4): 475. https://doi.org/10.3390/v12040475
- Liitsola K., Tashkinova I., Laukkanen T., Korovina G., Smolskaja T., Momot O., et al. HIV-1 genetic subtype A/B recombinant strain causing an explosive epidemic in injecting drug users in Kaliningrad. AIDS. 1998; 12(14): 1907–19. https://doi.org/10.1097/00002030-199814000-00023
- Lebedev A., Pasechnik O., Ozhmegova E., Antonova A., Blokh A., Grezina L., et al. Prevalence and spatiotemporal dynamics of HIV-1 Circulating Recombinant Form 03_AB (CRF03_AB) in the Former Soviet Union countries. PLoS One. 2020; 15(10): e0241269. https://doi.org/10.1371/journal.pone.0241269
- Kumar S., Stecher G., Tamura K. MEGA7: Molecular Evolutionary Genetics Analysis version 7.0 for bigger datasets. Mol. Biol. Evol. 2016; 33(7): 1870–4. https://doi.org/10.1093/molbev/msw054
- Stanford University. HIV Drug Resistance Database. REGA HIV-1 Subtyping Tool – Version 3.0. Available at: http://dbpartners.stanford.edu:8080/RegaSubtyping/stanford-hiv/typingtool/
- Stanford University. HIV Drug Resistance Database. HIVdb Program: Mutations Analysis. Available at: https://hivdb.stanford.edu/hivdb/by-patterns/
- Gottfried B. A comparative study on linear and region based diagrams. J. Spat. Inf. Sci. 2015; (10): 3–20.
- Лаповок И.А., Салеева Д.В., Кириченко А.А., Мурзакова А.В., Лопатухин А.Э., Киреев Д.Е. Исследование частоты встречаемости двойной ВИЧ-инфекции в России. Инфекционные болезни. 2020; 18(4): 138–48. https://doi.org/10.20953/1729-9225-2020-4-138-148
- Los Alamos National Laboratory. RIP: Recombinant Identification Program. Available at: https://www.hiv.lanl.gov/content/sequence/RIP/RIP.html
- Пасечник О.А., Блох А.И. Распространенность рекомбинантных форм ВИЧ-1 в регионах Российской Федерации и стран СНГ: систематический обзор и метаанализ. Инфекция и иммунитет. 2018; 8(2): 127–38. https://doi.org/10.15789/2220-7619-2018-2-127-138
- Федеральный центр по борьбе со СПИД. Российская база данных. ЛУ ВИЧ у наивных пациентов; 2020. Available at: http://www.hivrussia.info/wp-content/uploads/2020/12/2020-Rossijskaya-baza-dannyh-LU-VICH-u-naivnyh-patsientov.pdf
- Останкова Ю.В., Щемелев А.Н., Зуева Е.Б., Чурина М.А., Валутите Д.Э., Семенов А.В. Молекулярная эпидемиология и фармакорезистентность ВИЧ у пациентов с вирусологической неэффективностью антиретровирусной терапии в Архангельской области. ВИЧ инфекция и иммуносупрессии. 2019; 11(4): 65–72. https://doi.org/10.22328/2077-9828-2019-11-4-79-90
- Щемелев А.Н., Семенов А.В., Останкова Ю.В., Зуева Е.Б., Валутите Д.Э., Семенова Д.А. и др. Генетическое разнообразие и мутации лекарственной устойчивости ВИЧ-1 в Ленинградской области. Журнал микробиологии, эпидемиологии и иммунобиологии. 2022; 99(1): 28–37. https://doi.org/10.36233/0372-9311-216
- Hung M., Tokarsky E.J., Lagpacan L., Zhang L., Suo Z., Lansdon E.B. Elucidating molecular interactions of L-nucleotides with HIV-1 reverse transcriptase and mechanism of M184V-caused drug resistance. Commun. Biol. 2019; 2: 469. https://doi.org/10.1038/s42003-019-0706-x
- De Luca A., Giambenedetto S.D., Trotta M.P., Colafigli M., Prosperi M., Ruiz L., et al. Improved interpretation of genotypic changes in the HIV-1 reverse transcriptase coding region that determine the virological response to didanosine. J. Infect. Dis. 2007; 196(11): 1645–53. https://doi.org/10.1086/522231
- Lanier E.R., Givens N., Stone C., Griffin P., Gibb D., Walker S., et al. Effect of concurrent zidovudine use on the resistance pathway selected by abacavir-containing regimens. HIV Med. 2004; 5(6): 394–9. https://doi.org/10.1111/j.1468-1293.2004.00243.x
- Hu Z., Giguel F., Hatano H., Reid P., Lu J., Kuritzkes D.R. Fitness comparison of thymidine analog resistance pathways in human immunodeficiency virus type 1. J. Virol. 2006; 80(14): 7020–7. https://doi.org/10.1128/jvi.02747-05
- Ibe S., Sugiura W. Clinical significance of HIV reverse-transcriptase inhibitor-resistance mutations. Future Microbiol. 2011; 6(3): 295–315. https://doi.org/10.2217/fmb.11.7
- Xu H.T., Colby-Germinario S.P., Huang W., Oliveira M., Han Y., Quan Y., et al. Role of the K101E substitution in HIV-1 reverse transcriptase in resistance to rilpivirine and other nonnucleoside reverse transcriptase inhibitors. Antimicrob. Agents Chemother. 2013; 57(11): 5649–57. https://doi.org/10.1128/aac.01536-13
- Madruga J.V., Cahn P., Grinsztejn B., Haubrich R., Lalezari J., Mills A., et al. Efficacy and safety of TMC125 (etravirine) in treatment-experienced HIV-1-infected patients in DUET-1: 24-week results from a randomised, double-blind, placebo-controlled trial. Lancet. 2007; 370(9581): 29–38. https://doi.org/10.1016/s0140-6736(07)61047-2
- Archer R.H., Wisniewski M., Bambara R.A., Demeter L.M. The Y181C mutant of HIV-1 reverse transcriptase resistant to nonnucleoside reverse transcriptase inhibitors alters the size distribution of RNase H cleavages. Biochemistry. 2001; 40(13): 4087–95. https://doi.org/10.1021/bi002328a
- Kolomeets A.N., Varghese V., Lemey P., Bobkova M.R., Shafer R.W. A uniquely prevalent nonnucleoside reverse transcriptase inhibitor resistance mutation in Russian subtype A HIV-1 viruses. AIDS. 2014; 28(17): F1–8. https://doi.org/10.1097/qad.0000000000000485
- Останкова Ю.В., Щемелев А.Н., Зуева Е.Б., Чурина М.А., Валутите Д.Э., Семенов А.В. Молекулярная эпидемиология и фармакорезистентность ВИЧ у пациентов с вирусологической неэффективностью антиретровирусной терапии в Архангельской области. ВИЧ инфекция и иммуносупрессии. 2019; 11(4): 65–72. https://doi.org/10.22328/2077-9828-2019-11-4-79-9
- Чурина М.А., Останкова Ю.В., Семенов А.В., Никитина Н.А., Росоловский А.П., Гребенкина Е.В. и др. Молекулярная эпидемиология и фармакорезистентность ВИЧ-1 у пациентов с неэффективностью АРВТ в Великом Новгороде. ВИЧ-инфекция и иммуносупрессии. 2017; 9(1): 82–92. https://doi.org/10.22328/2077-9828-2017-9-1-82-92
- Chen M., Zhu Q., Xing H., Chen H., Jin X., Dong L., et al. The characteristics of pretreatment HIV-1 drug resistance in western Yunnan, China. Epidemiol. Infect. 2020; 148: e102. https://doi.org/10.1017/s095026882000093x
- Cheung K.W., Peng Q., He L., Cai K., Jiang Q., Zhou B., et al. Rapid and simultaneous detection of major drug resistance mutations in reverse transcriptase gene for HIV-1 CRF01_AE, CRF07_BC and subtype B in China using sequenom MassARRAY® system. PLoS One. 2016; 11(4): e0153641. https://doi.org/10.1371/journal.pone.0153641
Дополнительные файлы