Детекция Liman tick virus (неклассифицированный представитель Chuviridae) в культуре клеток клещей HAE/CTVM8
- Авторы: Литов А.Г.1,2, Щетинин А.М.3, Холодилов И.С.1, Белова О.А.1, Калянова А.С.1, Гущин В.А.3,4, Карганова Г.Г.1,2
-
Учреждения:
- ФГАНУ «Федеральный научный центр исследований и разработки иммунобиологических препаратов имени М.П. Чумакова РАН» (Институт полиомиелита)
- Институт трансляционной медицины и биотехнологии ФГАОУ ВО «Первый Московский государственный медицинский университет имени И.М. Сеченова» Минздрава России (Сеченовский Университет)
- ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
- ФГАОУ ВО «Первый Московский государственный медицинский университет имени И.М. Сеченова» Минздрава России (Сеченовский Университет)
- Выпуск: Том 70, № 2 (2025)
- Страницы: 147-153
- Раздел: ОРИГИНАЛЬНЫЕ ИССЛЕДОВАНИЯ
- URL: https://virusjour.crie.ru/jour/article/view/16705
- DOI: https://doi.org/10.36233/0507-4088-283
- EDN: https://elibrary.ru/hrnscv
- ID: 16705
Цитировать
Аннотация
Введение. Культуры клеток клещей широко используются для изучения биологии этих членистоногих и переносимых ими патогенов, в особенности вирусов. Большинство имеющихся в настоящее время культур клеток были получены из эмбриональных клеток клещей и могут быть инфицированы вирусами. Клеточная линия HAE/CTVM8, полученная из клещей Hyalomma anatolicum, часто используется для выделения переносимых клещами внутриклеточных инфекционных агентов.
Цель работы – изучение клеточной линии HAE/CTVM8 с помощью высокопроизводительного секвенирования с целью поиска вирусов в ней.
Материалы и методы. Культуральную жидкость клеток HAE/CTVM8 ультрацентрифугировали. Полученный осадок использовали для высокопроизводительного секвенирования после выделения РНК, реакции обратной транскрипции и синтеза второй цепи. Полученные прочтения фильтровали по длине и качеству в программе Trimmomatic, после чего собирали контиги с помощью программы SPAdes и анализировали их на присутствие вирусных последовательностей. Финальная сборка генома вируса осуществлялась в программе Ugene. Выравнивание последовательностей производили с использованием программы MAFFT. Построение филогенетических деревьев производилось с применением программы IQ-TREE.
Результаты. Выявлена персистенция одного вируса – Liman tick virus (LMTV) – в культуре клеток HAE/CTVM8. Филогенетически LMTV принадлежит новому семейству Chuviridae, состоящему из вирусов, обнаруженных с помощью высокопроизводительного секвенирования, вирусологическая характеристика которых отсутствует.
Заключение. Полученная в настоящем исследовании информация крайне важна для использования культуры клеток HAE/CTVM8 в научных исследованиях и изоляции новых вирусов. Наше исследование показывает, что клеточная линия HAE/CTVM8 с персистирующим в ней LMTV представляет собой готовую систему для изучения репродукции представителей семейства Chuviridae.
Ключевые слова
Полный текст
Введение
Клещи являются переносчиками возбудителей опасных заболеваний, таких как болезнь Лайма, клещевой энцефалит и многие другие [1, 2]. В связи с этим изучение биологии, физиологии клещей и способов борьбы с ними приобретает все большую актуальность. Культуры клеток клещей стали незаменимым инструментом для исследователей [3]. В первую очередь они крайне полезны для изучения физиологии и генетики клещей [3, 4], однако, гораздо чаще их используют для размножения переносимых клещами патогенных вирусов [5, 6] и бактерий [7].
Попытки культивировать клетки клещей продолжаются более 50 лет. Ранние исследования привели к получению первичных культур клеток и/или тканей клещей, способных выживать до 6 мес [8]. Позднее были получены перевиваемые клетки разных видов клещей, способные культивироваться в лабораторных условиях длительное время [3, 9].
Большинство доступных в настоящее время клеточных линий клещей были получены из эмбриональных клеток. Такая специфика получения культур клеток, а также ограниченность наших знаний о вироме клещей приводит к тому, что во многих культурах клеток клещей персистируют вирусы [10–12]. Например, Orbivirus saintcroixense (St. Croix River virus) был обнаружен в культурах клеток IDE2, полученных из клещей Ixodes scapularis, и RA243 и RA257 из клещей Rhipicephalus appendiculatus [10, 11]. В культуре клеток IRE/CTVM19 было обнаружено сразу три рабдоподобных вируса: IRE/CTVM19-associated rhabdovirus, Chimay rhabdovirus и Norway mononegavirus 1 [12]. Кроме того, скрининг большого количества доступных культур клеток клещей при помощи пан-найровирусных олигонуклеотидов дал положительный результат, хотя остались сомнения, является ли амплификация специфичной [10]. Таким образом, сами по себе культуры клеток клещей могут быть источником новых вирусов. Тем не менее использование клеток с персистентной вирусной инфекцией может повлиять на результаты научных исследований, особенно при изоляции и изучении свойств других вирусных или бактериальных внутриклеточных агентов.
Современные подходы к обнаружению новых вирусов предполагают использование высокопроизводительного секвенирования в комбинации с биоинформатическими методами, что позволяет открывать десятки и сотни новых вирусов в процессе работы [13–16]. Такой подход показал себя крайне эффективным при описании новых вирусов членистоногих [14, 16], в том числе и клещей [13, 15], а также был использован нами ранее для описания вирусов, персистирующих в культуре клеток IRE/CTVM19 [12].
Одним из достижений высокопроизводительного секвенирования стало недавнее выделение нового семейства (−)РНК-содержащих вирусов – Chuviridae. На сегодняшний момент данное семейство включает в себя 16 родов и 43 вида вирусов, которые были детектированы в паукообразных, ракообразных, насекомых, рыбах и рептилиях. Самым крупным родом семейства является род Mivirus, включающий в себя 10 видов, которые в основном ассоциированы с разными видами иксодовых клещей. Неклассифицированные чувирусные последовательности были обнаружены в головоногих моллюсках, термитах и черепахах. Семейство Chuviridae является самым большим в порядке Jingchuvirales, который родственен порядку Mononegavirales, объединяющему многие хорошо известные (−)РНК-содержащие вирусы [17].
Согласно биоинформатическим данным, представители Chuviridae – это вирусы с одноцепочечной (−)РНК, которые кодируют от 2 до 4 открытых рамок считывания, включая полимеразу, гликопротеин и нуклеопротеин. Геномы разных представителей Chuviridae могут быть линейными или кольцевыми, сегментированными или несегментированными [17].
Культура клеток HAE/CTVM8 была получена из клещей Hyalomma anatolicum [9] и рутинно используется для изоляции и поддержания различных вирусов [8, 18–20]. При этом на данный момент остается неизвестным, содержит ли эта культура какие-либо персистирующие вирусы. Имеются лишь некоторые данные, позволяющие предположить присутствие неизвестных найроподобных вирусов в этой культуре клеток [10].
Цель работы – изучение клеточной линии HAE/CTVM8 с помощью высокопроизводительного секвенирования с целью поиска вирусов в ней.
Материалы и методы
Клетки HAE/CTVM8 культивировали в среде L-15 (ФГАНУ «ФНЦИРИП им. М.П. Чумакова РАН» (Институт полиомиелита), Россия) с 10% триптоз-фосфатного бульона (Difco, США), 20% фетальной бычьей сывороткой (Gibco, США), 2 мМ L-глутамина и антибиотиками, согласно ранее описанной методике [21]. Культуральную жидкость собирали и проводили осветление центрифугированием при 10 000 об/мин в течение 30 мин при +4 °C с использованием ротора SW-28 на центрифуге Optima L-90K Ultracentrifuge (Beckman Coulter, США). После этого супернатант ультрацентрифугировали при 25 000 об/мин в течение 6 ч при +4 °С в том же роторе и центрифуге.
Для проведения высокопроизводительного секвенирования осадок после ультрацентрифугирования растворяли в 200 мкл PBS. Выделение РНК проводили с помощью RNeasy Mini Kit (QIAGEN, Hilden, Германия). Реакцию обратной транскрипции проводили со случайным гексануклеотидным праймером, используя RevertAid (ThermoFisher Scientific, США). Далее осуществляли синтез второй цепи при помощи набора NEBNext Ultra II Non-Directional RNA Second Strand Synthesis Module (NEB, США). Полученную ДНК очищали с использованием Ampure XP (Beckman Coulter, США). Библиотеки готовили с использованием набора NEBNext Fast DNA Library Prep Set for Ion Torrent (NEB, США) и секвенировали на приборе Ion S5XL с использованием чипа Ion 530 Chip.
Полученные прочтения фильтровали по длине (не менее 35 нт) и качеству (Q20) с использованием программы Trimmomatic v. 0.39 [22]. Качество прочтений проверяли с помощью программы FaQCs [23]. Обработанные прочтения собирали в контиги с помощью программы SPAdes v. 3.13.0 используя опцию «-rnaviral». Полученные контиги фильтровали от последовательностей с низкой сложностью и заведомо невирусных последовательностей по ранее описанной методике [24]. В оставшихся контигах поиск вируссодержащих контигов выполняли по ранее описанной методике [24]. Для улучшения полученной сборки и отсечения возможных ошибок было дополнительно проведено картирование прочтений с использованием в качестве референсного генома Liman tick virus (номер GenBank MN542376) в программе Ugene v. 50.0 [25].
Для филогенетического анализа были получены полные аминокислотные последовательности белка L (РНК-полимеразы семейства Chuviridae) для выявленного вируса, все полные последовательности белка L LMTV, доступные в базе данных GenBank, несколько выбранных представителей Mivirus boleense, а также представители родов и филогенетических клад, входящих в семейство Chuviridae. Последовательности выравнивали с помощью программы MAFFT v. 7.310, используя алгоритм E-INS-i [26]. Неопределенно выравненные участки обрезали при помощи программы TrimAL v. 1.4. rev 15 с автоматической детекцией [27]. Филогенетическое дерево было построено при помощи метода максимального правдоподобия в программе IQ-TREE v. 2.3.2 [28] с 1000 bootstrap-реплик и автоматической детекцией модели замен [29]. Филогенетические деревья визуализировали в программе FigTree v. 1.4.4. Геном был аннотирован вручную и визуализировался при помощи инструмента GenomeDrawing (https://github.com/justNo4b/GenomeDrawing).
Результаты
В ходе работы было выполнено высокопроизводительное секвенирование транскриптома культуральной жидкости чистой культуры клеток HAE/CTVM8, полученной из клещей H. anatolicum. В результате фильтрования прочтений по длине и качеству было получено 2,2 млн прочтений. Последующая обработка данных позволила выявить в полученных прочтениях присутствие генома одного вируса. Предварительный анализ показал, что данный вирус генетически близок к LMTV: 92,5% сходства по нуклеотидному составу полного генома, согласно программе blastn.
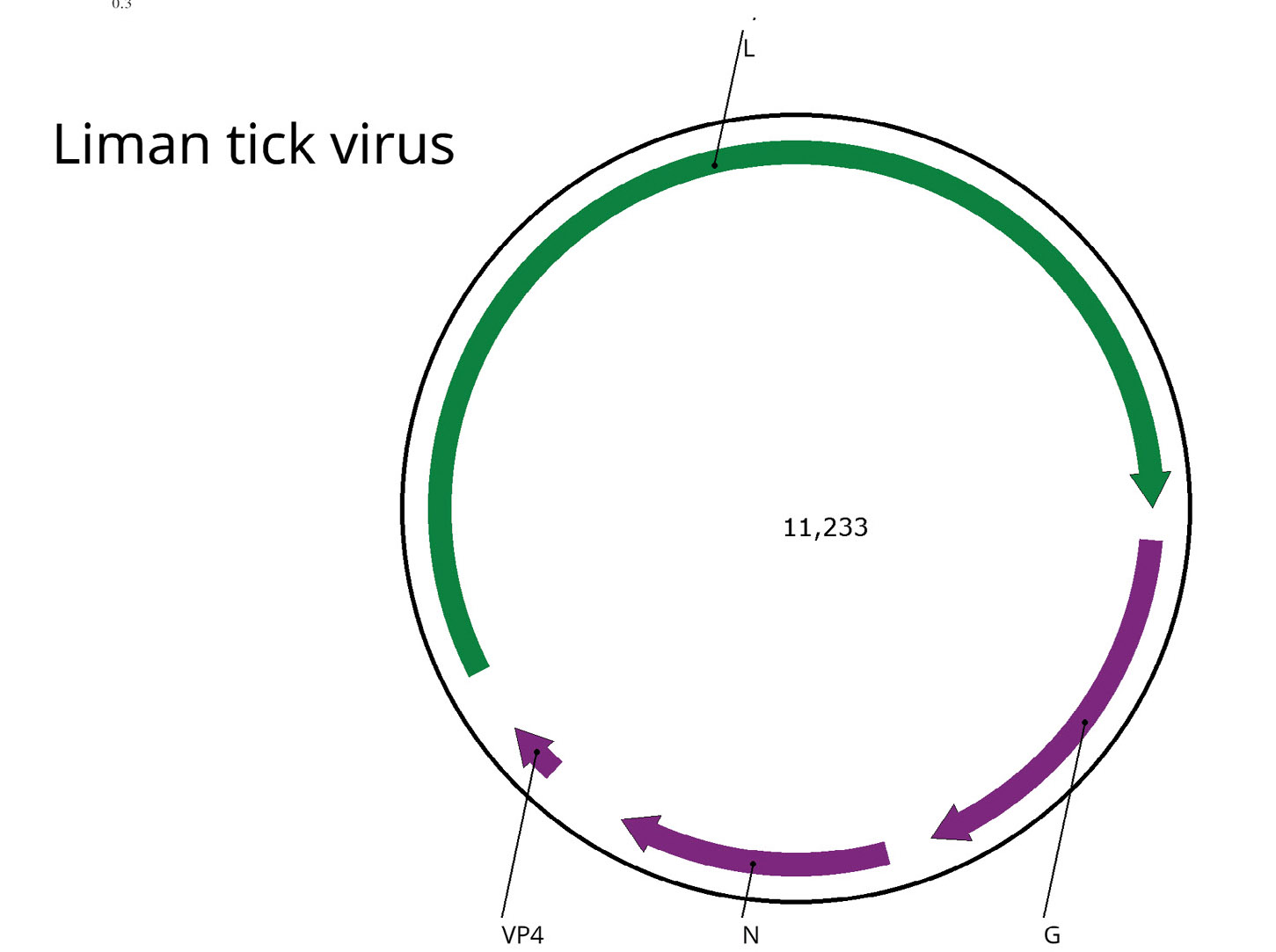
Детальный анализ сборки генома и сравнение его с референсным позволило установить, что в нашем случае имеются прямые повторы на концах контига, что свидетельствует о кольцевой природе генома LMTV. Это неоднократно описано для представителей семейства Chuviridae [17] и показано ранее для LMTV [30]. Таким образом, нам удалось собрать полный геном данного вируса. Карта генома представлена рисунке (б). Геном вируса был выложен в международной базе GenBank под номером PQ613839.
Рисунок. Структура генома и филогенетические взаимоотношения вируса Liman tick.
а ‒ укорененное в среднюю точку филогенетическое дерево семейства Chuviridae. Дерево было построено методом максимального правдоподобия с использованием аминокислотных последовательностей РНК-зависимой РНК-полимеразы (1000 реплик bootstrap; указаны узлы с поддержкой bootstrap ≥ 85%). Шкала представляет количество аминокислотных замен на сайт. Клада Liman tick вирусов выделена желтым цветом. Liman tick вирус, обнаруженный в данной работе, выделен красным цветом; б ‒ схема генома Liman tick вируса. Открытые рамки считывания (ОРС) показаны фиолетовым цветом. ОРС, кодирующая вирусную полимеразу, отмечена зеленым цветом.
Figure. Genomic structure and phylogenetic relationships of the Liman tick virus.
a ‒ Midpoint-rooted phylogenetic tree of the family Chuviridae. Maximum-likelihood phylogenetic tree was constructed using the amino acid sequences of the RdRp (1000 bootstrap replicates; nodes with ≥ 85% bootstrap support are indicated). The scale bar represents the number of amino acid substitutions per site. The Liman tick viruses clade is marked in yellow. Liman tick virus detected in the current work marked in red; b ‒ Scheme of the Liman tick virus genome. ORFs are shown in purple. The RdRp-encoding ORF is marked in green.
Количество прочтений LMTV в образце является достаточно большим – 2,42% относительно общего количества прочтений, что является косвенным показателем того, что вирус активно реплицируется при персистенции в культуре клеток HAE/CTVM8.
Филогенетический анализ, выполненный по последовательностям РНК-зависимой РНК-полимеразы, показал, что обнаруженный вирус оказался близким родственником и формировал монофилетическую группу с другими изолятами LMTV из клещей рода Hyalomma. Сама группа LMTV формировала монофилетическую группу с изолятами Bole tick virus 3 (Mivirus boleense) и попадала в род Mivirus (рисунок (а)).
Обсуждение
Культура клеток HAE/CTVM8 рутинно используется для вирусологических исследований [8, 18–20]. Несмотря на то что, по некоторым данным, эта культура содержит персистирующие вирусы [10], их геномы до сих пор оставались неизвестными, а сама культура не была исследована при помощи современных подходов. Ранее мы описали наличие трех рабдовирусов в культуре клеток IRE/CTVM19 [12].
В настоящей работе, применив схожую методику, мы обнаружили геном LMTV в культуре клеток HAE/CTVM8, которая была получена в 1991 г., что свидетельствует о долговременном присутствии LMTV в этой системе. При этом поскольку собранный геном является полным и кольцевым, встроенность его в геном хозяина крайне маловероятна. Настоящее исследование подтверждает эффективность метагеномного подхода в характеристике вновь полученных культур клеток.
Геном LTMV был впервые обнаружен в клещах H. anatolicum, собранных в Лиманском районе Астраханской области Российской Федерации, и опубликован в GenBank в 2020 г. д-ром Альховским и соавт. (MN542376.1). Позднее новый изолят этого вируса был обнаружен в клещах H. rufipes, собранных с верблюдов в Garrisa County (Кения). Более того, у одного из исследованных верблюдов были обнаружены антитела к LMTV, а сам вирус – в крови [30]. Эти данные указывают на то, что этот вирус может быть арбовирусом.
Согласно биоинформатическим данным, чувирусы обладают чрезвычайно интересной биологией, поскольку включают в себя вирусы с кольцевой одноцепочечной (−)РНК. При этом репликация и строение вириона представителей семейства Chuviridae остаются неизученными [17]. Таким образом, культура клеток HAE/CTVM8 является готовой моделью для изучения репликации и биологии семейства Chuviridae.
Хорошо описаны примеры, когда присутствие одного вируса в культуре клеток оказывает существенное влияние на размножение других – как близкородственных, так и филогенетически удаленных вирусов [31–33]. Пока остается неясным, как LMTV может взаимодействовать с другими вирусами, однако наличие данного вируса в культуре клеток HAE/CTVM8, несомненно, следует учитывать при проведении дальнейших экспериментов.
Заключение
Полученная в настоящем исследовании информация крайне важна для использования культуры клеток HAE/CTVM8 в научных исследованиях и изоляции новых вирусов. Клеточная линия HAE/CTVM8 с персистирующим в ней LMTV представляет собой готовую систему для изучения репродукции представителей семейства Chuviridae.
Об авторах
Александр Геннадьевич Литов
ФГАНУ «Федеральный научный центр исследований и разработки иммунобиологических препаратов имени М.П. Чумакова РАН» (Институт полиомиелита); Институт трансляционной медицины и биотехнологии ФГАОУ ВО «Первый Московский государственный медицинский университет имени И.М. Сеченова» Минздрава России (Сеченовский Университет)
Автор, ответственный за переписку.
Email: novosti-wxo@yandex.ru
ORCID iD: 0000-0002-6086-3655
канд. биол. наук, ведущий научный сотрудник лаборатории биологии арбовирусов
Россия, 108819, г. Москва; 119991, г. МоскваАлексей Михайлович Щетинин
ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
Email: shchetinin.alexey@yandex.ru
ORCID iD: 0000-0003-1842-3899
научный сотрудник лаборатории механизмов популяционной изменчивости патогенных микроорганизмов
Россия, 123098, г. МоскваИван Сергеевич Холодилов
ФГАНУ «Федеральный научный центр исследований и разработки иммунобиологических препаратов имени М.П. Чумакова РАН» (Институт полиомиелита)
Email: ivan-kholodilov@bk.ru
ORCID iD: 0000-0002-3764-7081
канд. мед. наук, ведущий научный сотрудник лаборатории биологии арбовирусов
Россия, 108819, г. МоскваОксана Андреевна Белова
ФГАНУ «Федеральный научный центр исследований и разработки иммунобиологических препаратов имени М.П. Чумакова РАН» (Институт полиомиелита)
Email: mikasusha@bk.ru
ORCID iD: 0000-0002-9040-0774
канд. биол. наук, ведущий научный сотрудник лаборатории биологии арбовирусов
Россия, 108819, г. МоскваАнна Сергеевна Калянова
ФГАНУ «Федеральный научный центр исследований и разработки иммунобиологических препаратов имени М.П. Чумакова РАН» (Институт полиомиелита)
Email: annakalyanova@bk.ru
ORCID iD: 0009-0003-1154-3852
младший научный сотрудник лаборатории биологии арбовирусов
Россия, 108819, г. МоскваВладимир Алексеевич Гущин
ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России; ФГАОУ ВО «Первый Московский государственный медицинский университет имени И.М. Сеченова» Минздрава России (Сеченовский Университет)
Email: wowaniada@gmail.com
ORCID iD: 0000-0002-9397-3762
д-р биол. наук, ведущий научный сотрудник, заведующий лабораторией механизмов популяционной изменчивости патогенных микроорганизмов
Россия, 123098, г. Москва; 119991, г. МоскваГалина Григорьевна Карганова
ФГАНУ «Федеральный научный центр исследований и разработки иммунобиологических препаратов имени М.П. Чумакова РАН» (Институт полиомиелита); Институт трансляционной медицины и биотехнологии ФГАОУ ВО «Первый Московский государственный медицинский университет имени И.М. Сеченова» Минздрава России (Сеченовский Университет)
Email: karganova@bk.ru
ORCID iD: 0000-0002-8901-6206
д-р биол. наук, профессор, ведущий научный сотрудник, заведующая лабораторией биологии арбовирусов
Россия, 108819, г. Москва; 119991, г. МоскваСписок литературы
- Marques A.R., Strle F., Wormser G.P. Comparison of Lyme disease in the United States and Europe. Emerg. Infect. Dis. 2021; 27(8): 2017–24. https://doi.org/10.3201/eid2708.204763
- Ruzek D., Avšič Županc T., Borde J., Chrdle A., Eyer L., Karganova G., et al. Tick-borne encephalitis in Europe and Russia: Review of pathogenesis, clinical features, therapy, and vaccines. Antiviral. Res. 2019; 164: 23–51. https://doi.org/10.1016/j.antiviral.2019.01.014
- Bell-Sakyi L., Zweygarth E., Blouin E.F., Gould E.A., Jongejan F. Tick cell lines: tools for tick and tick-borne disease research. Trends. Parasitol. 2007; 23(9): 450–7. https://doi.org/10.1016/j.pt.2007.07.009
- Mangia C., Vismarra A., Kramer L., Bell-Sakyi L., Porretta D., Otranto D., et al. Evaluation of the in vitro expression of ATP binding-cassette (ABC) proteins in an Ixodes ricinus cell line exposed to ivermectin. Parasit. Vectors. 2016; 9: 215. https://doi.org/10.1186/s13071-016-1497-2
- Kholodilov I.S., Litov A.G., Klimentov A.S., Belova O.A., Polienko A.E., Nikitin N.A., et al. Isolation and characterisation of Alongshan virus in Russia. Viruses. 2020; 12(4): 362. https://doi.org/10.3390/v12040362
- Palomar A.M., Premchand-Branker S., Alberdi P., Belova O.A., Moniuszko-Malinowska A., Kahl O., et al. Isolation of known and potentially pathogenic tick-borne microorganisms from European ixodid ticks using tick cell lines. Ticks Tick Borne Dis. 2019; 10(3): 628–38. https://doi.org/10.1016/j.ttbdis.2019.02.008
- Husin N.A., Khoo J.J., Zulkifli M.M.S., Bell-Sakyi L., AbuBakar S. Replication kinetics of Rickettsia raoultii in tick cell lines. Microorganisms. 2021; 9(7): 1370. https://doi.org/10.3390/microorganisms9071370
- Salata C., Moutailler S., Attoui H., Zweygarth E., Decker L., Bell-Sakyi L. How relevant are in vitro culture models for study of tick-pathogen interactions? Pathog. Glob. Health. 2021; 115(7-8): 437–55. https://doi.org/10.1080/20477724.2021.1944539
- Bell-Sakyi L. Continuous cell lines from the tick Hyalomma anatolicum anatolicum. J. Parasitol. 1991; 77(6): 1006–8.
- Alberdi M.P., Dalby M.J., Rodriguez-Andres J., Fazakerley J.K., Kohl A., Bell-Sakyi L. Detection and identification of putative bacterial endosymbionts and endogenous viruses in tick cell lines. Ticks Tick Borne. Dis. 2012; 3(3): 137–46. https://doi.org/10.1016/j.ttbdis.2012.05.002
- Attoui H., Stirling J.M., Munderloh U.G., Billoir F., Brookes S.M., Burroughs J.N., et al. Complete sequence characterization of the genome of the St Croix River virus, a new orbivirus isolated from cells of Ixodes scapularis. J. Gen. Virol. 2001; 82(Pt. 4): 795–804. https://doi.org/10.1099/0022-1317-82-4-795
- Litov A.G., Shchetinin A.M., Kholodilov I.S., Belova O.A., Gadzhikurbanov M.N., Ivannikova A.Y., et al. High-throughput sequencing reveals three rhabdoviruses persisting in the IRE/CTVM19 cell line. Viruses. 2024; 16(4): 576. https://doi.org/10.3390/v16040576
- Harvey E., Rose K., Eden J.S., Lo N., Abeyasuriya T., Shi M., et al. Extensive diversity of RNA viruses in Australian ticks. J. Virol. 2019; 93(3): e01358–18. https://doi.org/10.1128/JVI.01358-18
- Li C.X., Shi M., Tian J.H., Lin X.D., Kang Y.J., Chen L.J., et al. Unprecedented genomic diversity of RNA viruses in arthropods reveals the ancestry of negative-sense RNA viruses. Elife. 2015; 4: e05378. https://doi.org/10.7554/eLife.05378
- Ni X.B., Cui X.M., Liu J.Y., Ye R.Z., Wu Y.Q., Jiang J.F., et al. Metavirome of 31 tick species provides a compendium of 1,801 RNA virus genomes. Nat. Microbiol. 2023; 8(1): 162–73. https://doi.org/10.1038/s41564-022-01275-w
- Shi M., Lin X.D., Tian J.H., Chen L.J., Chen X., Li C.X., et al. Redefining the invertebrate RNA virosphere. Nature. 2016; 540(7634): 539–43. https://doi.org/10.1038/nature20167
- Kuhn J.H., Dheilly N.M., Junglen S., Paraskevopoulou S., Shi M., Di Paola N. ICTV virus taxonomy profile: Jingchuvirales 2023. J. Gen. Virol. 2023; 104(12): 001924. https://doi.org/10.1099/jgv.0.001924
- Kholodilov I.S., Belova O.A., Ivannikova A.Y., Gadzhikurbanov M.N., Makenov M.T., Yakovlev A.S., et al. Distribution and characterisation of tick-borne flavi-, flavi-like, and phenuiviruses in the Chelyabinsk region of Russia. Viruses. 2022; 14(12): 2699. https://doi.org/10.3390/v14122699
- Salata C., Monteil V., Karlberg H., Celestino M., Devignot S., Leijon M., et al. The DEVD motif of Crimean-Congo hemorrhagic fever virus nucleoprotein is essential for viral replication in tick cells. Emerg. Microbes Infect. 2018; 7(1): 190. https://doi.org/10.1038/s41426-018-0192-0
- Salvati M.V., Salaris C., Monteil V., Del Vecchio C., Palù G., Parolin C., et al. Virus-derived DNA forms mediate the persistent infection of tick cells by Hazara virus and Crimean-Congo hemorrhagic fever virus. J. Virol. 2021; 95(24): e0163821. https://doi.org/10.1128/JVI.01638-21
- Kholodilov I.S., Belova O.A., Morozkin E.S., Litov A.G., Ivannikova A.Y., Makenov M.T., et al. Geographical and tick-dependent distribution of flavi-like Alongshan and Yanggou tick viruses in Russia. Viruses. 2021; 13(3): 458. https://doi.org/10.3390/v13030458
- Bolger A.M., Lohse M., Usadel B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 2014; 30(15): 2114–20. https://doi.org/10.1093/bioinformatics/btu170
- Lo C.C., Chain P.S. Rapid evaluation and quality control of next generation sequencing data with FaQCs. BMC Bioinformatics. 2014; 15(1): 366. https://doi.org/10.1186/s12859-014-0366-2
- Litov A.G., Semenyuk I.I., Belova O.A., Polienko A.E., Thinh N.V., Karganova G.G., et al. Extensive diversity of viruses in millipedes collected in the Dong Nai biosphere reserve (Vietnam). Viruses. 2024; 16(9): 1486. https://doi.org/10.3390/v16091486
- Okonechnikov K., Golosova O., Fursov M. Unipro UGENE: a unified bioinformatics toolkit. Bioinformatics. 2012; 28(8): 1166–7. https://doi.org/10.1093/bioinformatics/bts091
- Katoh K., Standley D.M. MAFFT multiple sequence alignment software version 7: improvements in performance and usability. Mol. Biol. Evol. 2013; 30(4): 772–80. https://doi.org/10.1093/molbev/mst010
- Capella-Gutiérrez S., Silla-Martínez J.M., Gabaldón T. trimAl: a tool for automated alignment trimming in large-scale phylogenetic analyses. Bioinformatics. 2009; 25(15): 1972–3. https://doi.org/10.1093/bioinformatics/btp348
- Nguyen L.T., Schmidt H.A., von Haeseler A., Minh B.Q. IQ-TREE: a fast and effective stochastic algorithm for estimating maximum-likelihood phylogenies. Mol. Biol. Evol. 2015; 32(1): 268–74. https://doi.org/10.1093/molbev/msu300
- Kalyaanamoorthy S., Minh B.Q., Wong T.K.F., von Haeseler A., Jermiin L.S. ModelFinder: fast model selection for accurate phylogenetic estimates. Nat. Methods. 2017; 14(6): 587–9. https://doi.org/10.1038/nmeth.4285
- Zhang Y., Hu B., Agwanda B., Fang Y., Wang J., Kuria S., et al. Viromes and surveys of RNA viruses in camel-derived ticks revealing transmission patterns of novel tick-borne viral pathogens in Kenya. Emerg. Microbes Infect. 2021; 10(1): 1975–87. https://doi.org/10.1080/22221751.2021.1986428
- Abrao E.P., da Fonseca B.A. Infection of mosquito cells (C6/36) by Dengue-2 virus interferes with subsequent infection by yellow fever virus. Vector. Borne Zoonotic Dis. 2016; 16(2): 124–30. https://doi.org/10.1089/vbz.2015.1804
- Kuwata R., Isawa H., Hoshino K., Sasaki T., Kobayashi M., Maeda K., et al. Analysis of mosquito-borne flavivirus superinfection in Culex tritaeniorhynchus (Diptera: Culicidae) cells persistently infected with Culex flavivirus (Flaviviridae). J. Med. Entomol. 2015; 52(2): 222–9. https://doi.org/10.1093/jme/tju059
- Patterson E.I., Kautz T.F., Contreras-Gutierrez M.A., Guzman H., Tesh R.B., Hughes G.L., et al. Negeviruses reduce replication of alphaviruses during coinfection. J. Virol. 2021; 95(14): e0043321. https://doi.org/10.1128/JVI.00433-21
Дополнительные файлы