Вариабельность неструктурных белков у вариантов ВИЧ-1 суб-субтипа А6 (Retroviridae: Orthoretrovirinae: Lentivirus: Human immunodeficiency virus-1, sub-subtype A6), циркулирующих в разных регионах Российской Федерации
- Авторы: Антонова А.А.1, Лебедев А.В.1, Ожмегова Е.Н.1, Шлыкова А.В.2, Лаповок И.А.2, Кузнецова А.И.1
-
Учреждения:
- Институт вирусологии им. Д.И. Ивановского ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии им. почетного академика Н.Ф. Гамалеи» Минздрава России
- ФБУН «Центральный научно-исследовательский институт эпидемиологии» Роспотребнадзора
- Выпуск: Том 69, № 5 (2024)
- Страницы: 470-480
- Раздел: ОРИГИНАЛЬНЫЕ ИССЛЕДОВАНИЯ
- URL: https://virusjour.crie.ru/jour/article/view/16677
- DOI: https://doi.org/10.36233/0507-4088-262
- EDN: https://elibrary.ru/wbbkuq
- ID: 16677
Цитировать
Аннотация
Введение. Неструктурные белки ВИЧ-1 являются перспективными мишенями для разработки вакцин и для создания подходов персонализированной медицины. На протяжении многих лет на территории Российской Федерации доминирует суб-субтип А6 ВИЧ-1. Однако имеющиеся географические, экономические и демографические особенности страны могут способствовать формированию различий между вариантами вируса, циркулирующими в разных регионах России.
Цель работы: провести сравнительный анализ консенсусных последовательностей неструктурных белков ВИЧ-1 вариантов А6, циркулирующих в Амурской области, в гг. Архангельске, Иркутске и Мурманске.
Материалы и методы. Исследовали 48 образцов цельной крови, которые были получены от ВИЧ-инфицированных пациентов без опыта приема терапии, наблюдавшихся в центрах по профилактике и борьбе со СПИДом в гг. Архангельске, Иркутске, Мурманске и в Амурской области. Получали полногеномные нуклеотидные последовательности ВИЧ-1, субтипировали. Формировали консенсусные последовательности каждого неструктурного белка ВИЧ-1 для каждого анализируемого региона. Дополнительно формировали референсные последовательности неструктурных белков ВИЧ-1 суб-субтипа А6 на основе полногеномных последовательностей, загруженных из международной базы данных Los Alamos. Сравнение консенсусных и референсных последовательностей осуществляли с помощью программ MEGA v.10.2.2 и PSIPRED.
Результаты. Впервые представлены референсные последовательности белков Vif, Vpr и Nef ВИЧ-1 суб-субтипа А6. Для белка Vpr различий в консенсусных последовательностях вариантов вируса, циркулирующих в разных регионах РФ, не обнаружено. Для консенсусных последовательностей белков Tat, Rev, Vpu, Vif и Nef вариантов ВИЧ-1 из разных регионов РФ определены характерные особенности.
Заключение. Ограничением проведенного исследования является небольшая выборка образцов. В целом полученные результаты указывают на существующее разнообразие неструктурных белков ВИЧ-1 суб-субтипа А6 в разных регионах страны и обозначают актуальность исследования полиморфизма неструктурных белков вариантов вируса в разных регионах в будущем.
Ключевые слова
Полный текст
Введение
Геном вируса иммунодефицита человека 1-го типа (ВИЧ-1) (Retroviridae: Orthoretrovirinae: Lentivirus: Human immunodeficiency virus-1) кодирует 6 неструктурных белков: Tat, Rev, Vpu, Vif, Vpr, Nef. Неструктурные белки создают необходимые условия для репликации вируса и обеспечивают защиту вируса от действия иммунной системы хозяина [1]. Ранее проведенные исследования показали, что в неструктурных белках ВИЧ-1 содержатся эпитопы, которые могут быть использованы для разработки вакцин [2–4]. Отмечалось, что иммунизация конструкциями на основе белка Tat способствовала восстановлению уровня CD4-клеток и сокращению вирусных резервуаров [5]. В настоящее время разработка новых подходов к стимуляции иммунного ответа на основе неструктурных белков продолжается [6–8]. В связи с этим изучение вариабельности неструктурных белков ВИЧ-1 является важной задачей и создает основу для подобного рода разработок. Кроме того, в неструктурных белках ВИЧ-1 были определены аминокислотные замены, ассоциированные как с изменением скорости течения ВИЧ-инфекции, так и с развитием коморбидных заболеваний у ВИЧ-инфицированных пациентов [1]. Таким образом, изучение разнообразия неструктурных белков ВИЧ-1 также может стать платформой для разработки подходов персонализированной медицины.
Молекулярная эпидемиология ВИЧ-1 в России имеет свои характерные особенности. Изначально активное распространение ВИЧ-инфекции в России было связано с проникновением в 90-х годах XX в. в среду потребителей инъекционных наркотиков (ПИН) вируса суб-субтипа А6 и его последующим быстрым распространением внутри этой социальной группы во всех регионах страны [9]. Затем было отмечено постепенное снижение доли ПИН в популяции ВИЧ-инфицированных на территории РФ с одновременным увеличением числа случаев передачи вируса при гетеросексуальных контактах [10]. В настоящее время на территории России суб-субтип A6 по-прежнему остается доминирующим (82,9%) генетическим вариантом ВИЧ-1, однако при этом отмечается постоянный рост генетического разнообразия вируса [11, 12].
Эпидемический процесс ВИЧ-инфекции на территории Российской Федерации вызывает большой интерес и обусловлен рядом особенностей страны: Россия по размерам территории занимает 1-е место в мире, является многонациональной и многоконфессиональной страной, что определяет разницу культур и поведения, образа жизни и моделей мобильности населения. Кроме того, наша страна граничит с 18 государствами, обладает многочисленными транспортными коридорами, что способствует высокому генетическому разнообразию и быстрому распространению ВИЧ-1 за счет миграционных процессов [13, 14]. Так, отмечалось, что доля и разнообразие циркулирующих не-А6 вариантов может существенно отличаться между разными федеральными округами РФ [12]. Вероятно, географическое положение, особенности социально-экономического развития региона и состава населения могут оказывать влияние также на селекцию циркулирующих генетических вариантов вируса внутри суб-субтипа А6.
Принимая во внимание вышесказанное, мы предположили, что неструктурные белки могут различаться у вариантов ВИЧ-1 суб-субтипа А6, циркулирующих в разных регионах РФ. До настоящего момента исследований по изучению разнообразия неструктурных белков вариантов ВИЧ-1 суб-субтипа А6, циркулирующих в разных регионах России, не проводилось.
Цель ‒ сравнительный анализ консенсусных последовательностей неструктурных белков ВИЧ-1 суб-субтипа А6 у вариантов вируса, циркулирующих в разных регионах России: в Амурской области, в гг. Архангельске, Иркутске и Мурманске.
Материалы и методы
Материалом настоящего исследования служили клинические образцы цельной крови, полученные от 48 наивных (без опыта приема антиретровирусной терапии (АРТ), ВИЧ-инфицированных пациентов из следующих регионов России: Амурской области, гг. Архангельска, Иркутска и Мурманска (табл. 1). Забор крови у пациентов осуществляли единоразово в период 2012–2014 гг. в рамках реализации проекта CHAIN 7-й Рамочной программы Европейского сообщества «Единая сеть по изучению лекарственной устойчивости к антиретровирусным препаратам» (https://cordis.europa.eu/project/id/223131). Исследование проводилось при добровольном информированном согласии пациентов. Протокол исследования одобрен Этическим комитетом при ФГУН ГНЦ ВБ «Вектор» протокол № 1 от 30 марта 2010 г.
Таблица 1. Характеристики ВИЧ-1-инфицированных пациентов, включенных в исследование
Table 1. Characteristics of HIV-1 infected patients included in the study
Регион Region | Число пациентов Number of patients | Год забора образца Date of sampling | Пол Sex | Возраст Age | Путь инфицирования Route of infection | Стадия заболевания* Stage of HIV-infection* | |||||
муж. male | жен. female | ПИН IDU | гетеро hetero | 2 | 3 | 4 | неизвестно unknown | ||||
Амурская область Amur Region | 10 | 2012 | 8 | 2 | 30,8 (18–41) | 4 | 6 | – | 4 | 5 | 1 |
Архангельск Arkhangelsk | 12 | 2013 | 3 | 9 | 30,3 (18–42) | 5 | 7 | 1 | 10 | 1 | – |
Мурманск Murmansk | 13 | 2013–2014 | 6 | 7 | 33 (26–42) | 10 | 3 | – | 5 | 8 | – |
Иркутск Irkutsk | 13 | 2012 | 10 | 3 | 31,9 (23–49) | 10 | 3 | – | 7 | 6 | – |
Примечание. *В соответствии с клиническими рекомендациями [15].
Note. *In according to clinical recommendation [15].
Образцы анализировали путем массового параллельного секвенирования с помощью набора «АмплиСенс HIV-Resist-NGS» в соответствии с инструкцией производителя (ФБУН «ЦНИИЭ Роспотребнадзора», Россия). Полногеномное секвенирование образцов было выполнено с применением технологии MiSeq и соответствующих наборов MiSeq reagent kits V2 (Illumina, США) путем анализа 4 перекрывающихся специфических фрагментов (общая протяженность анализируемого фрагмента 704–9563 по HXB2).
Определение генетических вариантов полученных полногеномных последовательностей проводили с применением программы Comet (https://comet.lih.lu). Попарное и множественное выравнивание нуклеотидных последовательностей выполняли в программе MEGA v.10.2.2. (megasoftware.net), затем из полученных алайнментов «вырезали» участки, кодирующие соответствующие анализируемые неструктурные белки ВИЧ-1 (Tat, Rev, Vpu, Vif, Vpr, Nef). Для всех последовательностей генов tat, rev, vpu, vif, vpr, nef был проведен филогенетический анализ. Филогенетический анализ осуществляли методом максимального правдоподобия (Maximum Likelihood, ML) c использованием программы IQ-TREE (http://www.iqtree.org). Источником эталонных последовательностей служила база данных Лос-Аламосской лаборатории США (https://www.hiv.lanl.gov/). Модель замещения нуклеотидов определяли в программе jModelTest v.2.1.7 на основании информационного критерия Акаике (Akaike information criterion, AIC). Наиболее подходящей моделью для последующего анализа считали модель с наименьшим значением критерия. Достоверность выведенных филогений оценивали с помощью бутстрэп-теста (bootstrap) и критерия приблизительного отношения правдоподобия Шимодайры–Хасегавы (SH-aLRT) с 1000 послестартовых итераций. Кластеры с поддержкой SH-aLRT > 0,9 считали достоверно установленными. Визуализацию и графическую обработку результатов филогенетического анализа осуществляли в программе iTOL (https://itol.embl.de).
На следующем этапе исследования полученные нуклеотидные последовательности были переведены в аминокислотные с помощью онлайн-инструмента для трансляции, представленного на сайте https://www.bioinformatics.org/sms2/translate.html. Затем с помощью инструмента Simple Consensus Maker (https://www.hiv.lanl.gov/content/sequence/CONSENSUS/SimpCon.html) на основе полученных аминокислотных последовательностей были сформированы консенсусные последовательности для каждого неструктурного белка (Tat, Rev,Vpu, Vif, Vpr, Nef) для каждого региона РФ.
Для проведения дальнейшего сравнительного анализа консенсусных последовательностей дополнительно формировали референсные последовательности для каждого белка. Для этого из международной базы данных Los Alamos (Main Search Interface of HIV Sequence Database (lanl.gov)) были выгружены все имеющиеся полногеномные последовательности ВИЧ-1 суб-субтипа А6 (235 последовательностей на 13.08.2024). Выборка последовательностей не содержала дубликатов: только одна последовательность от одного пациента. Для каждого белка одновременно выгружали аминокислотные и нуклеотидные последовательности. Затем для каждого анализируемого белка в программе MEGA v.10.2.2. (megasoftware.net) нуклеотидные и аминокислотные последовательности сопоставляли между собой. Последовательности, кодирующие неполный белок, были удалены из анализа. Инсерции при формировании референсных последовательностей не учитывали. Референсные последовательности для каждого неструктурного белка (Tat, Rev,Vpu, Vif, Vpr, Nef) были сформированы с помощью инструмента Simple Consensus Maker (https://www.hiv.lanl.gov/content/sequence/CONSENSUS/SimpCon.html) на основе аминокислотных последовательностей.
Дальнейшее сравнение полученных референсных и консенсусных последовательностей проводили с применением программы MEGA v.10.2.2. При этом определяли позиции аминокислот (АК) в консенсусных последовательностях, которые содержали аминокислотные замены относительно референсных последовательностей.
Далее проводили сравнительный анализ вторичных структур консенсусных последовательностей, полученных для каждого региона, с референсными последовательностями анализируемых белков ВИЧ-1 с помощью программы PSIPRED (http://bioinf.cs.ucl.ac.uk/psipred/).
Результаты
Все полученные в ходе настоящего исследования нуклеотидные последовательности (48) были депонированы в международную базу данных генотипов GenBank под следующими номерами (табл. 2).
Таблица 2. Регистрационные номера GenBank использованных в работе нуклеотидных последовательностей ВИЧ-1
Table 2. GenBank accession numbers for the HIV-1 nucleotide sequences used in the work
Регион РФ Region of the Russian Federation | Номера последовательностей GenBank GenBank accession numbers for sequences |
Амурская область Amur region | MH330347, MH330348, MH330349, MH330350, MH330351, MH330352, MH330353, MH330354, MH330355, MH330356 |
Архангельск Arkhangelsk | MG902950, MG902951, MH330337, MH330338, MH330339, MH330340, MH330341, MH330342, MH330343, MH330344, MH330345, MH330346 |
Мурманск Murmansk | MH330370, PP816220, MH330371, MH330372, MH330373, MH330374, MH330375, MH330376, MH330377, MH330378, MH330379, MH330380, MH330381 |
Иркутск Irkutsk | MH330357, MH330358, MH330359, PP816221, MH330361, PP816222, MH330363, MH330364, MH330365, PP816223, MH330367, PP816224, PP816225 |
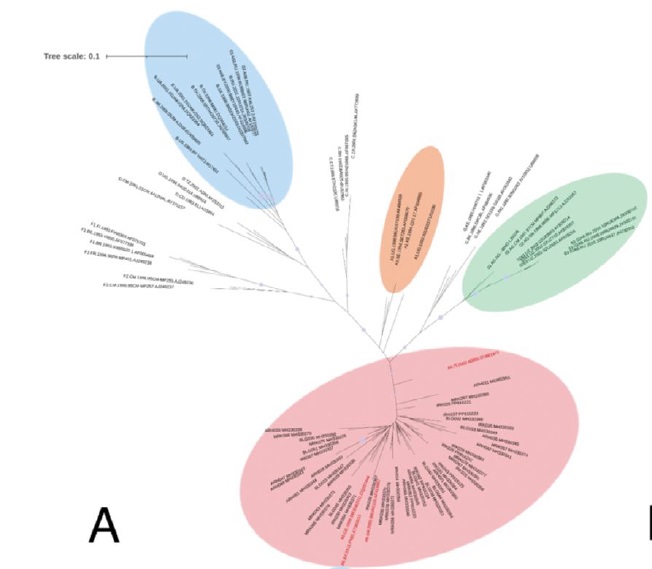
По результатам предварительного субтипирования, все исследуемые нуклеотидные последовательности принадлежали ВИЧ-1 суб-субтипа А6. Проведенный филогенетический анализ подтвердил результаты предварительного субтипирования (рис. 1).
Рис. 1. Филогенетический анализ полученных последовательностей: tat (A), rev (B), vpu (C), vif (D), vpr (E), nef (F).
Цветом на филогенетических деревьях отмечены кластеры наиболее характерных для территории РФ генетических вариантов ВИЧ-1: розовым цветом – ВИЧ-1 суб-субтипа A6, голубым – субтипа B, салатовым – циркулирующих рекомбинантных форм CRF02_AG и CRF63_02A6; также отмечен кластер, образованный референсными последовательностями ВИЧ-1 суб-субтипа A1. Внутри кластера ВИЧ-1 суб-субтипа A6 референсные последовательности обозначены красным цветом, исследуемые последовательности – черным цветом; все остальные кластеры ВИЧ-1 других генетических вариантов (A1, C, D, F1, F2, G) включают исключительно референсные последовательности (HIV Databases (lanl.gov).
Fig. 1. Phylogenetic analysis of the obtained sequences: tat (A), rev (B), vpu (C), vif (D), vpr (E), nef (F).
Clusters of the most typical HIV-1 genetic variants for the territory of the Russian Federation are marked in color on the phylogenetic trees: pink – HIV-1 sub-subtype A6, blue – subtype B, light green – circulating recombinant forms CRF02_AG and CRF63_02A6; a cluster formed by the reference sequences of HIV-1 sub-subtype A1 is also marked. Within the HIV-1 sub-subtype A6 cluster, the reference sequences are shown in red, the studied sequences are shown in black; all other clusters of HIV-1 of other genetic variants (A1, C, D, F1, F2, G) include exclusively reference sequences (HIV Databases (lanl.gov).
На следующем этапе исследования были сформированы консенсусные аминокислотные последовательности каждого неструктурного белка ВИЧ-1 (Tat, Rev, Vpu, Vif, Vpr, Nef) для каждого региона: для Амурской области – на основании 10 последовательностей, для г. Архангельска – 12 последовательностей, для Мурманска – 13, для Иркутска – 13. При их формировании инсерции (аминокислотные вставки) не учитывали. В табл. 3 указаны все инсерции и делеции (точечные мутации, связанные с отсутствием АК в заданной позиции), которые были обнаружены в процессе анализа.
Таблица 3. Инсерции и делеции аминокислот в анализируемых последовательностях*
Table 3. Insertions and deletions of amino acids (a.a.) in the analyzed sequences*
Белок ВИЧ-1 HIV-1 protein | Амурская область Amur region | Архангельск Arkhangelsk | Мурманск Murmansk | Иркутск Irkutsk | ||||
инсерция (N посл-ти) insertion (sequence ID) | делеция (N посл-ти) deletion (sequence ID) | инсерция (N посл-ти) insertion (sequence ID) | делеция (N посл-ти) deletion (sequence ID) | инсерция (N пос-ти) insertion (sequence ID) | делеция (N посл-ти) deletion (sequence ID) | инсерция (N пос-ти) insertion (sequence ID) | делеция (N посл-ти) deletion (sequence ID) | |
Tat | – | – | – | – | 79–80insE (MH330380) | – | 54–55insS (MH330364) | – |
Rev | – | del97–119 (MH330355, (MH330353) | – | del93–99 (MH330341), del94–115 (MH330339) | 33–34insR (MH330380) | – | 8–9insA (MH330364) | del91–97 (MH330358) |
Vpu | – | – | – | – | – | del77 (PP816220) | 7–8insTIV (PP816225) | del5 (PP816223) |
Vif | – | – | – | – | – | – | – | del109–115 (PP816224) |
Vpr | – | – | – | del85–86 (MH330345) | 84–85insI/M (MH330371) | – | – | del85–86 (PP816225) |
Nef | 25–26insPA (MH330352, MH330354), 25–26ins PAASGVE (MH330355), 63–64insEE (MH330355) | del8–11 (MH330351), del8–11 (MH330355) | 25–26insPA (MH330342, MH330343) | Del8–9 (MG902950) | 25–26insPA (MH330371), 25–26insP (MH330370), 25–26insPAAGG[G/V] (MH330378), 25–26ins PXARRAPE (MH330380), 63–64insE (PP816220, MH330380) | – | 25–26insPAP (PP816221), 25–26insPA (PP816225), 25–26insPAA (MH330363), 63–64insE (MH330358) | del8–11 (PP816221, MH330361), del10 (PP816223) |
Примечание. *Расположение инсерций и делеций указано относительно консенсусной последовательности неструктурного белка ВИЧ-1 соответствующего региона.
Note. *The locations of insertions and deletions are shown according to the consensus sequence of the corresponding HIV-1 protein.
Затем на основе последовательностей, загруженных из международной базы данных Los Alamos, формировали референсные последовательности белков. Референсные последовательности белков Tat, Rev, Vif, Vpr были сформированы на основе 235 последовательностей и имели следующие длины: 101 АК, 123 АК, 192 АК, 96 АК соответственно. Референсная последовательность белка Vpu была сформирована на основе 232 последовательностей и содержала 81 АК. Референсная последовательность белка Nef была сформирована на основе 223 последовательностей и содержала 207 АК (рис. 2).
Рис. 2. Референсные последовательности белков Tat, Rev, Vpu, Vif, Vpr, Nef.
Неполярные аминокислоты: G (глицин), A (аланин), V (валин), L (лейцин), I (изолейцин), P (пролин), M (метионин) и F (фенилаланин) ‒ отмечены синим цветом; полярные незаряженные, нейтральные, аминокислоты: S (серин), T (треонин), C (цистеин), N (аспарагин), Q (глутамин) и W (триптофан) – зеленым; gполярные кислые, отрицательно заряженные, аминокислоты: D (аспарагиновая кислота) и E (глутаминовая кислота), Y (тирозин) – оранжевым; полярные основные, положительно заряженные, аминокислоты: K (лизин), R (аргинин) и H (гистидин) – красным [16].
Fig. 2. Reference sequences of the proteins Tat, Rev, Vpu, Vif, Vpr, Nef.
Non-polar amino acids: G (glycine), A (alanine), V (valine), L (leucine), I (isoleucine), P (proline), M (methionine) and F (phenylalanine), – are marked in blue; Polar uncharged, neutral, amino acids: S (serine), T (threonine), C (cysteine), N (asparagine), Q (glutamine) and W (tryptophan) – green; polar acidic, negatively charged, amino acids: D (aspartic acid) and E (glutamic acid), Y (tyrosine) – orange; polar basic, positively charged amino acids: K (lysine), R (arginine) and H (histidine) [16].
Полученные консенсусные последовательности неструктурных белков сравнивали с референсными последовательностями. В табл. 4 указаны позиции, в которых консенсусные последовательности неструктурных белков ВИЧ-1 отдельных регионов России отличались от референсных последовательностей.
Таблица 4. Аминокислотные замены в консенсусных последовательностях неструктурных белков вариантов вирусов, циркулирующих в Амурской области, гг. Архангельске, Мурманске, Иркутске, относительно референсных последовательностей*
Table 4. Amino acid substitutions in the consensus sequences of non-structural proteins of virus variants circulating in the Amur Region, Arkhangelsk, Murmansk, and Irkutsk, relative to the reference sequences*
Белок ВИЧ-1 HIV-1 protein | Амурская область Amur region | Архангельск Arkhangelsk | Мурманск Murmansk | Иркутск Irkutsk |
Tat | P68L, R99P/R | – | – | P68L |
Rev | K39R | K39R, A68E, V109I | K39R, V109I | – |
Vpu | F16S/A | F16F/S/A | L33V | Y73L |
Vif | F39V | E37G, R50K, E92E/K, V125V/L | E37G, R50K, E92R, I98I/V, H127Q | E37G, R50K/R |
Vpr | – | – | – | – |
Nef | I10I/L, K179R | R29T,Y82F, E152E/S, K179R | R29T, I134I/E, Y144Y/F, E152D, K179R, T193T/K | R29P/T, G84A, F136Y, K179R, T193K |
Примечание. *R99P/R обозначает, что в консенсусной последовательности с равной вероятностью в позиции встречались аминокислоты P и R. Жирным шрифтом выделены замены, ассоциированные с изменениями свойств аминокислот: заряженная/незаряженная, полярная/неполярная.
Note. *R99P/R indicates that the consensus sequence contained amino acids P and R with equal probability at the position. Substitutions associated with changes in the properties of amino acids, charged/uncharged or polar/non-polar, are highlighted in bold.
Консенсусы белка Vpr вариантов вирусов, циркулирующих в анализируемых регионах, не содержали замен относительно референсных. В связи с этим сравнительный анализ вторичной структуры белка Vpr в дальнейшем исследовании не проводили.
Результаты прогнозирования вторичных структур референсных последовательностей анализируемых белков ВИЧ-1 представлены в табл. 5.
Таблица 5. Спрогнозированное расположение элементов спиралей и цепей во вторичных структурах референсных последовательностей неструктурных белков ВИЧ-1
Table 5. Predicted arrangement of helical and strand elements in secondary structures of reference sequences of HIV-1 nonstructural proteins
Тип вторичной структуры Secondary structure type | Tat (позиция AК / a.a. position) | Rev (позиция AК / a.a. position) | Vpu (позиция AК/ a.a. position) | Vif (позиция AК / a.a. position) | Nef (позиция AК / a.a. position) |
Спираль / Helix | 32–33, 36–39, 86–95 | 9–24, 35–61 | 3–52, 61–70 | 15–31, 78–80, 100–110, 117–124, 145–153 | 13–21, 38–40,50–51, 57–66, 82–94, 106–110, 151–156, 168–171, 188–192, 196–199, 201–204 |
Цепь / Strand | – | – | – | 4–13, 38–41, 50–59, 63–69, 85–91, 93–97, 128–129 | 102–104, 111–118, 142–148, 181–186 |
Аналогичным образом были спрогнозированы вторичные структуры консенсусных последовательностей неструктурных белков вариантов ВИЧ-1 суб-субтипа А6, циркулирующих в анализируемых регионах России.
При сравнительном анализе спрогнозированных вторичных структур консенсусных последовательностей с референсными были обнаружены следующие отличия:
‒ для варианта белка Tat, характерного для ВИЧ-1 суб-субтипа А6 в Амурской области, содержащего замены P68L и R99P, выявлено смещение расположения спирали из положения 86–95 в положение 85–94;
‒ для варианта белка Vif, характерного для ВИЧ-1 суб-субтипа А6 в Амурской области, – смещение расположения цепи из положения 38–41 в положение 39–41. При этом для вариантов белка Vif ВИЧ-1 суб-субтипа А6, циркулирующих в гг. Архангельске, Мурманске и Иркутске, было отмечено смещение расположения цепи из положения 38–41 в положение 38–39. Дополнительно для белка Vif, характерного для вариантов вируса в г. Мурманске, установлено смещение цепи из положения 93–97 в положение 94–97, а для белка Vif, характерного для вариантов вируса в г. Архангельске, – отсутствие структуры цепи в положении 128–129;
– для вариантов белка Nef, характерного для ВИЧ-1 суб-субтипа А6 в гг. Архангельске, Мурманске и Иркутске, – смещение элемента спирали из положении 13–21 в 14–21. Дополнительно для варианта белка Nef вируса, характерного для г. Архангельска, смещение элемента спирали из положения 57–66 в положение 56–66.
Обсуждение
В настоящее время в России регулярно проводятся исследования гена pol ВИЧ-1 как у вариантов вирусов, циркулирующих в отдельных регионах [17–19], так и в целом по стране [11, 12, 20]. Это объясняется тем, что для лечения ВИЧ-инфекции в основном используются ингибиторы вирусных ферментов – интегразы, протеазы и обратной транскриптазы, которые кодируются геном pol [1, 15]. Соответственно, в гене pol возникает большинство мутаций лекарственной устойчивости к антиретровирусным препаратам, и изучение этой области генома регламентировано нормативными документами в клинической практике. Гены неструктурных белков находятся за пределами анализируемой области генома, и вариабельность неструктурных белков вариантов вируса, циркулирующих в разных регионах страны, остается неизученной. В недавно проведенном нами исследовании было показано, что в некоторых участках белка Tat у вариантов ВИЧ-1 суб-субтипа А6, циркулирующих на территории Московской области, консервативность меньше, чем в общей популяции вариантов ВИЧ-1 суб-субтипа А6 [21].
В основу настоящего исследования было положено предположение о существующей вариабельности неструктурных белков у вариантов ВИЧ-1 суб-субтипа А6, циркулирующих в разных регионах нашей страны. Были проанализированы последовательности неструктурных белков вариантов ВИЧ-1 суб-субтипа А6, полученные в результате анализа клинических образцов цельной крови наивных пациентов, т.е. ранее не получавших АРТ, в период 2012–2014 гг. Пациенты находились под наблюдением в центрах СПИДа Амурской области, гг. Архангельска, Мурманска и Иркутска (табл. 1).
При формировании консенсусных последовательностей для каждого неструктурного белка ВИЧ-1 были отмечены последовательности, содержащие делеции/инсерции (табл. 3), при этом в белке Vif только одна последовательность (PP816224) содержала делецию – del109–115, а наибольшее число делеций/инсерций анализируемые последовательности содержали в белке Nef (табл. 3). Такой результат может быть объяснен тем, что основной функцией белка Vif является противодействие клеточному белку APOBEG3G, тогда как белок Nef обладает многочисленными видами деятельности и контактирует с бо́льшим количеством белков клетки-хозяина, что, соответственно, предполагает наличие более гибкой структуры [1].
Референсные последовательности неструктурных белков ВИЧ-1 суб-субтипа А6 формировали на основе 235 полногеномных последовательностей, загруженных из международной базы данных Los Alamos.
Сформированная референсная последовательность белка Tat в положении 54 и 60 содержала гистидин (H), в положении 57 – глицин (G), а также мотив 78QRD80, характерные для ВИЧ-1 суб-субтипа A6 [21, 22].
Сформированная референсная последовательность белка Rev в положении 41 содержала глутамин (Q) и после положения 95 – мотив QSQGTET, характерные для ВИЧ-1 суб-субтипа A6 [23].
Сформированная референсная последовательность белка Vpu относительно ранее опубликованной последовательности Vpu суб-субтипа А6 в положении 73 вместо лейцина (L) содержала тирозин (Y) [24].
Референсные последовательности белков Vif, Vpr и Nef ВИЧ-1 суб-субтипа А6 были сформированы и представлены впервые.
Несмотря на характерную для белка Vpr вариабельность в COOH-концевой области, о которой сообщалось ранее, консенсусные последовательности белка Vpr из разных регионов РФ не содержали замен относительно референсной последовательности [25].
Консенсусные последовательности белков Tat, Rev, Vpu, Vif и Nef отличались от референсных последовательностей и различались между собой наличием характерных аминокислотных замен. Некоторые из выявленных аминокислотных замен были ассоциированы с изменением химических свойств аминокислот, при этом для белков Tat, Vif и Nef были обнаружены изменения во вторичной структуре белка относительно референсных последовательностей.
Полученные результаты указывают на существование различий в неструктурных белках у вариантов ВИЧ-1 суб-субтипа А6, циркулирующих в разных регионах Российской Федерации, что может быть объяснено «эффектом основателя».
Проведенное исследование имеет ограничение, связанное с небольшой выборкой анализируемых последовательностей. Для подтверждения полученных результатов необходимо проведение дальнейшего ряда исследований полиморфизма неструктурных белков вариантов ВИЧ-1 суб-субтипа А6, циркулирующих в разных регионах страны.
Заключение
Впервые проведен сравнительный анализ консенсусных последовательностей неструктурных белков вариантов ВИЧ-1 суб-субтипа А6, циркулирующих в разных регионах Российской Федерации. В процессе исследования впервые были получены и представлены референсные последовательности белков Vif, Vpr и Nef ВИЧ-1 суб-субтипа А6. Белок Vpr определен как наиболее консервативный. В целом полученные результаты свидетельствуют о присутствующих особенностях в неструктурных белках вариантов ВИЧ-1 суб-субтипа А6 в разных регионах России.
Об авторах
Анастасия Александровна Антонова
Институт вирусологии им. Д.И. Ивановского ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии им. почетного академика Н.Ф. Гамалеи» Минздрава России
Email: anastaseika95@mail.ru
ORCID iD: 0000-0002-9180-9846
канд. биол. наук, научный сотрудник лаборатории вирусов лейкозов
Россия, 123098, г. МоскваАлексей Владимирович Лебедев
Институт вирусологии им. Д.И. Ивановского ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии им. почетного академика Н.Ф. Гамалеи» Минздрава России
Email: lebedevalesha236@gmail.com
ORCID iD: 0000-0001-6787-9345
канд. биол. наук, научный сотрудник лаборатории вирусов лейкозов
Россия, 123098, г. МоскваЕкатерина Никитична Ожмегова
Институт вирусологии им. Д.И. Ивановского ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии им. почетного академика Н.Ф. Гамалеи» Минздрава России
Email: belokopytova.01@mail.ru
ORCID iD: 0000-0002-3110-0843
канд. биол. наук, научный сотрудник лаборатории вирусов лейкозов
Россия, 123098, г. МоскваАнастасия Вениаминовна Шлыкова
ФБУН «Центральный научно-исследовательский институт эпидемиологии» Роспотребнадзора
Email: murzakova_a.v@mail.ru
ORCID iD: 0000-0002-1390-8021
научный сотрудник лаборатории диагностики и молекулярной эпидемиологии ВИЧ-инфекции
Россия, 111123, г. МоскваИлья Андреевич Лаповок
ФБУН «Центральный научно-исследовательский институт эпидемиологии» Роспотребнадзора
Email: i_lapovok@mail.ru
ORCID iD: 0000-0002-6328-1415
канд. биол. наук, старший научный сотрудник
Россия, 111123, г. МоскваАнна Игоревна Кузнецова
Институт вирусологии им. Д.И. Ивановского ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии им. почетного академика Н.Ф. Гамалеи» Минздрава России
Автор, ответственный за переписку.
Email: a-myznikova@list.ru
ORCID iD: 0000-0001-5299-3081
канд. биол. наук, заведующая лаборатории вирусов лейкозов, ведущий научный сотрудник
Россия, 123098, г. МоскваСписок литературы
- Кузнецова А.И. Роль полиморфизма ВИЧ-1 в патогенезе заболевания. ВИЧ-инфекция и иммуносупрессии. 2023; 15(3): 26–37. https://doi.org/10.22328/2077-9828-2023-15-3-26-37 https://elibrary.ru/cwjjai
- Frahm N., Korber B.T., Adams C.M., Szinger J.J., Draenert R., Addo M.M., et al. Consistent cytotoxic-T-lymphocyte targeting of immunodominant regions in human immunodeficiency virus across multiple ethnicities. J. Virol. 2004; 78(5): 2187–200. https://doi.org/10.1128/jvi.78.5.2187-2200.2004
- Mothe B., Hu X., Llano A., Rosati M., Olvera A., Kulkarni V., et al. A human immune data-informed vaccine concept elicits strong and broad T-cell specificities associated with HIV-1 control in mice and macaques. J. Transl. Med. 2015; 13: 60. https://doi.org/10.1186/s12967-015-0392-5
- Frahm N., Kiepiela P., Adams S., Linde C.H., Hewitt H.S., Sango K., et al. Control of human immunodeficiency virus replication by cytotoxic T lymphocytes targeting subdominant epitopes. Nat. Immunol. 2006; 7(2): 173–8. https://doi.org/10.1038/ni1281
- Sgadari C., Monini P., Tripiciano A., Picconi O., Casabianca A., Orlandi C., et al. Continued decay of HIV proviral DNA upon vaccination with HIV-1 Tat of subjects on long-term ART: An 8-year follow-up study. Front. Immunol. 2019; 10: 233. https://doi.org/10.3389/fimmu.2019.00233
- Kardani K., Hashemi A., Bolhassani A. Comparison of HIV-1 Vif and Vpu accessory proteins for delivery of polyepitope constructs harboring Nef, Gp160 and P24 using various cell penetrating peptides. PLoS One. 2019; 14(10): e0223844. https://doi.org/10.1371/journal.pone.0223844
- Kardani K., Hashemi A., Bolhassani A. Comparative analysis of two HIV-1 multiepitope polypeptides for stimulation of immune responses in BALB/c mice. Mol. Immunol. 2020; 119: 106–22. https://doi.org/10.1016/j.molimm.2020.01.013
- Sadeghi L., Bolhassani A., Mohit E., Baesi K., Aghasadeghi M.R. Heterologous DNA prime/protein boost immunization targeting Nef-Tat fusion antigen induces potent T-cell activity and in vitro anti-SCR HIV-1 effects. Curr. HIV Res. 2024; 22(2): 109–19. https://doi.org/10.2174/011570162X297602240430142231
- Лаповок И.А., Лопатухин А.Э., Киреев Д.Е., Казеннова Е.В., Лебедев А.В., Бобкова М.Р. и др. Молекулярно-эпидемиологический анализ вариантов ВИЧ-1, циркулировавших в России в 1987–2015 гг. Терапевтический архив. 2017; 89(11): 44–9. https://doi.org/10.17116/terarkh2017891144-49 https://elibrary.ru/zwosol
- Адгамов Р.Р., Антонова А.А., Огаркова Д.А., Кузнецова А.И., Почтовый А.А., Клейменов Д.А. и др. ВИЧ-инфекция в Российской Федерации: современные тенденции диагностики. ВИЧ-инфекция и иммуносупрессии. 2024; 16(1): 45–59. https://doi.org/10.22328/2077-9828-2024-16-1-45-59 https://elibrary.ru/rlhujr
- Антонова А.А., Кузнецова А.И., Ожмегова Е.Н., Лебедев А.В., Казеннова Е.В., Ким К.В. и др. Генетическое разнообразие ВИЧ-1 на современном этапе эпидемии в Российской Федерации: увеличение распространенности рекомбинантных форм. ВИЧ-инфекция и иммуносупрессии. 2023; 15(3): 61–72. https://doi.org/10.22328/2077-9828-2023-15-3-61-72 https://elibrary.ru/tpwttn
- Antonova A., Kazennova E., Lebedev A., Ozhmegova E., Kuznetsova A., Tumanov A., et al. Recombinant forms of HIV-1 in the last decade of the epidemic in the Russian Federation. Viruses. 2023; 15(12): 2312. https://doi.org/10.3390/v15122312
- Гришина Е.А. Особенности географического положения Российской Федерации. Теория и практика решения сложных заданий ГИА по географии. Вестник науки и образования. 2019; (6-1): 38–42. https://elibrary.ru/zbmuep
- Ившина И.Н. Многонациональность и многоконфессиональность как социально-культурные предпосылки Федерализации России. Вестник МГПУ. Серия: Юридические науки. 2018; (4): 19–24. https://doi.org/10.25688/2076-9113.2018.32.4.02 https://elibrary.ru/ypwurf
- Рубрикатор КР. Клинические рекомендации. ВИЧ-инфекция у взрослых. Available at: https://cr.minzdrav.gov.ru/schema/79_1
- Якубке Х.Д., Ешкайт Х. Аминокислоты. Пептиды. Белки. М.: Мир; 1985.
- Щемелев А.Н., Семенов А.В., Останкова Ю.В., Найденова Е.В., Зуева Е.Б., Валутите Д.Э. и др. Генетическое разнообразие вируса иммунодефицита человека (ВИЧ-1) в Калининградской области. Вопросы вирусологии. 2022; 67(4): 310–21. https://doi.org/10.36233/0507-4088-119 https://elibrary.ru/bkswno
- Lebedev A., Lebedeva N., Moskaleychik F., Pronin A., Kazennova E., Bobkova M. Human immunodeficiency virus-1 diversity in the Moscow Region, Russia: Phylodynamics of the most common subtypes. Front. Microbiol. 2019; 10: 320. https://doi.org/10.3389/fmicb.2019.00320
- Антонова А.А., Туманов А.С., Лебедев А.В., Казеннова Е.В., Глинкина Л.Н., Кулагин В.В. и др. Генетический профиль и характеристика мутаций лекарственной устойчивости ВИЧ-1 на территории Краснодарского края в период 2014–2019 гг. ВИЧ-инфекция и иммуносупрессии. 2022; 14(2): 20–30. https://doi.org/10.22328/2077-9828-2022-14-2-20-30 https://elibrary.ru/vzklej
- Kirichenko A., Kireev D., Lapovok I., Shlykova A., Lopatukhin A., Pokrovskaya A., et al. HIV-1 drug resistance among treatment-naïve patients in Russia: analysis of the national database, 2006–2022. Viruses. 2023; 15(4): 991. https://doi.org/10.3390/v15040991
- Kuznetsova A., Kim K., Tumanov A., Munchak I., Antonova A., Lebedev A., et al. Features of Tat protein in HIV-1 sub-subtype A6 variants circulating in the Moscow region, Russia. Viruses. 2023; 15(11): 2212. https://doi.org/10.3390/v15112212
- Кузнецова А.И., Громов К.Б., Киреев Д.Е., Шлыкова А.В., Лопатухин А.Э., Казеннова Е.В. и др. Анализ особенностей белка Tat вируса иммунодефицита человека 1 типа суб-субтипа А6 (Retroviridae: Orthoretrovirinae: Lentivirus: Human immunodeficiency virus-1). Вопросы вирусологии. 2021; 66(6): 452–63. https://doi.org/10.36233/0507-4088-83 https://elibrary.ru/cmzgyc
- Lebedev A., Kim K., Ozhmegova E., Antonova A., Kazennova E., Tumanov A., et al. Rev protein diversity in HIV-1 group M clades. Viruses. 2024; 16(5): 759. https://doi.org/10.3390/v16050759
- Антонова А.А., Лебедев А.В., Казеннова Е.В., Ким К.В., Ожмегова Е.Н., Туманов А.С. и др. Вариабельность белка VPU ВИЧ-1 суб-субтипа А6 у пациентов с различными стадиями ВИЧ-инфекции. ВИЧ-инфекция и иммуносупрессии. 2024; 16(2): 40–50. https://doi.org/10.22328/2077-9828-2024-16-2-40-50 https://elibrary.ru/lpjxqk
- Лаповок И.А. Анализ полиморфизма неструктурных областей генома варианта ВИЧ-1, доминирующего в России: Автореф. дисс. … канд. биол. наук. М.; 2009. https://elibrary.ru/nkranl
Дополнительные файлы