Генетическое разнообразие метапневмовируса человека (Pneumoviridae: Metapneumovirus) в России: результаты молекулярного анализа
- Авторы: Фадеев А.В.1, Иванов Я.В.1, Петрова П.А.1, Передерий А.А.1, Писарева М.М.1, Мошкин А.Д.2, Комиссаров А.Б.1, Даниленко Д.М.1, Лиознов Д.А.1
-
Учреждения:
- ФГБУ «НИИ гриппа им. А.А. Смородинцева» Минздрава России
- Научно-исследовательский институт вирусологии ФГБНУ «Федеральный исследовательский центр фундаментальной и трансляционной медицины»
- Выпуск: Том 70, № 2 (2025)
- Страницы: 164-176
- Раздел: ОРИГИНАЛЬНЫЕ ИССЛЕДОВАНИЯ
- URL: https://virusjour.crie.ru/jour/article/view/16718
- DOI: https://doi.org/10.36233/0507-4088-294
- EDN: https://elibrary.ru/vfpkwg
- ID: 16718
Цитировать
Аннотация
Актуальность. Метапневмовирус человека (Human metapneumovirus, hMPV) имеет большое эпидемиологическое значение, являясь доминирующей причиной инфекций нижних дыхательных путей у детей младше 2 лет и лиц старше 65 лет. Возможно многократное инфицирование hMPV в течение жизни человека ввиду антигенной и генетической изменчивости возбудителя. При этом генетическая изменчивость hMPV, циркулирующих в России, остается неизученной.
Цель работы. Апробация протокола полногеномного секвенирования hMPV для оценки генетического разнообразия метапневмовирусов, циркулирующих в отдельных субъектах России.
Материалы и методы. Исследовали назофарингеальные мазки от пациентов разного возраста с острыми респираторными вирусными инфекциями, положительные в полимеразной цепной реакции на hMPV. Из части образцов вирус выделяли на клеточной культуре. На платформе Illumina MiSeq проведено полногеномное секвенирование вирусов hMPV из мазков и изолятов с последующим филогенетическим анализом.
Результаты. Впервые в России проведено полногеномное секвенирование 44 hMPV, циркулировавших в период с 2017 по 2024 г., описана их принадлежность к генетическим группам, показано доминирование клайда A2b2. Подтверждено, что наибольшее разнообразие в генах, кодирующих поверхностные белки вируса, отмечено для гена G, в то время как в гене F в изучаемый период изменения были минимальны.
Заключение. Проведенное исследование дает представление о генетическом разнообразии вирусов hMPV, циркулирующих в отдельных субъектах Российской Федерации. Изучение генетической изменчивости hMPV имеет решающее значение для понимания вирусной эволюции, динамики передачи и механизмов иммунного ускользания, которые влияют на разработку вакцин и противовирусных препаратов.
Ключевые слова
Полный текст
Введение
Метапневмовирус человека (Orthornavirae: Pneumoviridae: Metapneumovirus: Metapneumovirus hominis (ICTV 2024), hMPV) впервые идентифицирован в Голландии в 2001 г. [1]. Геном вируса представлен одноцепочечной РНК с отрицательной полярностью. Ближайшим родственником метапневмовируса человека является респираторно-синцитиальный вирус человека (Orthopneumovirus hominis, hRSV), который также относится к семейству Pneumoviridae [2]. Оба вируса обладают рядом общих характеристик, таких как структура генома и пути передачи, однако различаются по специфическим особенностям взаимодействия с клетками хозяина и патогенетическим механизмам. Изучение эволюционных связей и генетических особенностей hMPV является важным для понимания механизмов патогенности и разработки эффективных методов профилактики и лечения.
Геном hMPV, длина которого составляет около 13 000 нуклеотидов, состоит из 8 генов, кодирующих 9 белков: нуклеопротеин (N), фосфопротеин (P), матричный белок (M), белок слияния (F), матричные белки 2 (M2-1 и M2-2), малый гидрофобный (SH) белок, гликопротеин (G) и большой (L) полимеразный белок [3, 4]. Среди них ключевую роль играют два поверхностных гликопротеина: белок слияния (F) и белок прикрепления (G) [4, 5]. Эти молекулы обеспечивают проникновение вируса в клетки хозяина и способствуют уклонению от иммунного ответа [6]. G-белок опосредует прикрепление вируса к клеточной поверхности, а F-белок отвечает за процессы слияния мембран, что делает эти белки важными мишенями для изучения и разработки терапевтических подходов [7]. Вирус обладает тропизмом к эпителиальным клеткам дыхательных путей, где вызывает выраженные цитопатические эффекты [8]. Одним из наиболее характерных проявлений патоморфологического процесса является формирование гигантских многоядерных эпителиальных клеток – синцития, что способствует распространению вируса в тканях и усиливает его патогенность [9].
Метапневмовирус человека классифицируется на две основные генетические линии – A и B, которые дополнительно подразделяются на соответствующие сублинии A1, A2, B1 и B2 [10, 11], учитывая генетическое разнообразие вируса [12]. Повторное заражение hMPV связано с антигенной изменчивостью поверхностных белков вируса, что затрудняет формирование стойкого иммунитета [12, 13]. Наиболее генетически близким пневмовирусом к hMPV является метапневмовирус птиц типа С. Согласно расчетам, ближайший общий предок этих вирусов мог существовать 215–268 лет назад [14, 15]. В настоящее время случаи зоонозной передачи метапневмовирусов неизвестны, а эксперименты по заражению птиц hMPV не приводили к продуктивной инфекции [16]. Предположительно, разделение типов метапневмовирусов человека могло произойти менее 100 лет назад [14].
В большинстве стран систематический надзор за этим вирусом отсутствовал, и на протяжении долгого времени молекулярная эпидемиология hMPV оставалась недостаточно изученной [6, 17]. Однако в последние годы в связи с возросшим интересом к изучению респираторно-синцитиального вируса увеличилось и количество исследований, посвященных метапневмовирусу1. В условиях глобализации и растущей мобильности населения антигенная и молекулярная характеристика современных изолятов hMPV приобрела критическое значение для понимания его глобального распространения и динамики эволюции [11, 13].
С эпидемиологической точки зрения hMPV является одной из ведущих причин острых респираторных инфекций негриппозной этиологии во всем мире [9, 18]. В первую очередь он поражает новорожденных и детей до 2 лет, людей старше 65 лет и пациентов с ослабленным иммунитетом, сезонные пики обычно приходятся на конец зимы и весну [10, 19]. Передача возбудителя происходит преимущественно воздушно-капельным путем, возможен контактно-бытовой путь, инкубационный период оценивается в 4–6 сутки [4, 12, 20–22]. Клинические проявления варьируют от легких симптомов поражения верхних дыхательных путей до тяжелого течения инфекции нижних дыхательных путей, включая бронхиолит и пневмонию [23, 24]. Эпидемиологические исследования показывают, что серопревалентность среди населения превышает 90% по достижении 5-летнего возраста, что предполагает почти всеобщее раннее инфицирование (как и для hRSV) [25].
В настоящее время в средствах массовой информации активно обсуждается рост заболеваемости метапневмовирусной инфекцией в северных провинциях Китайской Народной Республики и в ряде других стран1. Вместе с тем, учитывая ярко-выраженную сезонность циркуляции, необходимо отметить, что увеличение числа случаев метапневмовирусной инфекции является закономерным для данного времени года [4, 12, 20]. По официальной информации, размещенной на сайте Всемирной организации здравоохранения, «согласно последним данным эпиднадзора за острыми респираторными инфекциями, предоставленным Центром по контролю и профилактике заболеваний Китая по состоянию на 29 декабря 2024 года, наблюдается тенденция к росту распространенных острых респираторных инфекций, в том числе вызванных сезонными вирусами гриппа, RSV и hMPV, как и ожидалось для этого времени года (зимы) в Северном полушарии»1.
Несмотря на то что hMPV известен более 20 лет, его генетическое разнообразие охарактеризовано недостаточно как в мире, так и в России [1, 4, 24]. Помимо этого, несмотря на важную роль пневмовирусов в этиологической структуре респираторных инфекций, сведения о распространенности hMPV в разных регионах отрывочны, а характер его циркуляции практически не изучен [19, 26]. Таким образом, исследование закономерностей эволюции пневмовирусов, в частности hMPV, является необходимым и актуальным, учитывая сохраняющееся бремя пневмовирусных инфекций, особенно среди детей младших возрастных групп.
Цель работы заключалась в изучении генетического разнообразия метапневмовирусов человека, выявленных на территории России, на основе анализа генетических вариаций белков слияния (F) и прикрепления (G).
Материалы и методы
Клинические образцы. В исследование были включены образцы назофарингеальных мазков от госпитализированных больных с респираторными симптомами, собранные в период 2017–2024 гг. в основном в Санкт-Петербурге и Ленинградской области в рамках госпитального надзора за острыми респираторными вирусными инфекциями (ОРВИ). Экстракцию общей РНК проводили с использованием набора для выделения РНК NAmagP 2000 (ООО «Биолабмикс», Россия). Детекцию РНК метапневмовируса в клиническом материале осуществляли с помощью набора реагентов «АмплиСенс ОРВИ-скрин-FL» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия) методом полимеразной цепной реакции (ПЦР) с обратной транскрипцией в режиме реального времени.
Клеточные линии. Для выделения hMPV была использована клеточная линия LLC-MK2 (почка обезьяны Macaca mulatta), полученная из коллекции ATCC (American Type Culture Collection, США). Для культивирования монослоя клеток использовали питательную среду DMEM (ООО «Биолот», Россия) с добавлением 10% фетальной эмбриональной бычьей сыворотки (ООО «Биолот», Россия), 1% смеси антибиотиков пенициллина G и стрептомицина (50 000 ед/фл и 50 г/фл соответственно) (ООО «Биолот», Россия). Пересев клеточной культуры осуществляли на 7-е сутки. Посевная концентрация составляла 2,5–5,0 × 105 кл/мл. Клетки выращивали при температуре 37 °C в инкубаторе с 5% CO2.
Среды. Поддерживающая среда для вирусологических опытов на культуре клеток LLC-MK2. На 100 мл среды DMEM вносили 2,6 мл сывороточного альбумина V фракции (Sigma, Германия), 1,6 мл буфера HEPES (Sigma, Германия), 100 мкл раствора гентамицина для клеточных культур (ООО «Биолот», Россия), 100 мкл раствора TPCK-трипсина (2 мкг/мл) (Sigma, Германия). Среда для отмывания клеток. На 100 мл среды DМЕМ вносили 100 мкл раствора гентамицина для клеточных культур, 100 мкл раствора ТРСК – трипсина (2 мкг/мл).
Культивирование метапневмовируса. Суточный монослой клеток LLC-MK2, находящийся в специальных пластиковых пробирках со скошенным дном (Nunc, Дания), двукратно промывали средой для отмывания клеток по 2 мл. ПЦР-положительные материалы от больных вносили по 0,2 мл в 2 флакона с культурой клеток и после инкубации 40–60 мин при 36 °С добавляли 1,8 мл поддерживающей среды. После этого флаконы инкубировали при 36 °С, ежедневно контролируя состояние монослоя в инвертированный микроскоп. При отсутствии выраженного цитопатического действия (ЦПД) пробы выдерживали при 36 °С вплоть до 14 сут. При отсутствии ЦПД на 14-е сутки клетки разрушали замораживанием/оттаиванием, подготавливали объединенные пулы культуральной жидкости (из 2 флаконов от каждого образца) и проводили следующий пассаж в 2 флаконах с суточным монослоем клеток LLC-MK2 с регистрацией репродукции по ЦПД. Для каждой пробы проводили три последовательных пассажа. При наличии начального или выраженного ЦПД присутствие вируса дополнительно подтверждали с помощью ПЦР-тест-системы с детекцией результатов в реальном времени. Серия праймеров и условия проведения реакции описаны в работе S. Sugimoto и соавт. [27].
Полногеномную амплификацию проводили по протоколу K. Groen и соавт. [28] с модификациями. Секвенирование полноразмерного генома hMPV осуществляли с помощью набора реагентов DNA Prep компании Illumina (США). Очистку комплементарной ДНК производили на магнитных частицах. Геномные библиотеки секвенировали на платформе MiSeq Illumina с использованием MiSeq Reagent Kit v3 600-cycle. Для выравнивания полученных прочтений на референсную последовательность использовали алгоритм BWA. Для получения консенсусных последовательностей применяли инструменты Samtools и Ivar.
Сравнительный филогенетический анализ нуклеотидных последовательностей проводили методом максимального правдоподобия (maximum likelihood) с использованием алгоритма RAxML и модели нуклеотидных замен GTRGAMMA. Статистическую поддержку ветвей дерева определяли методом быстрого бутстрепа (rapid bootstrap) c 1000 репликаций. Филогенетическое дерево укореняли на среднюю точку (midpoint rooting). Реконструкцию предковых последовательностей проводили с использованием алгоритма PAML в Treesub2. Визуализацию и аннотацию филогенетических деревьев проводили с использованием инструментов Figtree и Inkscape [28]. Для визуализации полногеномного филогенетического дерева использовали библиотеку ggtree в R. Для анализа был использован глобальный датасет полных геномов метапневмовирусов человека, доступный на платформе Nextstrain (https://nextstrain.org/hmpv) – 682 генома, депонированных в базе данных GenBank. Фильтрацию набора данных осуществляли с помощью инструмента Nextclade [31]. В качестве критерия использовали количество несеквенированных позиций (totalMissing < 1500) и показатель качества по наличию уникальных мутаций (qc.privateMutations.status не равно «bad»). Определение филогенетического сигнала в полногеномных последовательностях hMPV осуществляли с помощью программы TempEst. Анализ внешнего домена белка G hMPV проводили инструментом Chi-Score.
Этическое утверждение. Исследование проводилось при добровольном информированном согласии пациентов. Протокол исследования одобрен Этическим комитетом ФГБУ «НИИ гриппа им. А.А. Смородинцева» (протоколы № 215 от 31.01.2024, № 194 от 12.12.2022, № 178 от 10.01.2022, № 161 от 14.12.2020, № 149 от 18.12.2019, № 136 от 21.12.2018, № 3120 от 18.12.2017).
Результаты
По данным ПЦР-диагностики, в течение эпидемических сезонов 2016–2024 гг. циркуляция метапневмовируса в Санкт-Петербурге и Ленинградской области была нестабильной и низкой, составляя в среднем 9,7% от всех положительных на вирусные патогены случаев, исключая возбудители гриппа и COVID-19 (рис. 1). Доля положительных случаев метапневмовирусной инфекции была относительно высокой только в сезон 2020–2021 гг. и составила 21,4%. В следующем сезоне 2021–2022 гг. вклад hMPV был минимальным (2,5%), для сравнения — доля лабораторно подтвержденной риновирусной инфекции в этот сезон составила 49,2%.
Рис. 1. Доля положительных на метапневмовирус образцов в структуре ОРВИ по сезонам 2016–2024 гг.
* – образцы положительны только на респираторные вирусы (рино-, адено-, бока-, метапневмо-, коронавирусы, вирусы парагриппа и респираторно-синцитиальный вирус человека) исключая вирусы гриппа и SARS-CoV-2.
Fig. 1. The percentage of samples positive for metapneumovirus in the structure of acute respiratory viral infections by seasons 2016–2024.
* – samples are positive only for respiratory viruses (rhino-, adeno-, boca-, metapneumo-, coronavirus, parainfluenza viruses and human respiratory syncytial virus) excluding influenza viruses and SARS-CoV-2.
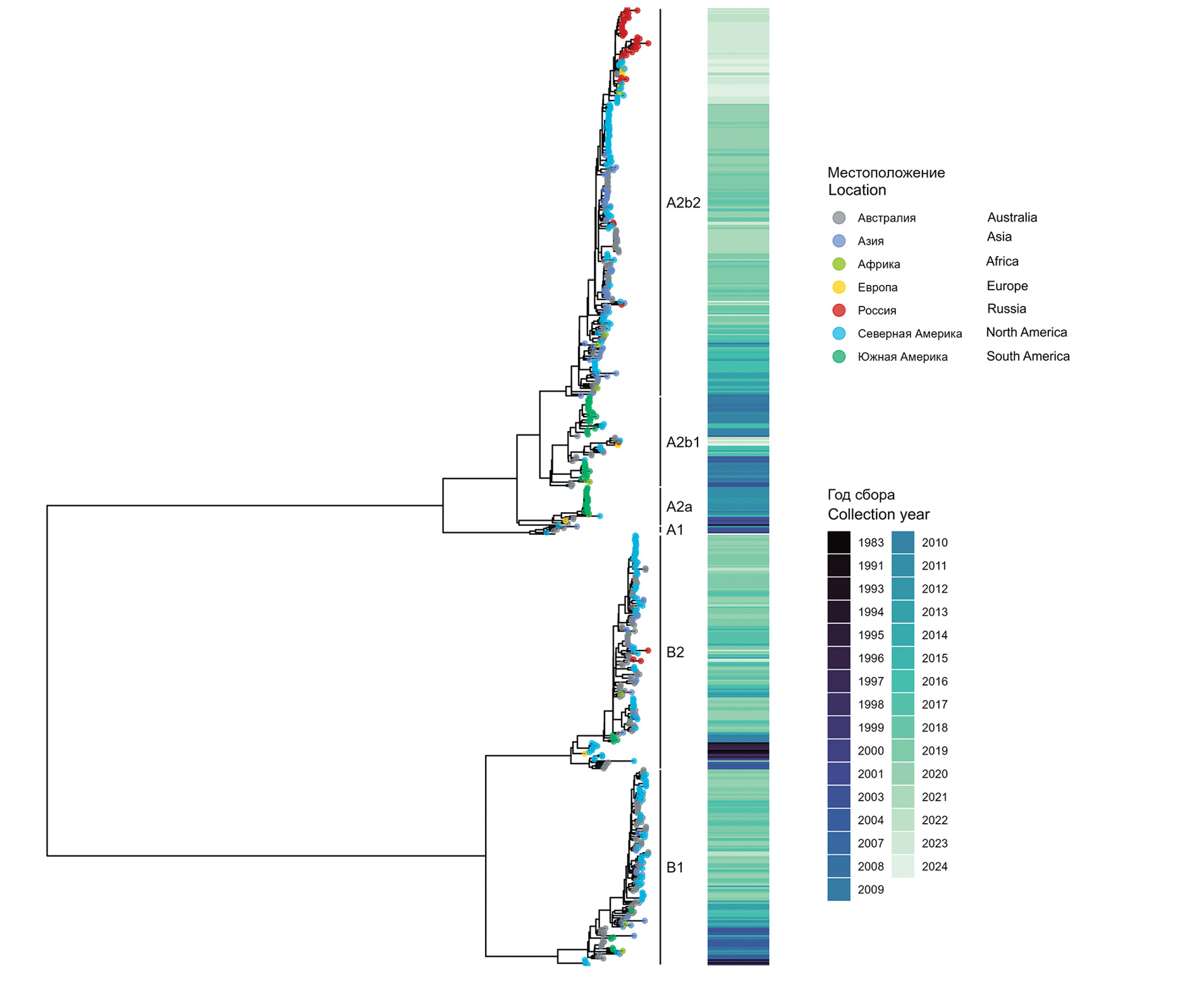
В результате проведенного секвенирования были получены 44 полногеномные последовательности метапневмовирусов, выделенных от больных из Санкт-Петербурга и Ленинградской области (43) и Ямало-Ненецкого автономного округа (1) в период 4 эпидемических сезонов 2018–2024 гг. Из них 11 циркулировали до пандемии (эпидемические сезоны 2017–2018 и 2018–2019 гг.), и 33 – в эпидемические сезоны 2022–2023 и 2023–2024 гг. Несмотря на высокую долю hMPV в циркуляции в сезоне 2020–2021 гг., полногеномных последовательностей вируса из первичных образцов получить не удалось. Вирусовыделение также оказалось неудачным, что может быть связано со средой для забора образцов на пике пандемии, не предназначенной для выделения вирусов. По данным филогенетического анализа, большинство секвенированных метапневмовирусов относилось к генетическому клайду A2b2 (40 из 44), 1 вирус был идентифицирован как принадлежащий к клайду A2b1 и 3 – к клайду B2 (рис. 2, табл. 1). По результатам анализа глобального набора данных, доля клайда A2b2 составляла в разные годы от 27 до 100%.
Рис. 2. Глобальное разнообразие метапневмовирусов человека по данным полногеномного секвенирования. Красным отмечены изоляты из Российской Федерации.
Fig. 2. Global diversity of human metapneumoviruses based on whole-genome sequencing data. Isolates from the Russian Federation are marked in red.
Таблица 1. Принадлежность метапневмовирусов человека к генетическим линиям в эпидемические сезоны 2017–2024 гг.
Table 1. Genetic lineages of human metapneumoviruses in the epidemic seasons of 2017–2024
Эпидемический сезон Epidemic season | Всего Total | A2b1 | A2b2 | B2 |
2017–2018 | 8 | 0 | 6 | 2 |
2018–2019 | 3 | 0 | 3 | 0 |
2022–2023 | 10 | 0 | 10 | 0 |
2023–2024 | 23 | 1 | 21 | 1 |
Вирусы hMPV генетической линии A2b2 из отдельных субъектов России по данным полногеномного анализа формировали три филогенетических кластера, близких вирусам из Швейцарии, США и Австралии 2021–2024 гг. Интересно отметить, что в каждый из кластеров входили вирусы, собранные в разные эпидемические сезоны. Это может объясняться слабым временны́м сигналом (temporal signal [30]) или невысокой скоростью эволюции данного патогена в сочетании с глобальным распространением и многочисленными переносами вируса из одних географических регионов в другие и обратно. Для проверки уровня временно́го сигнала в полногеномных последовательностях hMPV использовали программу TempEst.
Проведенный анализ продемонстрировал значительную корреляцию (r > 0,9, R2 > 0,8) между генетической дистанцией и временем сбора образцов, что свидетельствует о наличии временно́го сигнала в данных hMPV разных субтипов (рис. 3). Таким образом, объединение геномов hMPV 2018–2024 гг. в кластеры может свидетельствовать о том, что за период 5–6 лет в геноме не происходит достаточных эволюционных изменений для надежной дифференциации вирусов разных эпидемических сезонов. При этом на более длительных временны́х промежутках такая дифференциация становится возможной (рис. 2), что видно по тому, как группируются вирусы разных лет на цветовой шкале.
Рис. 3. Определение временно́го сигнала в полногеномных последовательностях hMPV. Субтип A2 (слева), субтип B2 (справа). D – генетическая дистанция от корня до листьев филогенетического дерева (root-to-tip divergence).
Fig. 3. Temporal signal in whole genome sequences of hMPV. Subtype A2 (left), subtype B2 (right). D – genetic distance of root-to-tip divergence.
По результатам анализа генов F и G показаны минимальные генетические изменения в гене F и значительная генетическая вариабельность гликопротеина G (рис. 4–7). Метапневмовирусы клайда А2b2, собранные в 2017–2024 гг., за исключением двух вирусов, образуют единую группу на филогенетическом дереве (рис. 4). Интересно отметить, что генетическая линия A2b2 не имеет характерных отличительных аминокислотных замен в F.
Рис. 4. Филогенетическое дерево по гену F метапневмовируса человека подтипа А2. Красным цветом отмечены вирусы, выявленные в трех регионах Российской Федерации.
Fig. 4. F gene phylogenetic tree of subtype A2 human metapneumovirus. Viruses identified in three regions of the Russian Federation are marked in red.
Рис. 5. Филогенетическое дерево по гену G метапневмовируса человека подтипа А2. Красным цветом отмечены вирусы, выявленные в трех регионах Российской Федерации.
Fig. 5. G gene phylogenetic tree of subtype A2 human metapneumovirus. Viruses identified in three regions of the Russian Federation are marked in red.
Рис. 6. Филогенетическое дерево по гену F метапневмовируса человека подтипа B2. Красным цветом отмечены вирусы, выявленные в Санкт-Петербурге.
Fig. 6. F gene phylogenetic tree of subtype B2 human metapneumovirus. Viruses identified in Saint Petersburg are marked in red
Рис. 7. Филогенетическое дерево по гену G метапневмовируса человека подтипа B2. Красным цветом отмечены вирусы, выявленные в Санкт-Петербурге.
Fig. 7. G gene phylogenetic tree of subtype B2 human metapneumovirus. Viruses identified in Saint Petersburg are marked in red.
Большинство обнаруженных аминокислотных полиморфизмов локализовались в субъединице F1 белка слияния F. Аминокислотных замен, приводящих к изменению профиля N-гликозилирования, в белке F обоих подтипов hMPV не выявлено (рис. 4, 6).
Ген G является высоковариабельным и кодирует муцин-подобный гликопротеин неупорядоченной структуры с большим количеством потенциальных сайтов О-гликозилирования. Кодирующую последовательность гена G можно разделить на интравирионный (1–30 aa), трансмембранный (31–51 aa) и внешний домены. Интравирионный и трансмембранный домены hMPV разных субтипов имеют высокую степень сходства: 36 аминокислотных остатков (а.о.) из 51 (более 70%), консервативны у 99–100% вирусов субтипов A2 и B2. Для внешнего домена этот показатель снижается до уровня шума (менее 30%). Интересно отметить, что для hMPV субтипа A2 характерно возникновение протяженных дупликаций: так, в гене G 36 из 37 метапневмовирусов субтипа A2 из всех трех регионов России выявлен участок с дупликацией длиной 111 нуклеотидов. В гене G субтипа B2 протяженные дупликации выявлены не были.
Показано, что неупорядоченные участки белков могут иметь неслучайную организацию и возможно их разделение на модули с помощью теста χ2 на гомогенность [31]. С помощью инструмента Chi-Score нами был проведен анализ внешнего домена белка G hMPV подтипов A2 и B2 (рис. 8).
Рис. 8. Тепловая карта внешних доменов белка G вирусов hMPV подтипов A2 (слева) и B2 (справа) по данным инструмента Chi-Score (Brendan S. и соавт., 2023 [31]). Красными стрелками отмечены участки протяженных дупликаций.
Fig. 8. Heat map of G protein external domains of hMPV subtypes A2 (left) and B2 (right) based on Chi-Score data (Brendan S, et al., 2023 [31]). Red arrows indicate regions of extended duplications.
Было показано, что внешний домен белка G hMPV подтипа A2 может быть разделен на два модуля – модуль 1 (52–139 а.о.) и модуль 2 (140 а.о. – C-конец белка), причем для модуля 2 характерны протяженные дупликации (длиной до 37 а.о.). Повторы состоят из участков S1, L1, S2, L2, разделенных консервативными спейсерными последовательностями RTSSA (рис. 9). Следует отметить, что повторяющиеся участки не на 100% гомологичны и могут содержать замены в разных положениях.
Рис. 9. Структура последовательности с повторами внешнего домена белка G hMPV подтипа A2.
Fig. 9. Structure of the duplication sequences in the G protein external domain of the hMPV subtype A2.
Около 57% метапневмовирусов субтипа A2, взятых в анализ, содержали только участки S1 и L2 (в том числе 1 вирус линии A2b1 (hMPV/Russia/SPE-RII-7947S/2023), 41% – дополнительно содержали участок L1 (в том числе 36 российских вирусов линии A2b2) и лишь менее 2% вирусов содержали все повторы (табл. 2).
Таблица 2. Различные комбинации повторов последовательностей внешнего домена белка G подтипа A2 у исследованных вирусов hMPV
Table 2. Different combinations of duplications in the G protein external domain of the studied hMPV subtype A2
Наличие повторов Duplications | Количество изолятов, содержащих данную комбинацию/N секвенированных Number of isolates containing this combination/Number of sequenced isolates | Примечание Note | |||
S1 | L1 | S2 | L2 | 6/338 | Нет российских изолятов No isolates from Russia |
S1 | L1 | – | L2 | 140/338 | 36 российских изолятов линии A2b2 36 subtype A2b2 isolates from Russia |
S1 | – | – | L2 | 192/338 | 1 российский изолят линии A2b1 1 subtype A2b1 isolate from Russia |
Внешний домен белка G hMPV субтипа B2 может быть разделен на четыре модуля – модуль 1, модули 2 и 4, являющиеся муцин-подобными доменами, и модуль 3 между ними, представляющий собой Glu-Lys-богатый спейсер 14–18 а.о.
Обсуждение
Метапневмовирус человека представляет собой один из респираторных патогенов, которые оказывают значительное влияние на здоровье, прежде всего детей и пожилых людей. Проведенное исследование позволило выявить важные аспекты циркуляции и генетической изменчивости hMPV в трех регионах Российской Федерации, что способствует пониманию динамики вирусной эволюции.
Одним из ключевых результатов работы стало установление доминирования генетического клайда A2b2 в российских изолятах hMPV в период с 2017 по 2024 г.
Проведенный филогенетический анализ показал высокую степень сходства между российскими изолятами hMPV и изолятами, циркулирующими в Европе и Азии, что подчеркивает важность трансграничного эпидемиологического мониторинга.
Сравнение результатов филогенетического анализа по полным геномам и по кодирующим поверхностные антигенам последовательностям показывает одинаковое разделение на клайды для всех исследованных вирусов, что позволяет предполагать низкую вероятность рекомбинации между разными штаммами hMPV.
В гене G 84% российских изолятов, относящихся к линии A2b, были выявлены дупликации размером 54 и 111 нуклеотидов. В исследовании P. Parida и соавт. обнаружена 180-нуклеотидная дупликация в гене G hMPV линии A2c [32]. Аналогичные результаты были получены в более ранних исследованиях в Испании между 2014 и 2016 гг. [33], в Хорватии между 2014 и 2017 гг. [34] и в Японии в 2014 г. [35]. Также в тот же период времени более 50% изолятов hMPV имели дупликации 111 нуклеотидов (Япония, 2014–2016 гг. [36], Хорватия, 2014–2017 гг. [34], Китай, 2017 г. [37]). В исследовании, проведенном M. Pinana и соавт. в Испании, было выдвинуто предположение, что сублиния A2c может заменить сублинии A2a и A2b, а штаммы A2c с дупликациями могут вскоре заменить дикий тип A2c из-за лучшего механизма уклонения от иммунного ответа, возникающего в результате дупликации [33]. Наличие дупликаций в гене G у большинства российских изолятов указывает на возможное формирование новых антигенных вариантов, которые могут иметь эпидемиологическое значение. Эти изменения требуют дальнейшего изучения, поскольку они потенциально могут повлиять на эффективность вакцинных препаратов.
Заключение
В работе изучена молекулярная структура метапневмовируса человека, циркулировавшего в 2017–2024 гг. в отдельных субъектах России, что позволило получить уникальные данные о его генетическом разнообразии. Результаты исследования подчеркивают значимость комплексного подхода к изучению вируса, включающего мониторинг циркуляции, определение генетической изменчивости и оценку эволюции вирусов. Проведенное исследование закладывает фундамент для дальнейшего анализа эволюционных процессов вируса hMPV и оценки его влияния на здоровье населения, что имеет практическое и научное значение для совершенствования методов молекулярной диагностики, оценки риска распространения новых генотипов, эпидемиологического мониторинга, стратегий контроля за острыми респираторными инфекциями и создания вакцин.
1 World Health Organization: Disease Outbreak News; Trends of acute respiratory infection, including human metapneumovirus, in the Northern Hemisphere 2025 [7 January 2025]. Доступно по: https://www.who.int/emergencies/disease-outbreak-news/item/2025-DON550
2 Treesub: annotating ancestral substitutions on a tree [1 February 2025]. Доступно по: https://github.com/tamuri/treesub.
Об авторах
Артём Викторович Фадеев
ФГБУ «НИИ гриппа им. А.А. Смородинцева» Минздрава России
Email: artem.fadeev@influenza.spb.ru
ORCID iD: 0000-0003-3558-3261
старший научный сотрудник лаборатории молекулярной вирусологии
Россия, 197376, г. Санкт-ПетербургЯн Владимирович Иванов
ФГБУ «НИИ гриппа им. А.А. Смородинцева» Минздрава России
Email: ivanov.yan.vladimirovich@yandex.ru
ORCID iD: 0009-0006-0187-4000
аспирант 2-го года обучения лаборатории молекулярной вирусологии ФГБУ
Россия, 197376, г. Санкт-ПетербургПолина Александровна Петрова
ФГБУ «НИИ гриппа им. А.А. Смородинцева» Минздрава России
Email: suddenkovapolina@gmail.com
ORCID iD: 0000-0001-8527-7946
научный сотрудник лаборатории эволюционной изменчивости вирусов гриппа
Россия, 197376, г. Санкт-ПетербургАлександр Александрович Передерий
ФГБУ «НИИ гриппа им. А.А. Смородинцева» Минздрава России
Email: gilagalex@gmail.com
ORCID iD: 0000-0002-5961-1856
лаборант-исследователь лаборатории молекулярной вирусологии
Россия, 197376, г. Санкт-ПетербургМария Михайловна Писарева
ФГБУ «НИИ гриппа им. А.А. Смородинцева» Минздрава России
Email: maria.pisareva@influenza.spb.ru
ORCID iD: 0000-0002-1499-9957
канд. биол. наук, ведущий научный сотрудник лаборатории молекулярной вирусологии
Россия, 197376, г. Санкт-ПетербургАлексей Дмитриевич Мошкин
Научно-исследовательский институт вирусологии ФГБНУ «Федеральный исследовательский центр фундаментальной и трансляционной медицины»
Email: alex.moshkin727@gmail.com
ORCID iD: 0000-0002-1182-8247
младший научный сотрудник
Россия, 630060, г. НовосибирскАндрей Борисович Комиссаров
ФГБУ «НИИ гриппа им. А.А. Смородинцева» Минздрава России
Автор, ответственный за переписку.
Email: andrey.komissarov@influenza.spb.ru
ORCID iD: 0000-0003-1733-1255
заведующий лабораторией молекулярной вирусологии
Россия, 197376, г. Санкт-ПетербургДарья Михайловна Даниленко
ФГБУ «НИИ гриппа им. А.А. Смородинцева» Минздрава России
Email: daria.danilenko@influenza.spb.ru
ORCID iD: 0000-0001-6174-0836
канд. биол. наук, заместитель директора по научной работе
Россия, 197376, г. Санкт-ПетербургДмитрий Анатольевич Лиознов
ФГБУ «НИИ гриппа им. А.А. Смородинцева» Минздрава России
Email: dmitry.lioznov@influenza.spb.ru
ORCID iD: 0000-0003-3643-7354
д-р мед. наук, профессор, директор
Россия, 197376, г. Санкт-ПетербургСписок литературы
- Uddin S., Thomas M. Human Metapneumovirus. StatPearls. StatPearls Publishing LLC; 2025.
- Sanz-Munoz I., Sanchez-de Prada L., Castrodeza-Sanz J., Eiros J.M. Microbiological and epidemiological features of respiratory syncytial virus. Rev. Esp. Quimioter. 2024; 37(3): 209–20. https://doi.org/10.37201/req/006.2024
- Cifuentes-Munoz N., Branttie J., Slaughter K.B., Dutch R.E. Human metapneumovirus induces formation of inclusion bodies for efficient genome replication and transcription. J. Virol. 2017; 91(24): e01282-17. https://doi.org/10.1128/JVI.01282-17
- Shafagati N., Williams J. Human metapneumovirus – what we know now. F1000Res. 2018; 7: 135. https://doi.org/10.12688/f1000research.12625.1
- Leyrat C., Paesen G.C., Charleston J., Renner M., Grimes J.M. Structural insights into the human metapneumovirus glycoprotein ectodomain. J. Virol. 2014; 88(19): 11611–6. https://doi.org/10.1128/JVI.01726-14
- Jesse S.T., Ludlow M., Osterhaus A.D.M.E. Zoonotic origins of human metapneumovirus: a journey from birds to humans. Viruses. 2022; 14(4): 677. https://doi.org/10.3390/v14040677
- Chang A., Masante C., Buchholz U.J., Dutch R.E. Human metapneumovirus (HMPV) binding and infection are mediated by interactions between the HMPV fusion protein and heparan sulfate. J. Virol. 2012; 86(6): 3230–43. https://doi.org/10.1128/JVI.06706-11
- Ribo-Molina P., van Nieuwkoop S., Mykytyn A.Z., van Run P., Lamers M.M., Haagmans B.L., et al. Human metapneumovirus infection of organoid-derived human bronchial epithelium represents cell tropism and cytopathology as observed in in vivo models. mSphere. 2024; 9(2): e0074323. https://doi.org/10.1128/msphere.00743-23
- Kinder J.T., Moncman C.L., Barrett C., Jin H., Kallewaard N., Dutch R.E. Respiratory syncytial virus and human metapneumovirus infections in three-dimensional human airway tissues expose an interesting dichotomy in viral replication, spread, and inhibition by neutralizing antibodies. J. Virol. 2020; 94(20): e01068-20. https://doi.org/10.1128/JVI.01068-20
- Herfst S., de Graaf M., Schickli J.H., Tang R.S., Kaur J., Yang C.F., et al. Recovery of human metapneumovirus genetic lineages a and B from cloned cDNA. J. Virol. 2004; 78(15): 8264–70. https://doi.org/10.1128/JVI.78.15.8264-8270.2004
- Groen K., van Nieuwkoop S., Meijer A., van der Veer B., van Kampen J.J.A., Fraaij P.L., et al. Emergence and Potential extinction of genetic lineages of human metapneumovirus between 2005 and 2021. mBio. 2023; 14(1): e0228022. https://doi.org/10.1128/mbio.02280-22
- Kahn J.S. Epidemiology of human metapneumovirus. Clin. Microbiol. Rev. 2006; 19(3): 546–57. https://doi.org/10.1128/CMR.00014-06
- van den Hoogen B.G., Herfst S., Sprong L., Cane P.A., Forleo-Neto E., de Swart R.L., et al. Antigenic and genetic variability of human metapneumoviruses. Emerg. Infect. Dis. 2004; 10(4): 658–66. https://doi.org/10.3201/eid1004.030393
- Yang C.F., Wang C.K., Tollefson S.J., Piyaratna R., Lintao L.D., Chu M., et al. Genetic diversity and evolution of human metapneumovirus fusion protein over twenty years. Virol. J. 2009; 6: 138. https://doi.org/10.1186/1743-422X-6-138
- Yang C.F., Wang C.K., Tollefson S.J., Lintao L.D., Liem A., Chu M., et al. Human metapneumovirus G protein is highly conserved within but not between genetic lineages. Arch. Virol. 2013; 158(6): 1245–52. https://doi.org/10.1007/s00705-013-1622-x
- van den Hoogen B.G., de Jong J.C., Groen J., Kuiken T., de Groot R., Fouchier R.A., et al. A newly discovered human pneumovirus isolated from young children with respiratory tract disease. Nat. Med. 2001; 7(6): 719–24. https://doi.org/10.1038/89098
- Feng Y., He T., Zhang B., Yuan H., Zhou Y. Epidemiology and diagnosis technologies of human metapneumovirus in China: a mini review. Virol. J. 2024; 21(1): 59. https://doi.org/10.1186/s12985-024-02327-9
- Esposito S., Mastrolia M.V. Metapneumovirus infections and respiratory complications. Semin. Respir. Crit. Care Med. 2016; 37(4): 512–21. https://doi.org/10.1055/s-0036-1584800
- Hamelin M.E., Abed Y., Boivin G. Human metapneumovirus: a new player among respiratory viruses. Clin. Infect. Dis. 2004; 38(7): 983–90. https://doi.org/10.1086/382536
- Yi L., Zou L., Peng J., Yu J., Song Y., Liang L., et al. Epidemiology, evolution and transmission of human metapneumovirus in Guangzhou China, 2013–2017. Sci. Rep. 2019; 9(1): 14022. https://doi.org/10.1038/s41598-019-50340-8
- Matsuzaki Y., Itagaki T., Ikeda T., Aoki Y., Abiko C., Mizuta K. Human metapneumovirus infection among family members. Epidemiol. Infect. 2013; 141(4): 827–32. https://doi.org/10.1017/S095026881200129X
- Hacker K., Kuan G., Vydiswaran N., Chowell-Puente G., Patel M., Sanchez N., et al. Pediatric burden and seasonality of human metapneumovirus over 5 years in Managua, Nicaragua. Influenza Other Respir. Viruses. 2022; 16(6): 1112–21. https://doi.org/10.1111/irv.13034
- Howard L.M., Edwards K.M., Zhu Y., Griffin M.R., Weinberg G.A., Szilagyi P.G., et al. Clinical features of human metapneumovirus infection in ambulatory children aged 5-13 years. J. Pediatric Infect. Dis. Soc. 2018; 7(2): 165–8. https://doi.org/10.1093/jpids/pix012
- Шарипова Е.В., Бабаченко И.В., Орлова Е.Д. Метапневмовирусная инфекция у детей. Педиатр. 2020; 11(5): 13–9. https://doi.org/10.17816/PED11513-19 https://elibrary.ru/xqnxgf
- Lu G., Gonzalez R., Guo L., Wu C., Wu J., Vernet G., et al. Large-scale seroprevalence analysis of human metapneumovirus and human respiratory syncytial virus infections in Beijing, China. Virol. J. 2011; 8: 62. https://doi.org/10.1186/1743-422X-8-62
- Яцышина СБ. Пневмовирусы в инфекционной патологии человека. Журнал микробиологии, эпидемиологии и иммунобиологии. 2017; 94(6): 95–105. https://doi.org/10.36233/0372-9311-2017-6-95-105 https://elibrary.ru/zaddtv
- Sugimoto S., Kawase M., Suwa R., Kakizaki M., Kume Y., Chishiki M., et al. Development of a duplex real-time RT-PCR assay for the detection and identification of two subgroups of human metapneumovirus in a single tube. J. Virol. Methods. 2023; 322: 114812. https://doi.org/10.1016/j.jviromet.2023.114812
- Groen K., van Nieuwkoop S., Bestebroer T.M., Fraaij P.L., Fouchier R.A.M., van den Hoogen B.G. Whole genome sequencing of human metapneumoviruses from clinical specimens using MinION nanopore technology. Virus Res. 2021; 302: 198490. https://doi.org/10.1016/j.virusres.2021.198490
- Aksamentov I., Roemer C., Hodcroft E., Neher R. Nextclade: clade assignment, mutation calling and quality control for viral genomes. J. Open Source Softw. 2021; 6(67): 3773. https://doi.org/10.21105/joss.03773
- Rambaut A., Lam T.T., Max Carvalho L., Pybus O.G. Exploring the temporal structure of heterochronous sequences using TempEst (formerly Path-O-Gen). Virus Evol. 2016; 2(1): vew007. https://doi.org/10.1093/ve/vew007
- McConnell B.S., Parker M.W. Protein intrinsically disordered regions have a non-random, modular architecture. Bioinformatics. 2023; 39(12): btad732. https://doi.org/10.1093/bioinformatics/btad732
- Parida P., Sudheesh N., Sanjay E.R., Jagadesh A., Marate S., Govindakaranavar A. The emergence of human metapneumovirus G gene duplication in hospitalized patients with respiratory tract infection, India, 2016–2018. Mol. Biol. Rep. 2023; 50(2): 1109–16. https://doi.org/10.1007/s11033-022-08092-8
- Piñana M., Vila J., Maldonado C., Galano-Frutos J.J., Valls M., Sancho J., et al. Insights into immune evasion of human metapneumovirus: novel 180- and 111-nucleotide duplications within viral G gene throughout 2014–2017 seasons in Barcelona, Spain. J. Clin. Virol. 2020; 132: 104590. https://doi.org/10.1016/j.jcv.2020.104590
- Jagusic M., Slovic A., Ivancic-Jelecki J., Ljubin-Sternak S., Vilibić-Čavlek T., Tabain I., et al. Molecular epidemiology of human respiratory syncytial virus and human metapneumovirus in hospitalized children with acute respiratory infections in Croatia, 2014–2017. Infect. Genet. Evol. 2019; 76: 104039. https://doi.org/10.1016/j.meegid.2019.104039
- Saikusa M., Kawakami C., Nao N., Takeda M., Usuku S., Sasao T., et al. 180-nucleotide duplication in the G gene of human metapneumovirus A2b subgroup strains circulating in Yokohama city, Japan, since 2014. Front. Microbiol. 2017; 8: 402. https://doi.org/10.3389/fmicb.2017.00402
- Saikusa M., Nao N., Kawakami C., Usuku S., Sasao T., Toyozawa T., et al. A novel 111-nucleotide duplication in the G gene of human metapneumovirus. Microbiol. Immunol. 2017; 61(11): 507–12. https://doi.org/10.1111/1348-0421.12543
- Yi L., Zou L., Peng J., Yu J., Song Y., Liang L., et al. Epidemiology, evolution and transmission of human metapneumovirus in Guangzhou China, 2013–2017. Sci. Rep. 2019; 9(1): 14022. https://doi.org/10.1038/s41598-019-50340-8
Дополнительные файлы