Идентификация нового альфакоронавируса (Coronaviridae: Alphacoronavirus), ассоциированного с большим подковоносом (Rhinolophus ferrumequinum), на юге европейской части России
- Авторы: Леншин С.В.1, Вишневская Т.В.2, Ромашин А.В.3, Булычева Ю.И.2, Вышемирский О.И.4, Соловьева С.А.2, Гительман А.К.2, Пазилин А.С.2, Львов Д.К.2, Ху Б.5, Ши Ч.5,6, Альховский С.В.2
-
Учреждения:
- ФКУЗ «Ставропольский противочумный институт» Роспотребнадзора
- Институт вирусологии имени Д.И. Ивановского ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
- ФГБУ «Сочинский национальный парк» Минприроды России
- НИЦ «Курчатовский институт»
- Уханьский институт вирусологии Китайской академии наук
- Национальная лаборатория Гуанчжоу
- Выпуск: Том 69, № 6 (2024)
- Страницы: 546-557
- Раздел: ОРИГИНАЛЬНЫЕ ИССЛЕДОВАНИЯ
- URL: https://virusjour.crie.ru/jour/article/view/16702
- DOI: https://doi.org/10.36233/0507-4088-279
- EDN: https://elibrary.ru/yiclrv
- ID: 16702
Цитировать
Аннотация
Введение. Летучие мыши рассматриваются как основной природный резервуар для альфа- и бетакоронавирусов. В результате преодоления межвидового барьера коронавирусы летучих мышей передаются другим видам млекопитающих, включая сельскохозяйственных животных и человека, что часто приводит к возникновению эпидемических или эпизоотических вспышек и пандемий.
Цель работы. Идентифицировать зоонозные коронавирусы, циркулирующие в популяции подковоносых летучих мышей (Rhinolophus spp.) в южных регионах европейской части России. Определить их генетические и экологические характеристики.
Материалы и методы. Материал для исследования (фекалии летучих мышей) был собран в пещерах на Южном макросклоне большого Кавказа (район Сочи-Адлер) в 2020, 2021 и 2024 гг. Использованы методы метагеномного анализа на основе NGS и ОТ-ПЦР.
Результаты. Идентифицирован новый альфакоронавирус (вирус Кудеп, GenBank № PQ649435), ассоциированный с большим подковоносом (R. ferrumequinum). Вирус Кудеп предположительно представляет новый вид подрода Decacovirus рода Alphacoronavirus. Максимальное генетическое сходство вирус Кудеп имеет с коронавирусом летучих мышей (от ложного вампира Cardioderma cor) из Кении (72% идентичных н.о.) и с группой вирусов YN2012, найденных у подковоносых летучих мышей в Китае (до 67% н.о.). Результаты ОТ-ПЦР-скрининга показывают, что вирус Кудеп и ранее описанный нами SARS-подобный бетакоронавирус Хоста-1 активно циркулируют на обследованной территории. Зараженность этими вирусами в одной из колоний большого подковоноса осенью 2021 г. достигала 59,2 и 70,5% соответственно. Выявлены частые случаи коинфекции отдельных особей одновременно двумя коронавирусами.
Заключение. Полученные данные расширяют представления о распространении альфакоронавиурусов летучих мышей и их генетическом разнообразии. Показано наличие стойкого природного очага потенциально зоонозных коронавирусов (Хоста-1 и Кудеп), связанных с R. ferrumequinum, на юге европейской части России.
Полный текст
Введение
Коронавирусы летучих мышей, принадлежащие родам Alphacoronavirus и Betacoronavirus (Coronaviridae: Orthocoronavirinae), обладают значительным зоонозным потенциалом [1–4]. Результаты многочисленных исследований показывают, что большинство известных коронавирусов человека, включая сезонные альфакоронавирусы (α-CoV) HCoV-229E, HCoV-NL63 и бетакоронавирусы (β-CoV) SARS-CoV, SARS-CoV-2, MERS-CoV происходят от коронавирусов, ассоциированных с разными видами летучих мышей [5–7]. Коронавирусы, эволюционно связанные с вирусами летучих мышей, найдены в последние время у разных видов диких и домашних животных [8, 9]. Это позволяет рассматривать летучих мышей как один из основных природных резервуаров и источников зоонозных коронавирусов, который играет ключевую роль в распространении коронавирусов или их отдельных генов между разными видами млекопитающих.
Летучие мыши относятся к отряду рукокрылых (Chiroptera), который включает более 1400 видов, объединенных в 21 семейство и 234 рода. По числу видов рукокрылые занимают второе место после грызунов среди всех млекопитающих. На территории России зарегистрированы не менее 45 видов летучих мышей, включая 3 вида подковоносов (Rhinolophidae: Rhinolophus): большой (R. ferrumequinum (Schreber, 1774), малый (R. hipposideros (Bechstein, 1800) и южный (R. euryale (Blasius, 1853) [10]. Подковоносы рассматриваются как природный резервуар SARS-подобных коронавирусов, два из которых (SARS-CoV и SARS-CoV-2) вызвали эпидемическую вспышку тяжелого острого респираторного синдрома (ТОРС) в 2002–2004 гг. и пандемию COVID-19 в 2019 г. соответственно. В Китае и странах Юго-Восточной Азии у различных видов подковоносов были обнаружены несколько групп дивергентных альфакоронавирусов, такие как Rhinolophus bat coronavirus HKU32 или группа HKU2 (подрод Rhinacovirus), к которой принадлежит возбудитель синдрома острой диареи свиней (SADS-CoV), вызвавший крупную эпизоотию в Китае в 2016–2019 гг. [9, 11–13]. Особый интерес представляет R. ferrumequinum, чей ареал практически непрерывно тянется от Северной Африки, Южной и Западной Европы через Среднюю Азию до Гималаев, Кореи и Японии, обеспечивая возможность распространения ассоциированных с ним вирусов в отдаленные регионы. В России ареал подковоносов ограничен территориями, лежащими южнее 44° северной широты, включая Северный Кавказ и северное побережье Черного моря. Исследования коронавирусов в российских популяциях летучих мышей носят фрагментарный характер [14–17]. Ранее при обследовании подковоносов в районе Большого Сочи (Краснодарский край) мы описали два новых SARS-подобных бетакоронавируса летучих мышей (вирусы Хоста-1 и Хоста-2), эволюционно связанных с вирусами SARS-CoV и SARS-CoV-2, а также с сарбековирусами летучих мышей из Европы, Африки и Азиатского региона [14]. Распространение близкородственных Хоста-1 вирусов отмечено и в других районах Большого Кавказа – в колониях большого подковоноса в Дагестане [17] и у южного подковоноса в Грузии [18]. В этих работах также был обнаружен альфакоронавирус летучих мышей, предварительно отнесенный авторами к подроду Decacovirus на основе консервативного домена RdRp (397 нуклеотидных остатков (н.о.)) [17]. В настоящей работе мы идентифицировали и полногеномно описали новый альфакоронавирус летучих мышей (названный вирусом «Кудеп», от названия реки Кудепста), ассоциированный с R. ferrumequinum. Результаты ПЦР-скрининга показали, что альфа- и бетакоронавирусы совместно коциркулируют в популяции большого подковоноса в данном регионе.
Материалы и методы
Материал для исследования. Сбор материала проводили в пещерах южного макросклона Большого Кавказа на северном побережье Черного моря (район Сочи-Адлер, Краснодарский край) в 2020‒2024 гг. (таблица). Сбор осуществляли или поздней осенью, когда колония летучих мышей слетается на зимовку (2020, 2021 гг.), или ранней весной во время первых вылетов после спячки (2024 г.). Животных отлавливали руками со сводов пещер. Вид определял на основе морфологических признаков опытный зоолог. Дополнительно видовая принадлежность R. hipposideros и R. ferrumequinum была подтверждена путем секвенирования митохондриального гена цитохрома b для нескольких выбранных проб. Для сбора фекалий летучих мышей каждую отдельную особь помещали в чистый хлопковый мешочек на 10‒15 мин, после чего животное выпускали, а фекалии со стенки мешочка помещали в криопробирку. Материал доставляли в лабораторию на льду и хранили при температуре −70 °С. Ни одно животное при сборе не было забито или повреждено. Сбор материала был одобрен Научным советом и комитетом по этике Сочинского национального парка (Протокол № 5 от 27 декабря 2023 г.).
Таблица. Результаты ОТ-ПЦР-скрининга подковоносых летучих мышей (Rhinolophus spp.) на наличие коронавирусов Хоста-1, Хоста-2 (Betacoronavirus) и Кудеп (Alphacoronavirus) в 2020, 2021 (осень) и 2024 (весна) гг., северное побережье Черного моря
Table. Results of RT-PCR screening of horseshoe bats (Rhinolophus spp.) on coronaviruses Khosta-1, Khosta-2 (Betacoronavirus), and Kudep (Alphacoronavirus), collected in 2020, 2021 (autumn), and 2024 (spring), Northern coast of the Black Sea
Локация Location | Вид Bat species | 2020 (осень) 2020 (autumn) | 2021 (осень) 2021 (autumn) | 2024 (весна) 2024 (spring) | ||||||
Хоста-1 Khosta-1 (β-CoV) | Хоста-2 Khosta-2 (β-CoV) | Кудеп Kudep (α-CoV) | Хоста-1 Khosta-1 (β-CoV) | Хоста-2 Khosta-2 (β-CoV) | Кудеп Kudep (α-CoV) | Хоста-1 Khosta-1 (β-CoV) | Хоста-2 Khosta-2 (β-CoV) | Кудеп Kudep (α-CoV) | ||
Подвал НИИ мед. приматологии Institute of Medical Primatology (43°26’06.3” N 39°59’26.4” E) | R. hipposideros | 0/24 | 2/24 (8,3%) | 0/24 | 0/13 | 1/13 (7,7%) | 0/13 | ‒ | ‒ | ‒ |
R. euryale | 0/1 | 0/1 | 0/1 | ‒ | ‒ | ‒ | ‒ | ‒ | ‒ | |
Пещера Музейная (Хостинская 2) Museinaya cave (Khosta 2 cave) (43°33’34.3” N 39°53’46.2” E) | R. ferrumequinum | 0/2 | 0/2 | 0/2 | ‒ | ‒ | ‒ | ‒ | ‒ | ‒ |
R. hipposideros | 0/2 | 0/2 | 0/2 | ‒ | ‒ | ‒ | ‒ | ‒ | ‒ | |
Пещера Хостинская 1 Khosta 1 cave (43°33’49.5” N 39°53’57.2” E) | R. ferrumequinum | 1/13 (7,7%) | 0/13 | 2/13 (15,3%) | 1/2 | 0/2 | 1/2 | ‒ | ‒ | ‒ |
Пещера Колокольная Kolokolnaya cave (43°33’08.3” N, 39°56’02.4” E) | R. ferrumequinum | 15/24 (62,5%) 1* | 0/24 | 2/24 (8,3%) 1* | 36/51 (70,5%) 21* | 0/51 | 27/51 (52,9%) 21* | 3/22 (13,6%) | 0/22 | 3/22 (13,6%) |
R. euryale | 0/2 | 0/2 | 0/2 | ‒ | ‒ | ‒ | ‒ | ‒ | ‒ | |
R. hipposideros | ‒ | ‒ | ‒ | ‒ | ‒ | ‒ | 0/2 | 0/2 | 0/2 | |
Пещера Партизанская Partizanskaya cave (43°37’38.86” N, 39°54’46.06” E) | R. ferrumequinum | 0/1 | 0/1 | 0/1 | ‒ | ‒ | ‒ | ‒ | ‒ | ‒ |
R. hipposideros | 1/3 (33%) | 0/3 | 0/3 | ‒ | ‒ | ‒ | ‒ | ‒ | ‒ | |
Всего Total | 17/70 (14,9%) | 2/70 (1,75%) | 4/70 (3,5%) | 37/66 (56,0%) | 1/66 (1,5%) | 28/66 (42,4%) | 3/24 (12,5%) | 0/24 | 3/24 (12,5%) | |
Примечание. Указано число положительных проб/общее число обследованных проб для каждого вида и локации. Указанное значение долей (%) является индикативным и не подтверждено статистическими методами. * – число проб, одновременно положительных по вирусу Хоста-1 и вирусу Кудеп.
Note. The number of positive samples/number of samples is shown for each species and location. The percentage value (%) is indicative and is not confirmed by statistical methods. * – number of samples simultaneously positive for Khosta-1 virus and Kudep virus.
Выделение РНК и полимеразная цепная реакция с обратной транскрипцией (ОТ-ПЦР). Образцы суспендировали в 0,2 мл фосфатно-солевого буфера (PBS). Суммарная РНК была выделена из 0,1 мл полученной суспензии с использованием набора РНК-Экстран (НПК «Синтол», Россия) в соответствии с инструкцией производителя. Тестирование проб на наличие РНК вирусов Хоста-1 и Хоста-2 проводили, как было указано ранее [14], с использованием специфических праймеров и зондов (Kh1_pr FAM-ACCTGTGCCTGTGAGTCCATT-BQ1, Kh1_F CACTGTTGGTGTAGGTTAC, и Kh1_R CTGGAATGACTGAAATCTCTTA для Хоста-1; Kh2_pr HEX-AAGCACACCAACGACACCAGCATCTC-BQ2, Kh2_F CGCCAAGCACTATTAAAGACAG, и Kh2_R CGAAGTCGTACCAGTTTCCA для вируса Хоста-2) и реагента TaqPath 1-Step Multiplex Master Mix (ThermoFisher Scientific, США). Кратко: 5 мкл РНК добавляли к 15 мкл реакционной смеси, содержащей 1× TaqPath 1-Step Multiplex Master Mix, по 400 нМ прямого и обратного праймера и 200нМ соответствующего зонда. Температурный режим реакции: инкубация 30 мин при 50 °C для реакции обратной транскрипции, инкубация 30 с при температуре 95 °C для активации Taq-полимеразы. Далее 45 циклов ПЦР: 10 с при 95 °C для денатурации и 30 с при 55 °C для элонгации и регистрации сигнала. Выявление РНК нового альфакоронавируса Кудеп проводили с использованием подобранных праймеров и зондов (Alph_pr ROX-TGCCACAAGTTGCCACCGTCA-BQ2, Alph_F GCTTGCTGCTGAAGATCC, Alph_R CCACATCATTAACAGTGCGAATA) по такой же методике.
Высокопроизводительное секвенирование (NGS) и анализ данных. Секвенирование проб проводили, как описано ранее [14]. Часть проб были пулированы по видам и локациям (10‒12 проб в пуле). Положительные в ПЦР пробы секвенировали индивидуально. Для удаления рибосомальной РНК использовали набор NEBNext rRNA Depletion kit (NEB, США). Для получения кДНК библиотек использовали набор NEBNext Ultra II RNA library kit for Illumina (NEB, США) в соответствии с инструкцией производителя. Измерение молярности полученных библиотек проводили методом ПЦР в реальном времени согласно рекомендациям, изложенным в руководстве Sequencing Library qPCR Quantification Guide (Illumina, США). Секвенирование кДНК-библиотек осуществляли в формате 2 × 100 на приборе NovaSeq 6000 (Illumina, США) на базе центра Genetico (Москва). В среднем получали 80‒110 млн парных ридов для пулированных проб и 25‒40 млн парных ридов для индивидуальных проб. Полученные риды были отфильтрованы по качеству и после удаления адаптеров собраны de novo с использованием программного обеспечения CLC Genomics Workbench 7.0 (Qiagen, Германия). Полученные контиги анализировали по алгоритму blastx с использованием программы DIAMOND против базы данных протеинов вирусов семейства Coronaviridae, загруженных из базы GenBank с использованием собственного Python скрипта (модуль BioPython).
Генетический и филогенетический анализ. Нуклеотидные и аминокислотные последовательности альфакоронавирусов были выровнены по алгоритму ClustalW, имплементированного в программное обеспечение MEGAX (https://www.megasoftware.net/). Всего 50 доступных в GenBank геномов альфакоронавирусов, принадлежащих 9 (из 16) подродам были использованы для анализа. Аминокислотные выравнивания для отдельных белков (ORF1a, ORF1b, S, ORF3, E, M, и N) были использованы для расчета значения идентичности (identity, %) между разными вирусами. Для филогенетического анализа подбирали лучшую модель замен с помощью «model selection module» в программе MEGAX. Филогенетические деревья были построены на основе аминокислотных последовательностей RdRp, поверхностного спайкового белка S и белка нуклеокапсида N с использованием выбранной модели (LG + G + I + F для RdRp, LG + G + F для белков S и N соответственно) и бутстреп тестированием (1000×) в программе MEGAX. Для таксономического анализа использовали критерии, принятые Международным комитетом по таксономии вирусов (ICTV, https://ictv.global/report/chapter/coronaviridae/coronaviridae) [19]. Для этого аминокислотные последовательности консервативных доменов гена ORF1ab (nsp5(3CLpro), NiRAN, nsp12(RdRp), ZBD, nsp13(Hel1)) для каждого из анализируемых вирусов были объединены в одну последовательность (конкатенация), выровнены по алгоритму ClustalW и использованы для расчета процентов различающихся аминокислот. Вирусы, принадлежащие разным видам, должны иметь не более 92% идентичных а.о. в данных последовательностях консервативных доменов.
Результаты
Идентификация и генетическая характеристика нового альфакоронавируса летучих мышей. После сборки de novo геном нового альфакоронавируса Кудеп был идентифицирован в виде протяженного контига длиной 28 142 н.о. (включая поли(А)-хвост) в пробе F2, содержащей материал от R. ferrumequinum, собранный в п. Колокольная в 2020 г. После подбора специфических праймеров и зонда и ПЦР-скрининга (см. ниже) было проведено секвенирование индивидуальных положительных проб, содержащих РНК вируса Кудеп в высоком титре (значение Ct < 25 в ОТ-ПЦР в реальном времени). В результате получены практически полные геномные последовательности для 7 штаммов вируса Кудеп, включая один штамм 2020 г. (Kudep/F2/2020) и 6 штаммов 2021 г. (штаммы Kudep/2021/59, Kudep/2021/71, Kudep/2021/80, Kudep/2021/86, Kudep/2021/100 и Kudep/2021/107). Все секвенированные штаммы были получены от R. ferrumequinum из п. Колокольная в 2020 и 2021 гг. соответственно.
Анализ полученных данных показал, что геномы штаммов 59, 71 и 86 (все 2021 г.) на 100% идентичны и не содержат нуклеотидных замен при попарном сравнении геномов. Также 100% идентичность между собой имеют штаммы 80 и 107 (оба 2021 г.). Между этими двумя группами идентичных штаммов наблюдается 27 н.о. замен при полногеномном сравнении. Шестой штамм 2021 г. (под номером 100) отличается от группы 59/71/86 на 7 нуклеотидных замен и на 34 н.о. от группы 80/107. Прототипный штамм вируса Кудеп F3/2020 отличается от штаммов 2021 г. на 8 н.о. (штаммы 80, 107), 19 н.о. (штаммы 59, 71, 86) и 32 н.о. (штамм 100). Отличительной особенностью штаммов F3, 80, и107 является инсерция 9 н.о. в гене нуклеопротеина, приводящая к вставке 164QNN166 белке N, отсутствующая у штаммов 59, 71 и 86.
Геном нового альфакоронавируса Кудеп имеет характерную для альфакоронавирусов структуру. Две трети генома с 5’-конца занимает ген ORF1ab, который транслируется в виде протяженного полипротеина и нарезается на два основных белка – ORF1a и ORF1b. Дальнейший процессинг приводит к образованию 16 неструктурных белков репликативного комплекcа (nsp1‒nsp11 из ORF1a, nsp12‒nsp16 из ORF1b). РНК-зависимая РНК-полимераза RdRp (nsp12), которая является наиболее консервативным белком, кодируется регионом 1‒927 аминокислотных остатков (а.о.) полипротеина ORF1b. Остальная часть генома занята генами четырех структурных белков (S, E, M, и N) и генами нескольких неструктурных белков, число и структура которых может варьировать у разных коронавирусов. Всего в геноме вируса Кудеп обнаруживается 8 открытых рамок считывания (ORF), кодирующих характерные для коронавирусов белки: ORF1ab-S-ORF3a-ORF3b-E-M-N-ORFx. Отличительной особенностью генома вируса Кудеп является наличие дополнительной рамки считывания между генами S и E, в результате чего ORF3 разделена на два гена. Первый из них, ORF3a, кодирует белок длиной 120 а.о., который не обнаруживает гомологов среди известных вирусных белков при анализе в сервисе BLASTp (https://blast.ncbi.nlm.nih.gov/Blast.cgi). Следующая рамка считывания, ORF3b, кодирует неструктурный белок, имеющий сходство 55‒78% в а.о. с соответствующими белками других альфакоронавирусов летучих мышей. В геноме вируса Кудеп отсутствуют гены вспомогательных белков ORF4, ORF7, ORF8 и ORF9, часто встречающиеся у вирусов летучих мышей, но обнаруживается ген предполагаемого белка ORFX (107 а.о.) с неизвестной функцией.
Структура генома вируса Кудеп наиболее схожа с вирусом Cardioderma bat coronavirus/Kenya/KY43/2006 (штамм BtKY43), изолированным от Африканского ложного вампира (Cardioderma cor) в Кении в 2006 г. [20]. При полногеномном сравнении вирус Кудеп имеет максимальное значение идентичности (72% н.о.) с BtKY43 и его штаммами, и 67% н.о. идентичности с группой вирусов подковоносов из Китая (YN2012). Уровень идентичности RdRp (ORF1b) вируса Кудеп составляет 92% а.о. с вирусом BtKY43. С вирусами из Китая и Юго-Восточной Азии это значение составляет 87‒90% а.о. Большинство структурных белков вируса Кудеп также имеют максимальную схожесть с BtKY43, включая поверхностный белок Е (85% а.о. идентичности), мембранный протеин М (90‒92% а.о.), и нуклеокапсидный белок N (81% а.о.). При сравнении с вирусами из Азиатского региона наибольшие значения идентичности составляют 83% а.о. для протеина Е (штамм HuB2013, R. ferrumequinum, 2013 г., Китай), 90% для протеина М (штамм PH20, R. shameli, 2010 г., Камбоджа) и 69,7% для протеина N (штамм RfLN20, R. ferrumequinum, 2020 г., Китай) соответственно.
Последовательность поверхностного белка S вируса Кудеп имеет 58‒73% а.о. идентичности с известными альфакоронавирусами, причем наиболее близкими из них (73% а.о. идентичности) являются вирусы BtCoV/Rh/YN2012_Rs4125 и BtCoV/Rh/YN2012_Rs4259, изолированные от подковоносов в Китае в 2012‒2013 гг. Тогда как схожесть S-белка вируса Кудеп с вирусом BtKY43, который наиболее близок ему по RdRp, составляет только 62% а.о. Два неструктурных вспомогательных белка, ORF3b и предполагаемый ORFХ, имеют 77 и 55% а.о. идентичности с BtKY43 соответственно.
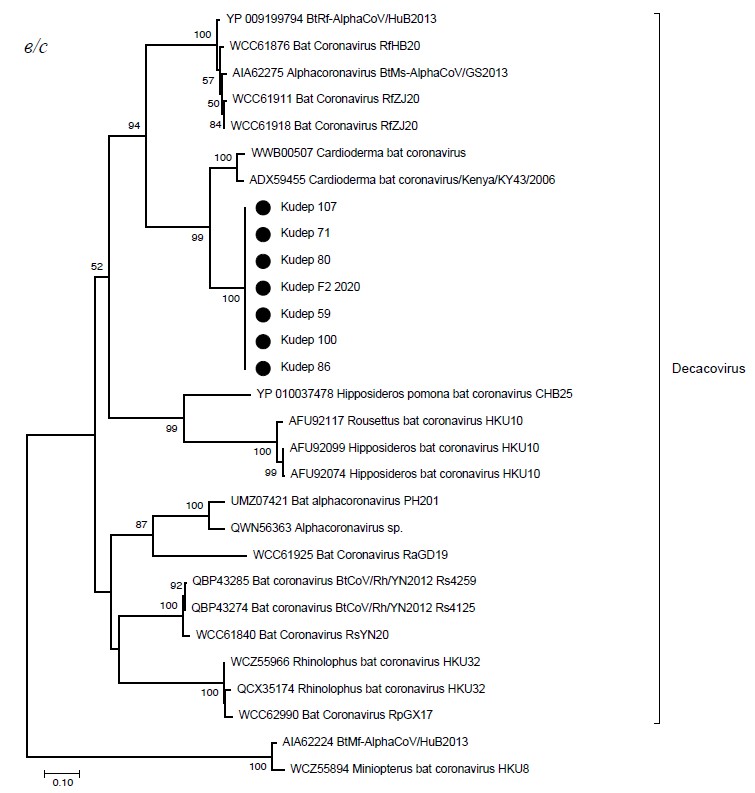
Филогенетический анализ. На рисунке представлена филогения отельных представителей рода Alphacoronavirus на основе консервативного протеина RdRp (рис. а), спайкового протеина S (рис. б) и нуклеопротеина N (рис. в). На всех филогенетических деревьях новый альфакоронавирус Кудеп формирует отдельную ветвь внутри подрода Decacovirus. На дендрограммах, построенных на основе RdRp и белка нуклеокапсида N, вирус Кудеп является сестринской кладой для африканского BtKY43. На дендрограмме для белка S ближайшая к вирусу Кудеп генетическая линия представлена штаммами от подковоносов из Китая.
Рисунок. Филогения вируса Кудеп, построенная на основе анализа аминокислотной последовательности RdRp (a), спайкового белка S (б), белка нуклеокапсида N (в) отдельных представителей рода Alphacoronavirus.
Вирус Кудеп отмечен черным кружком; вирусы человека ‒ прозрачным треугольником.
Figure. Phylogeny of Kudep virus based on analysis of RdRp (a), spike protein S (b), and nucleocapsid protein N (c) of certan representatives of the genus Alphacoronavirus.
The Kudep virus is marked with a black circle. Human coronaviruses are marked with a transparent triangle.
Таксономический анализ. При попарном сравнении консервативных доменов гена ORF1ab (nsp5(3CLpro), NiRAN, nsp12(RdRp), ZBD, nsp13(Hel1)) вируса Кудеп с другими альфакоронавирусами, максимальное сходство (91% идентичных а.о.) получено для BtKY43. С ближайшими штаммами от подковоносов из Китая это значение составило 88%.
Результаты ОТ-ПЦР-обследования. В таблице представлены результаты ОТ-ПЦР-обследования подковоносов на наличие сарбековирусов Хоста-1, Хоста-2 (Betacoronavirus) и нового альфакоронавируса Кудеп. Вирус Хоста-1 преимущественно ассоциирован с R. ferrumequinum, только одна положительная проба (из 3 протестированных из п. Партизанская) принадлежала R. hipposideros. Наибольшее число положительных проб было получено из колонии большого подковоноса в п. Колокольная. Общая зараженность вирусом Хоста-1 R. ferrumequinum в колонии в п. Колокольная в осенний период составила 62,5–70% (2020‒2021 гг.). В осенний период 2024 г. зараженность колонии определена в 13%. Общая зараженность подковоносов в регионе вирусом Хоста-1 составила от 12,5% (2024 г.) до 56% (2021 г.). Вирус Хоста-2 был выявлен только у R. hipposideros и только в одной локации за все время наблюдения
Новый альфакоронавирус Кудеп был обнаружен только у R. ferrumequinum в двух достаточно близко расположенных локациях – в п. Хостинская-1 и в п. Колокольная. Так же, как и в случае с вирусом Хоста-1, зараженность R. ferrumequinum новым альфакоронавирусом Кудеп достигала высоких значений в осенний период (до 52,9% в 2021 г.). Весной 2024 г. этот показатель составил 13,6%. Общая зараженность подковоносов новым альфакоронавирусом Кудеп составила от 3,5% (2020 г.) до 42,4% (2021 г.). При этом 21 проба из п. Колокольная в 2021 г. была одновременно положительной на вирус Хоста-1 и новый альфакоронавирус Кудеп. В материале из этой пещеры в 2020 г. обнаружена только одна такая проба (из 2 положительных по вирусу Кудеп). В п. Хостинская-1, второй локации, где обнаружен новый альфакоронавирус Кудеп, коинфекция с вирусом Хоста-1 выявлена в одной пробе в 2020 г. (из 2 положительных) и в одной пробе в 2021 г.
Обсуждение
Летучие мыши рассматриваются как основной природный резервуар для альфа- и бетакоронавирусов. В результате преодоления межвидового барьера коронавирусы летучих мышей передаются другим видам млекопитающих, включая сельскохозяйственных животных и человека, что часто приводит к возникновению эпидемических или эпизоотических вспышек и пандемий. Результаты многочисленных исследований показывают, что коронавирусы летучих мышей распространены повсеместно внутри их ареала, включая Европу, Африку, Китай и Юго-Восточную Азию и регионы Нового Света [21–24]. В настоящей работе мы идентифицировали новый альфакоронавирус Кудеп, циркулирующий в популяции R. ferrumequinum на юге России (субтропический регион Краснодарского края, Сочи-Адлер). Всего нами полногеномно охарактеризованы 7 штаммов вируса Кудеп, обнаруженных в 2020 г. (1 штамм) и 2021 г. (6 штаммов). Хотя все секвенированные штаммы вируса Кудеп получены из одной локации с интервалом в 1 год, между ними наблюдаются определенные различия как в виде отдельных нуклеотидных замен, так и в виде делеций/инсерций в структурном белке нуклеокапсида, что является отражением идущего эволюционного процесса. Дальнейшие наблюдения позволят нам точно определить скорость накопления мутаций для данной вирусной популяции.
Геном вируса Кудеп имеет характерные для альфакоронавирусов размер и структуру. Наибольшее сходство геном вируса Кудеп имеет с альфакоронавирусом Cardioderma bat coronavirus (штаммы BtKY43, 2A/Kenya/BAT2621/2015 и 2B/Kenya/BAT2618/2015), который был обнаружен у Африканского ложного вампира (Cardioderma cor) в Кении [20]. Оба вируса характеризуются отсутствием генов вспомогательных неструктурных белков ORF4, ORF7, ORF8 и ORF9, которые в том или ином виде, как правило, присутствуют у альфакоронавирусов летучих мышей из Азиатского региона [25]. При сравнении полных геномов вирус Кудеп имеет приблизительно одинаковый уровень идентичности (67‒72% н.о.) с BtKY43 и с рядом вирусов, найденных у Rhinolophus spp. в Китае. При анализе аминокислотных последовательностей структурных и неструктурных белков вирус Кудеп также оказывается равноудален и от африканских, и от азиатских вирусов (87‒92% идентичности по RdRp).
Филогенетический анализ относит вирус Кудеп в подрод Decacovirus. Подрод включает в себя несколько линий вирусов, связанных с разными видами подковоносов (Rhinolophus bat coronavirus HKU32, YN2012, и др.), а также вирусы HKU10, найденные у подковогубых летучих мышей (Hipposideros bat coronavirus HKU10) и крыланов (Rousettus bat coronavirus HKU10) в Китае. К этому же подроду относятся два вируса от ложных вампиров (Megadermatidae) – ранее упомянутый BtKY43 из Кении и Megaderma bat coronavirus из Бангладеш. Таким образом, альфакоронавирусы данного подрода обладают значительной экологической пластичностью, что позволяет им совершать межвидовую трансмиссию между различными семействами летучих мышей и даже между представителями разных подотрядов отряда рукокрылых [26]. Положение вируса Кудеп внутри подрода Decacovirus практически совпадает на деревьях, построенных на основе RdRp и на основе структурного белка нуклеокапсида N, где вирус Кудеп формирует отдельную генетическую линию и кластеризуется рядом с линией BtKY43. Филогения на основе S-белка помещает линию вируса Кудеп рядом с одной из линий вирусов подковоносов (YN2012) из Китая [11].
Для определения таксономического положения вируса Кудеп мы проанализировали схожесть его консервативных доменов гена ORF1ab (nsp5(3CLpro), NiRAN, nsp12(RdRp), ZBD, nsp13(Hel1)) c другими известными альфакоронавирусами. Полученные максимальные значения схожести составили 91% идентичных а.о. (с BtKY43), что соответствует демаркационным критериям ICTV (92% идентичных а.о.) при определении отдельного вида коронавирусов (https://ictv.global/report/chapter/coronaviridae/coronaviridae) [19]. Таким образом, мы предполагаем, что найденный нами вирус Кудеп представляет новый вид в составе подрода Decacovirus рода Alphacoronavirus.
Результаты ОТ-ПЦР-скрининга показывают, что ранее описанный нами SARS-подобный бетакоронавирус Хоста-1 и новый альфакоронавирус Кудеп активно циркулируют на обследованной территории. Зараженность этими вирусами в колонии R. ferrumequinum в п. Колокольная осенью 2021 г. достигала 70,5 и 59,2% соответственно. Многочисленные исследования демонстрируют, что зараженность летучих мышей коронавирусами может варьировать в широких пределах – от 0 до 60–70%, в зависимости от локации и времени года [27–30]. В весеннем сборе 2024 г. зараженность большого подковоноса оказалась значительно ниже (13%), но выявление вирусов Хоста-1 и Кудеп ранней весной может свидетельствовать об активной персистирующей инфекции.
В обоих колониях большого подковоноса (п. Хостинская-1 и п. Колокольная), где обнаружены вирусы Хоста-1 и новый альфакоронавирус Кудеп, выявлены случаи коинфекции отдельных особей двумя вирусами. В 2021 г. такие пробы составили более ½ от всех положительных проб. Обнаружение коронавирусов и в осенний, и в весенний периоды на протяжении 4 лет наблюдения (2020–2024 гг.) позволяет сделать вывод о наличии здесь стойкого природного очага двух коронавирусов ‒ бетакоронавируса Хоста-1 и нового альфакоронавируса Кудеп, экологически связанных с R. ferrumequinum.
Необходимо отметить, что в обследованных пещерах, населенных подковоносами, отмечены следы посещения пещер спелеологами, стадом домашних коз (летом в поиске прохлады), шакалами и дикими лесными котами. Это создает возможность экспозиции и передачи выявленных коронавирусов летучих мышей человеку или животным. Выявление вирусного разнообразия в природных биомах и изучение эволюционных процессов, приводящих к появлению новых вирусных инфекций, является актуальной задачей современной вирусологии. Эти исследования имеют серьезное прикладное значений в контроле появления новых и возвращающихся инфекций. Эпидемические ситуации будут возникать и в будущем, что требует объединения усилий на международном уровне, направленных на проведение постоянного мониторинга популяционного генофонда потенциально зоонозных вирусов [31, 32].
Об авторах
Сергей Викторович Леншин
ФКУЗ «Ставропольский противочумный институт» Роспотребнадзора
Email: lenshin-s@mail.ru
ORCID iD: 0000-0001-6815-2869
научный сотрудник бактериологической лаборатории Сочинского филиала
Россия, 354000, г. СочиТатьяна Викторовна Вишневская
Институт вирусологии имени Д.И. Ивановского ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
Email: t_vish77@mail.ru
ORCID iD: 0000-0002-6963-8681
научный сотрудник лаборатории биотехнологии
Россия, 123098, г. МоскваАлексей Владимирович Ромашин
ФГБУ «Сочинский национальный парк» Минприроды России
Email: romashin@sochi.com
ORCID iD: 0000-0003-4751-1484
канд. биол. наук, ведущий научный сотрудник
Россия, 354002, г. СочиЮлия Игоревна Булычева
Институт вирусологии имени Д.И. Ивановского ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
Email: boulychevayuli@yandex.ru
ORCID iD: 0000-0002-6798-7925
научный сотрудник лаборатории биологии и индикации арбовирусов
Россия, 123098, г. МоскваОлег Иванович Вышемирский
НИЦ «Курчатовский институт»
Email: olegvyshem@mail.ru
ORCID iD: 0000-0002-5345-8926
канд. мед. наук, ведущий научный сотрудник лаборатории инфекционной вирусологии Курчатовского комплекса медицинской приматологии
Россия, 354376, г. Сочи, с. ВеселоеСофья Алексеевна Соловьева
Институт вирусологии имени Д.И. Ивановского ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
Email: sassony@ya.ru
ORCID iD: 0009-0006-7021-7143
младший научный сотрудник лаборатории биологии и индикации арбовирусов
Россия, 123098, г. МоскваАся Калмановна Гительман
Институт вирусологии имени Д.И. Ивановского ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
Email: gitelman_ak@mail.ru
ORCID iD: 0000-0002-8410-2332
ведущий научный сотрудник лаборатории биотехнологии
Россия, 123098, г. МоскваАлексей Сергеевич Пазилин
Институт вирусологии имени Д.И. Ивановского ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
Email: pazilin@mail.ru
ORCID iD: 0009-0002-5665-409X
старший научный сотрудник лаборатории биотехнологии
Россия, 123098, г. МоскваДмитрий Константинович Львов
Институт вирусологии имени Д.И. Ивановского ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
Email: dk_lvov@mail.ru
ORCID iD: 0000-0001-8176-6582
академик РАН, профессор, д-р мед. наук, главный научный сотрудник
Россия, 123098, г. МоскваБен Ху
Уханьский институт вирусологии Китайской академии наук
Email: huben@wh.iov.cn
ORCID iD: 0000-0001-9194-3474
PhD, научный сотрудник Ключевой лаборатории вирусологии и биобезопасности
Китай, 430071, г. УханьЧженгли Ши
Уханьский институт вирусологии Китайской академии наук; Национальная лаборатория Гуанчжоу
Email: zlshi@wh.iov.cn
ORCID iD: 0000-0001-8089-163X
PhD, руководитель Ключевой лаборатории вирусологии и биобезопасности Уханьского института вирусологии Китайской академии наук; старший научный сотрудник, Национальная лаборатория Гуанчжоу
Китай, 430071, г. Ухань; Международный Био-остров Гуанчжоу, Гуанчжоу 51005Сергей Владимирович Альховский
Институт вирусологии имени Д.И. Ивановского ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
Автор, ответственный за переписку.
Email: s_alkhovsky@gamaleya.org
ORCID iD: 0000-0001-6913-5841
член-корр. РАН, д-р биол. наук, руководитель лаборатории биотехнологии
Россия, 123098, г. МоскваСписок литературы
- Львов Д.К. ред. Экология вирусов. В кн.: Руководство по вирусологии. Вирусы и вирусные инфекции человека и животных. М.: МИА; 2013.
- Letko M., Seifert S.N., Olival K.J., Plowright R.K., Munster V.J. Bat-borne virus diversity, spillover and emergence. Nat. Rev. Microbiol. 2020; 18(8): 461–71. https://doi.org/10.1038/s41579-020-0394-z
- Grange Z.L., Goldstein T., Johnson C.K., Anthony S., Gilardi K., Daszak P., et al. Ranking the risk of animal-to-human spillover for newly discovered viruses. Proc. Natl. Acad. Sci. USA. 2021; 118(15): e2002324118. https://doi.org/10.1073/pnas.2002324118
- Forni D., Cagliani R., Clerici M., Sironi M. Molecular Evolution of Human Coronavirus Genomes. Trends Microbiol. 2016; 25(1): 35–48. https://doi.org/10.1016/j.tim.2016.09.001
- Li W., Shi Z., Yu M., Ren W., Smith C., Epstein J.H., et al. Bats are natural reservoirs of SARS-like coronaviruses. Science. 2005; 310(5748): 676–9. https://doi.org/10.1126/science.1118391
- Львов Д.К., Альховский С.В. Истоки пандемии COVID-19: экология и генетика коронавирусов (Betacoronavirus: Coronaviridae) SARS-CoV, SARS-CoV-2 (подрод Sarbecovirus), MERS-CoV (подрод Merbecovirus). Вопросы Вирусологии. 2020; 65(2): 62–70. https://doi.org/10.36233/0507-4088-2020-65-2-62-70 https://elibrary.ru/hnouwn
- Woo P.C., Lau S.K., Lam C.S., Lau C.C., Tsang A.K., Lau J.H., et al. Discovery of seven novel Mammalian and avian coronaviruses in the genus deltacoronavirus supports bat coronaviruses as the gene source of alphacoronavirus and betacoronavirus and avian coronaviruses as the gene source of gammacoronavirus and deltacoronavi. J. Virol. 2012; 86(7): 3995–4008. https://doi.org/10.1128/jvi.06540-11
- Zhou P., Fan H., Lan T., Yang X.L., Shi W.F., Zhang W., et al. Fatal swine acute diarrhoea syndrome caused by an HKU2-related coronavirus of bat origin. Nature. 2018; 556(7700): 255–8. https://doi.org/10.1038/S41586-018-0010-9
- Леншин С.В., Ромашин А.В., Вышемирский О.И., Львов Д.К., Альховский С.В. Летучие мыши субтропической зоны Краснодарского края России как возможный резервуар зоонозных вирусных инфекций. Вопросы вирусологии. 2021; 66(2): 112–22. https://doi.org/10.36233/0507-4088-41 https://elibrary.ru/bimauw
- Wang N., Luo C., Liu H., Yang X., Hu B., Zhang W., et al. Characterization of a new member of alphacoronavirus with unique genomic features in Rhinolophus bats. Viruses. 2019; 11(4): 379. https://doi.org/10.3390/v11040379
- Wu Z., Yang L., Ren X., He G., Zhang J., Yang J., et al. Deciphering the bat virome catalog to better understand the ecological diversity of bat viruses and the bat origin of emerging infectious diseases. ISME J. 2016; 10(3): 609–20. https://doi.org/10.1038/ismej.2015.138
- Lau S.K.P., Wong A.C.P., Zhang L., Luk H.K.H., Kwok J.S.L., Ahmed S.S., et al. Novel bat alphacoronaviruses in southern China support Chinese horseshoe bats as an important reservoir for potential novel coronaviruses. Viruses. 2019; 11(5): 423. https://doi.org/10.3390/v11050423
- Alkhovsky S., Lenshin S., Romashin A., Vishnevskaya T., Vyshemirsky O., Bulycheva Y., et al. SARS-like Coronaviruses in Horseshoe Bats (Rhinolophus spp.) in Russia, 2020. Viruses. 2022; 14(1): 113. https://doi.org/10.3390/v14010113
- Yashina L.N., Zhigalin A.V., Abramov S.A., Luchnikova E.M., Smetannikova N.A., Dupal T.A., et al. Coronaviruses (Coronaviridae) of bats in the northern Caucasus and south of western Siberia. Vopr. Virusol. 2024; 69(3): 255–65. https://doi.org/10.36233//0507-4088-233
- Urushadze L., Babuadze G., Shi M., Escobar L.E., Mauldin M.R., Natradeze I., et al. A cross sectional sampling reveals novel coronaviruses in bat populations of Georgia. Viruses. 2021; 14(1): 72. https://doi.org/10.3390/v14010072
- Tao Y., Tang K., Shi M., Conrardy C., Li K.S., Lau S.K., et al. Genomic characterization of seven distinct bat coronaviruses in Kenya. Virus Res. 2012; 167(1): 67–73. https://doi.org/10.1016/j.virusres.2012.04.007
- Liu D.X., Fung T.S., Chong K.K., Shukla A., Hilgenfeld R. Accessory proteins of SARS-CoV and other coronaviruses. Antiviral. Res. 2014; 109: 97–109. https://doi.org/10.1016/j.antiviral.2014.06.013
- Lau S.K., Li K.S., Tsang A.K., Shek C.T., Wang M., Choi G.K., et al. Recent transmission of a novel alphacoronavirus, bat coronavirus HKU10, from Leschenault’s rousettes to pomona leaf-nosed bats: first evidence of interspecies transmission of coronavirus between bats of different suborders. J. Virol. 2012; 86(21): 11906–18. https://doi.org/10.1128/jvi.01305-12
- Drexler J.F., Corman V.M., Drosten C. Ecology, evolution and classification of bat coronaviruses in the aftermath of SARS. Antiviral. Res. 2014; 101: 45–56. https://doi.org/10.1016/j.antiviral.2013.10.013
- Tao Y., Tang K., Shi M., Conrardy C., Li K.S., Lau S.K., et al. Genomic characterization of seven distinct bat coronaviruses in Kenya. Virus Res. 2012; 167(1): 67–73. https://doi.org/10.1016/j.virusres.2012.04.007
- Anthony S.J., Johnson C.K., Greig D.J., Kramer S., Che X., Wells H., et al. Global patterns in coronavirus diversity. Virus Evol. 2017; 3(1): vex012. https://doi.org/10.1093/ve/vex012
- Cohen L.E., Fagre A.C., Chen B., Carlson C.J., Becker D.J. Coronavirus sampling and surveillance in bats from 1996–2019: a systematic review and meta-analysis. Nat. Microbiol. 2023; 8(6): 1176–86. https://doi.org/10.1038/s41564-023-01375-1
- Bueno L.M., Rizotto L.S., Viana A.O., Silva L.M.N., de Moraes M.V.D.S., Benassi J.C., et al. High genetic diversity of alphacoronaviruses in bat species (Mammalia: Chiroptera) from the Atlantic Forest in Brazil. Transbound. Emerg. Dis. 2022; 69(5): e2863–75. https://doi.org/10.1111/tbed.14636
- Caraballo D.A., Sabio M.S., Colombo V.C., Piccirilli M.G., Vico L., Hirmas Riade S.M., et al. The role of Molossidae and Vespertilionidae in shaping the diversity of alphacoronaviruses in the Americas. Microbiol. Spectr. 2022; 10(6): e0314322. https://doi.org/10.1128/spectrum.03143-22
- Liu D.X., Fung T.S., Chong K.K., Shukla A., Hilgenfeld R. Accessory proteins of SARS-CoV and other coronaviruses. Antiviral. Res. 2014; 109: 97–109. https://doi.org/10.1016/j.antiviral.2014.06.013
- Lau S.K., Li K.S., Tsang A.K., Shek C.T., Wang M., Choi G.K., et al. Recent transmission of a novel alphacoronavirus, bat coronavirus HKU10, from Leschenault’s rousettes to pomona leaf-nosed bats: first evidence of interspecies transmission of coronavirus between bats of different suborders. J. Virol. 2012; 86(21): 11906–18. https://doi.org/10.1128/jvi.01305-12
- Fan Y., Zhao K., Shi Z.L., Zhou P. Bat Coronaviruses in China. Viruses. 2019; 11(3): 210. https://doi.org/10.3390/v11030210
- Balboni A., Palladini A., Bogliani G., Battilani M. Detection of a virus related to betacoronaviruses in Italian greater horseshoe bats. Epidemiol. Infect. 2011; 139(2): 216–9. https://doi.org/10.1017/s0950268810001147
- Wang L.F., Shi Z., Zhang S., Field H., Daszak P., Eaton B.T. Review of bats and SARS. Emerg. Infect. Dis. 2006; 12(12): 1834–40. https://doi.org/10.3201/eid1212.060401
- Drexler J.F., Corman V.M., Drosten C. Ecology, evolution and classification of bat coronaviruses in the aftermath of SARS. Antiviral. Res. 2014; 101: 45–56. https://doi.org/10.1016/j.antiviral.2013.10.013
- Lvov D.K., Shchelkanov M.Y., Alkhovsky S.V., Deryabin P.G. Zoonotic viruses of Northern Eurasia: Taxonomy and ecology. London: Academic Press, Elsevier; 2015.
- Львов Д.К., Борисевич С.В., Альховский С.В., Бурцева Е.И. Современные подходы анализа вирусных геномов в интересах биобезопасности. Инфекционные болезни: Новости, Мнения, Обучение. 2019; 8(2): 96–101. https://doi.org/10.24411/2305-3496-2019-12012 https://elibrary.ru/xbkmpl
Дополнительные файлы